-
Články
- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Detekce proteinů, proteinových interakcí a modifikací s využitím „proximity ligation assay“ in situ
In Situ Proximity Ligation Assay for Detection of Proteins, Their Interactions and Modifications
To understand cellular processes and events responsible for their perturbations, proteomic analyses are needed in biomedical research and clinical diagnostics. Several techniques based on specifically binding reagents (antibodies) or recombinant proteins (GFP fusion protein, methods of fluorescence/ bioluminescence resonance energy transfer) are generally used to study protein location and activity resulting from secondary modifications and interactions. The in situ proximity ligation assay represents a novel technique of in situ protein imaging using DNA as a reporter molecule and DNA amplification processes. This method enables direct visualization of single molecules, their levels, modifications and pattern of interactions in individual fixed cells and tissues. Proximity probes consist of specific antibody with attached oligonucleotides that are used as reporter molecules for identification of such events. Proximity probes guide the formation of a circular DNA strand when bound in close proximity. The DNA circle after that serves as a template for rolling ‑ circle amplification allowing the interaction to be visualized. Compared to available proteomic techniques benefiting from genetic engineering, in situ PLA enables study of endogenous proteins in their natural environment and thus can be used for clinical specimens. The areas of applicability where proximity ligation procedure can be used include any research field where protein interaction measurements are important, such as signaling pathway studies, monitoring of pharmacological treatment targets and oncological diagnostics.
Key words:
in situ PLA – protein interaction – protein detection methods – proximity ligation
This work was supported by the European Regional Development Fund and the State Budget of the Czech Republic (RECAMO, CZ.1.05/2.1.00/03.0101) and by MH CZ – DRO (MMCI, 00209805) and BBMRI_CZ (LM2010004).
The authors declare they have no potential conflicts of interest concerning drugs, products, or services used in the study.
The Editorial Board declares that the manuscript met the ICMJE “uniform requirements” for biomedical papers.Submitted:
31. 1. 2014Accepted:
25. 3. 2014
Autoři: V. Brychtová; B. Vojtěšek
Působiště autorů: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno
Vyšlo v časopise: Klin Onkol 2014; 27(Supplementum): 87-91
Souhrn
Pro správné pochopení fyziologických procesů v buňce a případně jejich odchylek jsou molekulárně‑biologické analýzy nezbytným nástrojem využívaným v biomedicínském výzkumu a také v klinické diagnostice. Existuje množství technik, které umožňují určit lokalizaci studovaných proteinů a jejich interakční aktivitu. Tyto přístupy využívají především interakce specificky se vážících molekul s cílovými proteiny (protilátky) nebo synteticky připravené rekombinantní proteiny (GFP fúzní protein; metody fluorescenčního/ bioluminiscenčního rezonančního přenosu energie). „Proximity ligation assay“ (PLA) in situ představuje novou techniku zobrazující proteiny na úrovni jednotlivých buněk a tkání s využitím reportérové molekuly DNA a DNA modifikujících procesů. Tato metoda umožňuje přímou vizualizaci proteinů, jejich hladiny, modifikace a interakce v jednotlivých fixovaných buňkách a tkáních. Sondy jsou tvořeny specifickými protilátkami s navázaným oligonukleotidem, který slouží jako reportérová molekula. Pokud dojde k navázání sond v těsné blízkosti, následuje vznik kružnicové DNA, jež slouží jako templát pro amplifikaci otáčivou kružnicí. Amplifikační reakce umožňuje vizualizaci sledované interakce. Ve srovnání s dostupnými molekulárně‑biologickými metodami vycházejícími z genového inženýrství, PLA in situ umožňuje studovat endogenní proteiny v jejich přirozených podmínkách, a může být tudíž použita pro studium klinického materiálu. PLA in situ je využitelná v jakékoliv výzkumné oblasti zaměřené na studium proteinových interakcí, jako je studium buněčných signálních drah, identifikace cílů farmakologicky účinných látek či v onkologické diagnostice.
Klíčová slova:
in situ PLA – proteinové interakce – metody detekce proteinů – „proximity ligation“Úvod
Proteiny jsou biomakromolekuly, které v buňkách zprostředkovávají velké množství funkcí včetně enzymatických reakcí, transportu, buněčné signalizace, replikace a dalších. K zajištění fyziologických podmínek potřebných pro správnou funkci proteinu je mimo jiné nezbytná jeho správná subcelulární lokalizace. Ta určuje jeho aktivitu prostřednictvím vazeb s interakčními partnery a post‑translačních modifikací a umožňuje tak proteinům zapojit se do funkčních biologických drah [1]. Zjednodušeně řečeno, pokud je správné množství proteinu ve správný čas na správném místě, dochází v rámci buňky k fyziologickému transportu materiálu a informací. Nevhodná lokalizace proteinu vede k jeho inaktivaci nebo naopak ke škodlivé aktivaci na nevhodném místě, což přispívá ke vzniku mnoha onemocnění. Mezi onemocnění, jejichž etiologie zahrnuje aberantní lokalizace proteinů patří metabolické poruchy, neurodegenerativní, kardiovaskulární a nádorová onemocnění [1].
Je známo, že k tumorigenezi přispívá mimo jiné z nevhodné lokalizace plynoucí změna v proteinovém interaktomu (soubor interagujících proteinů) a změna post‑translačních modifikací proteinů. Pro správnou funkci nádorových supresorů je podstatná jejich jaderná lokalizace, a proto jejich translokace do cytoplazmy může představovat jeden z aktivačních mechanizmů, který vede k nekontrolované proliferaci buněk [2]. Například cytoplazmatická lokalizace proteinu FOXO3a představuje horší prognózu u pacientek s karcinomem mléčné žlázy a naopak jaderná lokalizace zmiňovaného proteinu koreluje se zvýšenou senzitivitou k radiační terapii [3,4]. Podobně inhibitory buněčného cyklu jako p21WAF1 a p27 se vlivem nevhodné lokalizace mimo jádro stávají tumorigenní. Cytoplazmatický p21WAF1 představuje marker negativní prognózy u HER2 pozitivního karcinomu mléčné žlázy [5]. Cytoplazmatická lokalizace p21WAF1 je markerem rezistence k léčbě cisplatinou u karcinomu vaječníku [6]. Podobným příkladem může být protein p27, jehož mimojaderná lokalizace představuje negativní prognostický faktor u mnoha nádorových onemocnění včetně karcinomu mléčné žlázy [7], hepatocelulárního karcinomu [8], karcinomu vaječníků [9] a karcinomu tlustého střeva [10]. Vedle nádorových supresorů mohou být také ve směru do jádra translokovány membránové receptory tyrozinkináz. Například jaderná lokalizace receptoru pro epidermální růstový faktor (EGFR) je asociována s rezistencí tumorů k léčbě cetuximabem a cisplatinou u řady nádorových onemocnění [11,12].
Metody používané pro detekci konkrétního proteinu, jejich post‑translačně modifikované formy nebo proteinových interakcí využívají k označení cílového proteinu převážně vazby se specifickou protilátkou nebo zavedení značky do cílového proteinu [13]. První přístup zahrnuje metody, jako je westernový přenos spojený s imunodetekcí, sandwichová ELISA (enzyme‑linked immunosorbent assay), ko ‑ imunoprecipitace s následnou identifikací pomocí hmotnostní spektrometrie a imunohistochemie. Metody přímého značení proteinu jsou založeny na principu syntézy fúzního proteinu s reportérovou molekulou, jako je např. GFP (green fluorescent protein) nebo luciferáza, které umožňují detekci cílového proteinu přímo ve vzorku. Mezi tyto metody patří metody FRET (fluorescence resonance energy transfer), BRET (bioluminiscence resonance energy transfer) [14]. Tyto metody, při nichž jsou buňky modifikovány genetickými konstrukty, jsou vynikající nástroj pro studium funkcí proteinů, avšak jsou zcela nepoužitelné v případě studií prováděných na klinickém materiálu nebo v diagnostice. „Proximity ligation assay“ (PLA) in situ, která je předmětem tohoto přehledového článku, představuje techniku kombinující vazbu specifické protilátky s reportérovým systémem a umožňující tak přímo sledovat interakce endogenních proteinů na úrovni jednotlivých buněk a tkání.
„Proximity ligation assay“
PLA představuje metodu navrženou pro detekci proteinů, proteinových komplexů a post‑translačních modifikací s využitím specifických protilátek, které na sobě nesou navázané krátké DNA vlákno s funkcí reportérové molekuly [15,16]. V případě navázání dvojice sond na cílové epitopy, které se nachází v těsné blízkosti v řádu desítek nanometrů, mohou oligonukleotidy hybridizovat s komplementárním DNA vláknem. Následnou ligací dochází k přemostění mezi oběma sondami a vzniká tak amplifikovatelná reportérová DNA. Ta je markerem vazby PLA sond v těsné blízkosti a může být kvantifikována metodou PCR. Množství signálu poskytnutého DNA reportérem odráží původní koncentraci cílových proteinů ve sledovaných vzorcích. Výhodou této metody je vznik reportérové molekuly pouze za předpokladu dvojí vazby PLA sond, čímž je snížen vznik nespecifických signálů a amplifikována tak může být pouze ligovaná DNA [13].
PLA technologie má několik modifikací vycházejících z charakteru studovaného materiálu: PLA v kapalné fázi (solution phase PLA), PLA v pevné fázi (solid phase PLA) a PLA in situ.
PLA v kapalné fázi představuje původní variantu „proximity ligation“ techniky, která pro detekci sledovaného analytu využívá identifikaci cílové molekuly pomocí rozpoznání dvou nebo více epitopů pomocí specifických PLA sond (obr. 1A) [16]. V případě navázání obou sond v těsné blízkosti vzniká reportérová DNA, jež je amplifikována a kvantifikována metodou kvantitativní PCR nebo sekvencováním nové generace [17]. Tato metoda nevyžaduje promývací kroky a je vzhledem ke své nenáročnosti na provedení vhodná pro analýzu velkého množství vzorků. Vysoká citlivost v řádu femtomolů umožňuje detekci proteinů i při objemu vzorku pouze 1 µl [16]. PLA v pevné fázi má podobný princip jako klasická sandwichová ELISA, neboť k detekci proteinů využívá protilátky navázané na pevnou fázi (mikrotitrační destička) (obr. 1B). Detekce navázaného proteinu probíhá pomocí dvou PLA sond, které vedou ke vzniku reportérového DNA vlákna. Detekce a kvantifikace reportérové DNA probíhá analogicky jako v případě PLA v kapalné fázi. Toto provedení PLA je značně citlivé, protože pro vznik signálu vyžaduje vazbu celkem tří specifických protilátek [17].
Obr. 1. Schematické znázornění principu PLA technik v kapalné (A) a pevné (B) fázi. 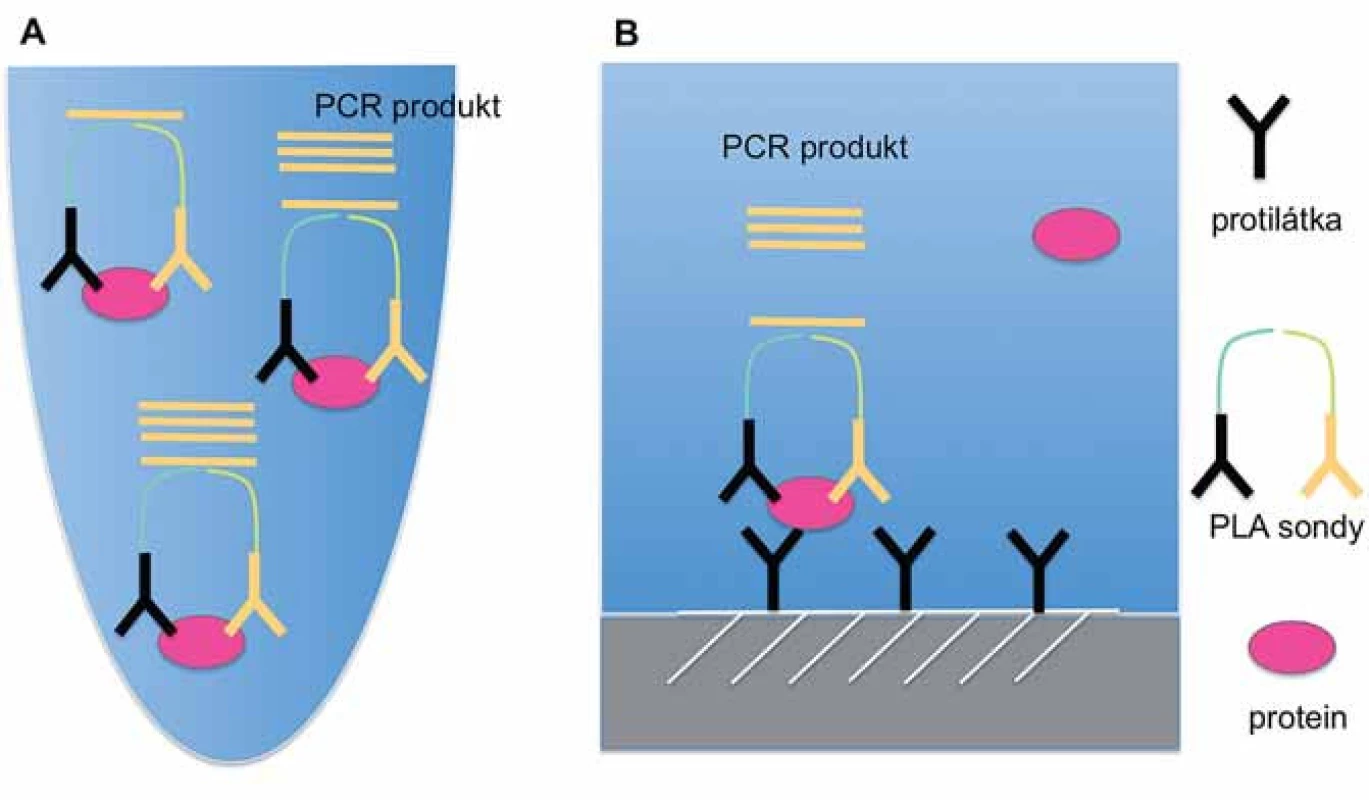
„Proximity ligation assay“ in situ
PLA in situ je varianta PLA techniky, která umožňuje studovat endogenní proteiny ve tkáních a buněčných kulturách. Tuto in situ techniku lze využít pro detekci jakýchkoliv molekul nebo komplexů, proti nimž je možné připravit specifickou protilátku. PLA sondou tak může být primární protilátka konjugovaná s oligonukleotidem, která přímo interaguje s cílovým proteinem (obr. 2A, B), nebo konjugovaná sekundární protilátka (komerčně dostupné sondy), která interaguje s primární protilátkou již navázanou k cílové molekule [18,19] (obr. 2C). Alternativně lze vedle klasických protilátek konjugovaných s oligonukleotidy použít i jiné specificky se vážící molekuly, jako jsou rekombinantní afinitní proteiny „DARPins“ (uměle připravené proteiny napodobující funkci protilátek) a DNA aptamery (uměle připravené oligonukleotidy specificky vážící cílovou molekulu) [13,15,20 – 22].
Obr. 2. Schéma detekce dimerního proteinového komplexu, post-translační modifikace proteinu a samotného proteinu metodou PLA in situ s využitím amplifikace otáčivou kružnicí (RCA) za vzniku amplifikačního produktu. 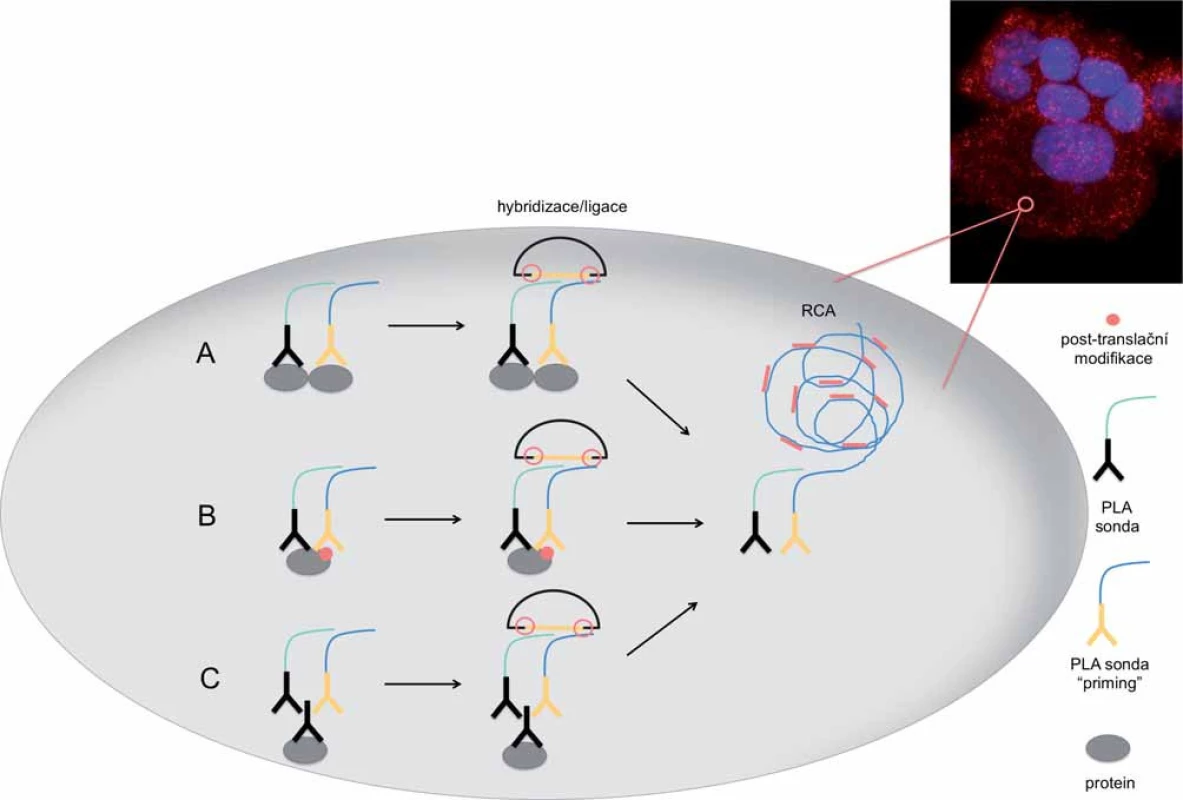
A. PLA sondy se váží k dvěma interagujícím proteinům s následnou hybridizací cirkularizačních oligonukleotidů a ligací. B. Identifi kace post-translační modifikace proteinu pomocí specifické PLA sondy. C. Detekce proteinu pomocí sekundárních PLA sond. Aby bylo dosaženo signálu v místě reálného výskytu sledované molekuly a nedocházelo k jeho difúzi, PCR je v in situ modifikaci nahrazena DNA amplifikací tak, že produkt amplifikace zůstává navázaný na cílový protein (obr. 2). PLA in situ je navržena tak, že dvojice sond slouží jako templát pro syntézu kružnicové DNA. Ta je následně amplifikována pomocí vysoce účinné Phi29 DNA polymerázy mechanizmem označovaným jako amplifikace otáčivou kružnicí (rolling circle amplification – RCA). RCA je spouštěna jednou z PLA sond označovanou jako „priming“. Produkt této reakce je prakticky prodloužením PLA sond, a tudíž zůstává fyzicky spojen s cílovými molekulami (obr. 2). RCA produkt má velikost několika set repetitivních elementů o velikosti přibližně 100 kb a je vizualizován hybridizací s fluoroforem značenými oligonukleotidy. Takto vzniklý signál o velikosti menší než 1 µm lze pozorovat fluorescenčním mikroskopem a je snadno odlišitelný od nespecifického signálu [18,23].
Kromě studia proteinových komplexů je možné PLA in situ využít také pro studium interakcí mezi proteiny a nukleovými kyselinami. V tomto případě se využívá jednovláknová DNA sonda, která zároveň slouží jako templát pro ligační reakci. Metodicky tento postup vyžaduje, aby byla genomová DNA nejprve tepelně nebo enzymaticky denaturována na jednotlivé řetězce, ke kterým by mohla specifická sonda hybridizovat [22,24].
Technika PLA in situ není omezena pouze na detekci blízkosti jednoho páru cílových molekul. Detekce většího množství molekul vyžaduje pouze použití většího množství specifických oligonukleotidů. Pro vizualizaci interakce mezi třemi molekulami je zapotřebí tří různých PLA sond, přičemž vznik kružnicové reportérové DNA je závislý na přítomnosti všech tří sond [18,23].
Mezi další aplikace PLA in situ patří identifikace proteinových post‑translačních modifikací. Pomocí specifických PLA sond lze sledovat fosforylační status proteinů [25] nebo jejich glykosylaci [26].
Kombinace PLA in situ s dalšími metodami
Velké množství onemocnění se projevuje změnami v proteinové expresi či funkci, které jsou způsobeny genetickými aberacemi nebo epigenetickými modifikacemi. Pro stanovení chromozomálních aberací na úrovni jednotlivých buněk bývá často využívána metoda fluorescenční in situ hybridizace (FISH). Kombinace PLA in situ s FISH poskytuje informace o amplifikačním statutu genů současně s hodnocením interakcí deregulovaných proteinů. Genetická analýza je takto doplněna o proteomickou analýzu ve shodném materiálu [27].
Alternativou k FISH může být využití „padlock“ sondy, což je oligonukleotid, jehož 3‘ a 5‘ konce jsou komplementární k přilehlým oblastem na cílové DNA sekvenci. Hybridizací jsou oba konce „padlock“ sondy přiblíženy k sobě a DNA je poté ligována [28]. Takto vzniklá kružnicová DNA je po navázání primeru templátem pro RCA. Ve srovnání s FISH je „padlock“ sonda mnohem specifičtější, neboť je citlivá i na jednonukleotidové záměny. Signál je podobně jako při PLA generován amplifikací otáčivou kružnicí a povahou je totožný se signálem poskytovaným PLA. Tento druh sondy je vhodný pro mRNA genotypizaci a v kombinaci s PLA in situ představuje nástroj pro simultánní detekci mutací, genové exprese a proteinových interakcí [23].
Diskuze a závěr
PLA in situ představuje relativně novou techniku rozšiřující portfolio experimentálních přístupů pro vizualizaci proteinů na úrovni buněk a tkání přibližně od poloviny minulé dekády. Od té doby prodělala vývoj, který rozšířil její možné využití. Tato metoda představuje efektivní nástroj pro analýzu funkčního stavu proteinů, jehož hlavní výhoda je schopnost amplifikace signálu, jež umožňuje detekci jednotlivých sledovaných molekul [18].
Perspektivou PLA techniky je souběžná analýza, která bude schopna paralelně sledovat více cílů v totožném materiálu. Z hlediska molekulární onkologie se nabízí využití této techniky k získání informací o povaze a chování nádorů v konkrétním mikroprostředí, čímž může přispět k lepší diagnostice a přesnější predikci léčebné odpovědi [18].
Vzhledem k ostatním metodám může PLA in situ nabídnout analýzu proteinových komplexů nebo sekundárních modifikací pro jakýkoliv protein, proti kterému je dostupná vhodná protilátka. Ve srovnání s metodami FRET/ BRET má PLA in situ dvě významné výhody, kterými je za prvé možnost studovat endogenní proteiny v přirozeném prostředí bez vedlejších vlivů způsobených nadměrnou nebo ektopickou expresí. Další výhoda je charakter signálu, při kterém PLA generuje díky RCA mnohonásobně amplifikovaný signál, jenž je jasně rozlišitelný od nespecifické fluorescence [18].
Ve farmakologii má PLA in situ využití při screeningu a validaci látek s potenciálně léčivými účinky v geneticky nemodifikovaných buněčných liniích. Multiplexní PLA in situ v tomto případě poskytuje pohled, jak látka – potenciální léčivo – ovlivňuje množství buněčných signálních drah a jejich vzájemné interakce. Ve srovnání s jinými metodami založenými na měření celkové fluorescenční intenzity je PLA in situ zvýhodněna měřením jednotlivých produktů amplifikace poskytujících jasný signál mnohem méně zatížený autofluorescencí nebo slábnutím signálu. Navíc je tato metoda citlivější při sledování méně abundantních proteinů, neboť i jednotlivý signál poskytnutý jedinou cílovou molekulou je metodou PLA in situ značně amplifikovaný, a tudíž detekovatelný [29].
Kromě studia komplexů a proteinových modifikací může být PLA teoreticky využita jako „molekulární pravítko“, které na základě variability v délce oligonukleotidu nebo charakteru protilátky umožňuje měřit vzdálenosti mezi sledovanými epitopy [18].
Mezi potenciální nedostatky této metody patří především nemožnost využití PLA in vivo, což znemožňuje studovat dynamiku a kinetiku proteinových interakcí. Dále tato metoda není využitelná pro de novo identifikaci nových interakčních partnerů, ale pouze ověřuje interakce známých interakčních partnerů. Diskutabilní je také spolehlivost signálů vzniklých PLA in situ. Přestože jedním z cílů PLA techniky je ověření proteinových interakcí, signál může být potenciálně generován i párem proteinů, které se nacházejí ve velmi těsné blízkosti, ale bez vzájemných interakcí. Experimenty využívající malé molekuly přerušující protein‑proteinové vazby však prokázaly specifičnost PLA in situ detekovat skutečné interakce [18].
Závěrem lze konstatovat, že PLA in situ má značné možnosti využití v základním i aplikovaném výzkumu, při vývoji nových terapeutik a v klinické diagnostice, protože umožňuje komplexně porozumět buněčným procesům na úrovni proteinů, jejich lokalizace, modifikací, vzájemných protein‑proteinových interakcí nebo interakcí s jinými makromolekulami, jako jsou RNA a DNA.
Práce byla podpořena Evropským fondem pro regionální rozvoj a státním rozpočtem České republiky (OP VaVpI – RECAMO CZ.1.05/2.1.00/03.0101) a MZ ČR – RVO (MOÚ, 00209805) a BBMRI_CZ (LM2010004).
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
Mgr. Veronika Brychtová
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: vebrychtova@mou.cz
Obdrženo: 31. 1. 2014
Přijato: 25. 3. 2014
Zdroje
1. Hung MC, Link W. Protein localization in disease and therapy. J Cell Sci 2011; 124(20): 3381 – 3392. doi: 10.1242/ jcs.089110.
2. Fabbro M, Henderson BR. Regulation of tumor suppressors by nuclear ‑ cytoplasmic shuttling. Exp Cell Res 2003(2); 282 : 59 – 69.
3. Hu MC, Lee DF, Xia W et al. IkappaB kinase promotes tumorigenesis through inhibition of forkhead FOXO3a. Cell 2011; 117(2): 225 – 237.
4. Chen MF, Fang FM, Lu C et al. Significance of nuclear accumulation of Foxo3a in esophageal squamous cell carcinoma. Int J Radiat Oncol Biol Phys 2008; 71 : 1220 – 1229. doi: 10.1016/ j.ijrobp.2008.02.077.
5. Xia W, Chen J, Zhou X et al. Phosphorylation/ cytoplasmic localization of p21Cip1/ WAF1 is associated with HER2/ neu overexpression and provides a novel combination predictor for poor prognosis in breast cancer patients. Clin Cancer Res 2004; 10(11): 3815 – 3824.
6. Xia X, Ma Q, Li X et al. Cytoplasmic p21 is a potential predictor for cisplatin sensitivity in ovarian cancer. BMC Cancer 2011; 11 : 399. doi: 10.1186/ 1471 - 2407 - 11 - 399.
7. Liang J, Zubovitz J, Petrocelli T et al. PKB/ Akt phosphorylates p27, impairs nuclear import of p27 and opposes p27 - mediated G1 arrest. Nat Med 2002; 8(10): 1153 – 1160.
8. Nan KJ, Jing Z, Gong L. Expression and altered subcellular localization of the cyclin‑dependent kinase inhibitor p27Kip1 in hepatocellular carcinoma. World J Gastroenterol 2004; 10(10): 1425 – 1430.
9. Rosen DG, Yang G, Cai KQ et al. Subcellular localization of p27kip1 expression predicts poor prognosis in human ovarian cancer. Clin Cancer Res 2005; 11(1): 632 – 637.
10. Ogino S, Shima K, Nosho K et al. A cohort study of p27 localization in colon cancer, body mass index, and patient survival. Cancer Epidemiol Biomarkers Prev 2009; 18(6): 1849 – 1858. doi: 10.1158/ 1055-9965.EPI ‑ 09 - 0181.
11. Li C, Iida M, Dunn E et al. Nuclear EGFR contributes to acquired resistance to cetuximab. Oncogene 2009; 28 : 3801 – 3813. doi: 10.1038/ onc.2009.234.
12. Hsu S, Miller S, Wang Y et al. Nuclear EGFR is required for cisplatin resistance and DNA repair. Am J Transl Res 2009; 1(3): 249 – 258.
13. Weibrecht I, Leuchowius KJ, Clausson CM et al. Proximity ligation assays: a recent addition to the proteomics toolbox. Expert Rev Proteomics 2010; 7(3): 401 – 409. doi: 10.1586/ epr.10.10.
14. Pfleger KD, Eidne KA. Illuminating insights into protein‑protein interactions using bioluminescence resonance energy transfer (BRET). Nat Methods 2006; 3(3): 165 – 174.
15. Fredriksson S, Gullberg M, Jarvius J et al. Protein detection using proximity ‑ dependent DNA ligation assays. Nat Biotechnol 2002; 20(5): 473 – 477.
16. Gullberg M, Gústafsdóttir SM, Schallmeiner E et al. Cytokine detection by antibody‑based proximity ligation. Proc Natl Acad Sci USA 2004; 101(22): 8420 – 8424.
17. Nong RY, Wu D, Yan J et al. Solid ‑ phase proximity ligation assays for individual or parallel protein analyses with readout via real ‑ time PCR or sequencing. Nat Protoc 2013; 8(6): 1234 – 1248. doi: 10.1038/ nprot.2013.070.
18. Soderberg O, Gullberg M, Jarvius M et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nat Methods 2006; 3(12): 995 – 1000.
19. Jarvius M, Paulsson J, Weibrecht I et al. In situ detection of phosphorylated platelet ‑ derived growth factor receptor beta using a generalized proximity ligation method. Mol Cell Proteomics 2007; 6(9): 1500 – 1509.
20. Gu GJ, Friedman M, Jost C et al. Protein tag ‑ mediated conjugation of oligonucleotides to recombinant affinity binders for proximity ligation. N Biotechnol 2013; 144 – 152. doi: 10.1016/j.nbt.2012.05.005.
21. Gustafsdottir SM, Schlingemann J, Rada ‑ Iglesias A et al. In vitro analysis of DNA ‑ protein interactions by proximity ligation. Proc Natl Acad Sci USA 2007; 104(9): 3067 – 3072.
22. Gomez D, Shankman LS, Nguyen AT et al. Detection of histone modifications at specific gene loci in single cells in histological sections. Nat Methods 2013; 10(2): 171 – 177. doi: 10.1038/ nmeth.2332.
23. Koos B, Andersson L, Clausson CM et al. Analysis of protein interactions in situ by proximity ligation assays. Curr Top Microbiol Immunol 2014; 377 : 111 – 126. doi: 10.1007/ 82_2013_334.
24. Weibrecht I, Gavrilovic M, Lindbom L et al. Visualising individual sequence ‑ specific protein‑DNA interactions in situ. N Biotechnol 2012; 29(5): 589 – 598. doi: 10.1016/ j.nbt.2011.08.002.
25. Koos B, Paulsson J, Jarvius M et al. Platelet ‑ derived growth factor receptor expression and activation in choroid plexus tumors. Am J Pathol 2009; 175(4): 1631 – 1637. doi: 10.2353/ ajpath.2009.081022.
26. Pinto R, Carvalho AS, Conze T et al. Identification of new cancer biomarkers based on aberrant mucin glycoforms by in situ proximity ligation. J Cell Mol Med 2012; 16(7): 1474 – 1484. doi: 10.1111/ j.1582 - 4934.2011.01436.x.
27. Renfrow JJ, Scheck AC, Dhawan NS et al. Gene ‑ protein correlation in single cells. Neuro Oncol 2011; 13(8): 880 – 885. doi: 10.1093/ neuonc/ nor071.
28. Nilsson M, Malmgren H, Samiotaki M et al. Padlock probes: circularizing oligonucleotides for localized DNA detection. Science 1994; 265(5181): 2085 – 2088.
29. Leuchowius KJ, Jarvius M, Wickström M et al. High content screening for inhibitors of protein interactions and post‑translational modifications in primary cells by proximity ligation. Mol Cell Proteomics 2010; 9(1): 178 – 183. doi: 10.1074/ mcp.M900331-MCP200.
Štítky
Dětská onkologie Chirurgie všeobecná Onkologie
Článek vyšel v časopiseKlinická onkologie
Nejčtenější tento týden
2014 Číslo Supplementum- Nejlepší kůže je zdravá kůže: 3 úrovně ochrany v moderní péči o stomii
- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejasný stín na plicích – kazuistika
- Neodolpasse je bezpečný přípravek v krátkodobé léčbě bolesti
-
Všechny články tohoto čísla
- Analýza proteinů pomocí hmotnostní spektrometrie
- p‑ SRM, SWATH a HRM – cílené proteomické přístupy na hmotnostním spektrometru TripleTOF 5600+ a jejich aplikace v onkologickém výzkumu
- Analýza fosfoproteínov a signálnych dráh kvantitatívno‑ proteomickými metódami
- Nové trendy ve studiu glykosylace proteinů u onkologických onemocnění
- Současné trendy ve využívání PET radiofarmak k onkologické diagnostice
- „Techneciová krize“ – příčiny, možná řešení a dopad na diagnostiku planární scintigrafií a SPECT
- Vitamin D jako významný steroidní hormon u karcinomu prsu
- Využití průtokové cytometrie pro analýzu mitochondriální buněčné smrti
- Soutěž o nejlepší práci
- Metody studia buněčné migrace a invazivity nádorových buněk
- Možnosti studia transendoteliální migrace in vitro
- Editorial
- Mechanizmy lékové rezistence a nádorové kmenové buňky
- Funkční testy pro detekci nádorových kmenových buněk
- Mikroprostředí nádoru – možnosti výzkumu v podmínkách in vitro
- Elektrochemická analýza nukleových kyselin, bílkovin a polysacharidů v biomedicíně
- Sekvenování nové generace a možnosti jeho využití v onkologické praxi
- Vývoj metod založených na PCR a jejich aplikace v onkologickém výzkumu a praxi
- Programovaná buněčná smrt v nádorových buňkách
- Metody pro studium protein‑proteinových a protein‑ligandových interakcí
- Detekce protein‑proteinových interakcí metodami FRET a BRET
- Detekce proteinů, proteinových interakcí a modifikací s využitím „proximity ligation assay“ in situ
- Exprese a purifikace proteinů
- Kvantitativní hmotnostní spektrometrie a její využití v onkologii
- Klinická onkologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Exprese a purifikace proteinů
- Metody studia buněčné migrace a invazivity nádorových buněk
- Sekvenování nové generace a možnosti jeho využití v onkologické praxi
- Analýza proteinů pomocí hmotnostní spektrometrie
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Autoři: MDDr. Eleonóra Ivančová, PhD., MHA
Autoři: prof. MUDr. Eva Kubala Havrdová, DrSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



