-
Články
- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Využití mikroRNA ve slinách pro diagnostiku nádorových onemocnění
Use of Salivary MicroRNAs for Diagnosis of Solid Cancers
Background:
A modern approach to identify biomarkers of solid cancers in tissues and body fluids is based on microRNA (miRNA) expression profiling. miRNAs are a group of approximately 3.000 short noncoding RNAs containing 18–25 nucleotides that regulate gene expression at the post-transcriptional (mRNA) level. The abilities of miRNAs to inhibit the translation or induce degradation of oncogenes and tumor suppressors indicate that they are involved in carcinogenesis. There is increasing evidence that miRNAs regulate apoptosis, cell proliferation, differentiation, and invasion. miRNA expression profiles are therefore often analyzed for molecular diagnostics of solid cancers, similar to analyses based on mRNA profiling. It is important that miRNAs are highly stable and present at high levels in body fluids, including saliva, for analytic usage. miRNAs in saliva have been successfully tested as potential diagnostic biomarkers of many solid cancers. The main advantage of these miRNAs is that saliva samples can be collected non-invasively.Aim:
This review aims to summarize current knowledge of circulating miRNAs in solid cancers, with a focus on the use of miRNAs in saliva for oncology diagnostics.Key words:
microRNA – saliva – diagnosis – cancerThe results of this research have been acquired within CEITEC 2020 (LQ1601) project with fi nancial contribution made by the Ministry of Education, Youths and Sports of the Czech Republic within special support paid from the National Programme for Sustainability II funds.
The authors declare they have no potential confl icts of interest concerning drugs, products, or services used in the study.
The Editorial Board declares that the manuscript met the ICMJE recommendation for biomedical papers.
Submitted: 8. 3. 2018
Accepted: 15. 5. 2018
Autoři: Kubíčková Adéla 1; Slabý Ondřej 1,2
Působiště autorů: CEITEC – Středoevropský technologický institut, MU, Brno 1; Masarykův onkologický ústav, Brno 2
Vyšlo v časopise: Klin Onkol 2018; 31(4): 249-259
Kategorie: Přehled
doi: https://doi.org/10.14735/amko2018249Souhrn
Východiska:
Jedním z moderních přístupů identifikace biomarkerů nádorových onemocnění, a to jak na tkáňové úrovni, tak i v tělních tekutinách, je profilování exprese mikroRNA (miRNA). miRNA tvoří skupinu téměř 3 000 krátkých, 18–25 nukleotidů dlouhých nekódujících RNA. Slouží jako regulační prvky, které řídí expresi genů na posttranskripční úrovni, tj. na úrovni molekul mRNA. Schopnost miRNA inhibovat translaci či indukovat degradaci onkogenů a nádorových supresorů je podstatou jejich zapojení do procesů kancerogeneze. Důkazů o funkcích miRNA v regulaci procesů, jako jsou apoptóza, buněčná proliferace, diferenciace či invazivita, neustále přibývá. Analýza expresních profilů miRNA je proto stále častěji využívána pro účely molekulární diagnostiky nádorových onemocnění, analogicky jako je tomu u studií založených na profilování kódujících RNA. Z hlediska analytického využití je podstatná skutečnost, že miRNA jsou vysoce stabilní v tělních tekutinách vč. slin a vyskytují se zde v relativně vysokých hladinách. miRNA ve slinách již byly pro diagnostické účely úspěšně testovány u řady nádorových onemocnění, přičemž hlavní výhodou slin jako biologického materiálu je skutečnost, že jsou získatelné zcela neinvazivně.Cíl:
Cílem přehledového článku je shrnout dosavadní míru poznání z oblasti cirkulujících miRNA u nádorových onemocnění se zaměřením na využití miRNA ve slinách pro účely onkologické diagnostiky.Klíčová slova:
mikroRNA – sliny – diagnostika – nádorové onemocněníÚvod
MikroRNA (miRNA) jsou krátké nekódující molekuly RNA o délce 18–25 nukleotidů, které posttranskripčně regulují genovou expresi. miRNA byly popsány teprve na začáku 90. let 20. století a od té doby bylo zjištěno, že se v organizmech účastní biologických dějů, jako je např. proliferace, buněčný cyklus, apoptóza, diferenciace či invazivita. Význam miRNA v diagnostice a prognostikaci nádorových onemocnění proto neustále narůstá. Téměř polovina současného výzkumu v oblasti miRNA je zaměřena na studium deregulace miRNA u nádorových onemocnění. Včasná dia-gnostika mnoha nádorových onemocnění zvyšuje pravděpodobnost úspěšné léčby, jedním z hlavních cílů výzkumu miRNA v onkologii je proto také nalézení nových neinvazivních diagnostických biomarkerů.
miRNA vyskytující se v tělních tekutinách jako tzv. cirkulující miRNA byly popsány v krvi, a to v plazmě i séru, moči, mateřském mléku, slzách a slinách. Tělní tekutiny představují snáze dostupná diagnostická média, než jsou např. vzorky tkání, a proto sledování pozměněných hladin miRNA v tělních tekutinách u různých onemocnění nabízí velmi zajímavé možnosti využití.
V tomto přehledovém článku je věnována pozornost miRNA ve slinách, které vykazují odlišné hladiny u pacientů s nádorovým onemocněním v porovnání se zdravými kontrolami, případně mezi pacienty v různých stadiích onemocnění. Až do současnosti byly popsány odlišné hladiny miRNA ve slinách u nádorů hlavy a krku, jícnu, slinivky břišní, tlustého střeva a konečníku.
Sliny – využití v diagnostice
Sliny představují jednoduše dostupné diagnostické médium, neboť slinné žlázy jsou obklopeny krevními kapilárami a pro jejich vzájemnou vysokou propustnost může docházet k výměně molekul. Biomarkery původem z krve se proto mohou vylučovat do slin, jejichž odběr za účelem diagnostiky různých patologických stavů je jednoduchý a neinvazivní [1]. Sliny jsou oproti krvi médiem, které lze odebírat bez rizika i u zvláštních skupin pacientů, jako jsou děti, hemofilici nebo osoby užívající intravenózně narkotika [2].
Termín „salivaomics“ lze volně přeložit jako přístup založený na vysokokapacitních metodách zabývající se studiem molekul přítomných ve slinách s cílem odhalit potenciál těchto molekul v diagnostice různých patologických stavů. V současnosti tento termín zahrnuje studium šesti typů molekul, a to DNA (tedy genom a epigenom v případě metylovaných genů), mRNA a miRNA (transkriptom), metabolických látek (metabolom), proteinů (proteom) a mikrobů (mikrobiom) [3,4]. Využití slin v diagnostice bylo hojně popisováno i ve 20. století [5,6] a ve 21. století tento trend stále sílí. Mezi onemocnění, u kterých bylo popsáno využití slinných biomarkerů, patří např. onemocnění periodontu [7], riziko zubního kazu [8], nádorové bujení [9,10], autoimunitní choroby, jako je Sjörgenův syndrom [11] či celiakie [12], kardiovaskulární choroby [13], metabolické choroby [14] či infekční onemocnění [15].
Genom ve slinách člověka je tvořen jak lidskou, tak mikrobiální DNA. Genetické a epigenetické změny při patologických procesech mohou být tedy sledovány i ve slinách. Kupříkladu byly stanoveny odlišné stupně metylace promotorů ve slinách u pacientů s karcinomem dutiny ústní [16]. Proteom ve slinách je tvořen přibližně z 3 000 proteinů vykonávajících různé biologické funkce [17]. Proteiny jako biomarkery byly popsány v mnoha studiích, např. u sledování odpovědi pacientek na terapii karcinomu prsu [18] nebo u pacientů s periodontitidou [19]. Metabolom je soubor všech metabolitů, které také mohou být využívány jako biomarkery onemocnění. Například studie z roku 2010 popsala odlišné profily metabolitů u pacientů s nádory dutiny ústní, prsu a pankreatu [20], dále byl identifikován metabolom pacientů s neurodegenerativní demencí [21]. Mikrobiom představuje mikroorganizmy kolonizující dutinu ústní, kterých bylo identifikováno přes 600 druhů [22]. Existuje ale i studie zvyšující toto číslo na 10 000 druhů [23]. Sliny mohou být použity k diagnostice infekčních chorob nejen kultivačními, ale i molekulárními metodami [24,25]. Navíc bylo popsáno specifické složení mikrobiomu u neinfekčních onemocnění, jako je např. karcinom pankreatu [26], nebo dokonce obezita [27]. Transkriptom (mRNA) ve slinách byl popsán roku 2004 [28] a od té doby bylo publikováno několik studií zabývajících se mRNA biomarkery u nádorových a jiných systémových onemocnění. Specifický transkriptom byl popsán u karcinomu pankreatu [29] či plic [30], dále například u Sjörgenova syndromu [11].
miRNA ve slinách byly prokázány roku 2009, a to v souvislosti s dlaždicobuněčným karcinomem dutiny ústní (oral squamous cell carcinoma – OSCC) [31]. Zájem o deregulaci těchto krátkých nekódujících RNA ve slinách u různých onemocnění poté narostl. Deregulace miRNA ve slinách byla popsána u pacientů s autismem [32], se Sjörgenovým syndromem [33], a dokonce bylo navrženo, že by mohla souviset se stárnutím [34]. Nejvíce studií v dané oblasti ale sleduje deregulace miRNA v souvislosti s nádorovými onemocněními, o kterých také pojednává tento přehledový článek.
MikroRNA
miRNA jsou krátké nekódující molekuly RNA o délce 18–25 nukleotidů vznikající v buňce dvoustupňovým procesem. Poprvé byly miRNA popsány roku 1993 u Caenorhabditis elegans. Bylo zjištěno, že jeden ze zkoumaných genů, lin-4, nekóduje žádný protein, ale pouze transkripty o přibližné délce 22 nukleotidů [35], které jsou díky komplementárním sekvencím k opakovaným sekvencím v nepřekládané oblasti na 3‘ konci (3‘untranslated region – 3’UTR) oblasti genu lin-14 schopny regulovat jeho expresi [36]. Roku 2000 bylo objeveno, že i gen let-7 u C. elegans kóduje 21-nukleotidové RNA komplementární k elementům v 3’UTR genu lin-41, které regulují expresi tohoto genu [37]. Záhy bylo zjištěno, že gen let-7 se nachází i u dalších organizmů vč. člověka a bylo navrženo, že jeho transkripty, tedy přibližně 21nukleotidové RNA, mají původ v prekurzorech vlásenkovitých struktur [38]. Název mikroRNA (se zkratkou miRNA nebo miR) vznikl roku 2001, kdy byly ve třech laboratořích identifikovány další druhy těchto krátkých molekul [39–41].
Význam miRNA v nádorové transformaci u člověka jako první popsali Carlo Croce a George Calin, kteří zkoumali deleci na chromozomu v oblasti 13q14, která se vyskytuje u více než poloviny pacientů s chronickou lymfocytární leukemií. Zjistili, že v dané oblasti se nacházejí geny mir-15 a mir-16, jejichž snížená exprese by mohla vést ke vzniku leukemie [42]. Brzy poté byly identifikovány snížené hladiny miR-143 a miR-145 u pacientů s kolorektálním karcinomem (colorectal cancer – CRC) [43], nižší exprese let-7 byla zaznamenána u pacientů s rakovinou plic [44] a další miRNA se změněnou expresí byly popsány u glioblastomu [45] a u rakoviny prsu [46].
Od té doby až do současnosti bylo dle databáze miRBase popsáno 2 588 lidských miRNA (miRBase Sequence Database, Release 21, dostupná online na www.mirbase.org) [47], přičemž jejich význam při vzniku rakoviny neustále narůstá. Díky profilování miRNA a hlubokému sekvenování [47,48] přibývá důkazů o možnostech využití miRNA v diagnostice, prognostikaci, a dokonce terapii nádorových onemocnění [49–51].
Biogeneze miRNA je proces, kdy jsou geny mir přepisovány a překládány v tzv. primární miRNA (pri-miRNA), které jsou následně upraveny v tzv. prekurzorové miRNA (pre-miRNA) a poté ve zralé miRNA za účasti ribonukleáz a dalších proteinů [52–55]. Zralé jednořetězcové miRNA jsou vázány do umlčujícího komplexu indukovaného RNA (RNA-induced silencing complex – RISC), který je schopen regulace genové exprese na posttranskripční úrovni [56–58]. Regulace probíhá dvojím způsobem v závislosti na komplementaritě mezi miRNA a cílovou mRNA. Je-li komplementarita úplná nebo téměř úplná, dochází k degradaci cílové mRNA, kde se uplatňuje princip RNA interference [59–61]. Pokud je komplementarita pouze částečná, dochází k represi translace vazbou na 3’UTR cílové mRNA [36]. Hlavní biologické funkce miRNA zahrnují regulaci diferenciace a proliferace buněk [35], apoptózy [62], metabolizmu lipidů [63] a glukózy [64] či odpověď na stres [63]. Dysregulace a porušená funkce miRNA mohou tedy vést ke vzniku různých onemocnění, jako jsou např. kardiovaskulární [65], neurologické choroby [66] a převážně onemocnění nádorová [42], kde miRNA fungují jako onkogeny i jako nádorové supresory [67].
Cirkulující miRNA
Roku 2008 bylo poprvé popsáno, že se miRNA vyskytují v tělních tekutinách, konkrétně v plazmě a séru, ve vysoce stabilní formě [68,69]. Navíc byly toho roku detekovány zvýšené hladiny miRNA v séru pacientů s nádorovými onemocněními, jako je difuzní velkobuněčný B lymfom [70] a karcinom prostaty [71]. Přítomnost miRNA byla v následujících letech popsána i v dalších tělních tekutinách, a to ve slinách [31,72], moči [73], slzách či mateřském mléce [74,75]. Sekrece miRNA do tělních tekutin probíhá dvěma hlavními způsoby. Sekrece může být buď pasivní, kdy se miRNA uvolňují do cirkulace z poškozených buněk či tkání [69,71,76], např. při metastázování či chronických zánětech nebo z krátce žijících krevních buněk, jako jsou trombocyty a monocyty [77]. Druhým způsobem je aktivní sekrece. miRNA mohou být uvolňovány v extracelulárních vezikulech, jako jsou mikrovezikuly [78,79], mezi které se řadí i exozomy [80]. Další vezikuly obsahující cirkulující miRNA jsou apoptická tělíska [81]. Bylo zjištěno, že se miRNA mohou vyskytovat i mimo vezikuly, a to ve vazbě na proteiny Ago2 [82] a Ago1, Ago3, Ago4 [83], nukleofosmin 1 [84] a HDL [85]. Jednotlivé formy cirkulujících miRNA popisuje obr. 1 [86].
Obr. 1. Vznik a formy cirkulujících miRNA [86]. ![Vznik a formy cirkulujících miRNA [86].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image_pdf/b9dc4c1ef8b1bc735bbce54e7d8b1f7f.jpeg)
Cirkulující miRNA vykazují vysoký stupeň stability. Hladiny miRNA se nemění při inkubaci při pokojové teplotě [71], při 4 °C a uchovávání při –70 °C [87], zůstávají stabilní po mnoha cyklech zamražování a rozmražování, po varu a po působení vysokých a nízkých hodnot pH [69]. Odolávají působení RNáz, které jsou přítomny v krvi [69,71,88]. Předpokládá se, že stabilita miRNA v tělních tekutinách je zajištěna uzavřením v mikrovezikulech [78–80] a vazbou na proteiny [82,84,85]. Mikrovezikuly se zdají být odolnější proti působení RNáz než miRNA v komplexu s proteiny [89]. Byla také vyslovena hypotéza, že cirkulující miRNA může být chráněna modifikacemi. Mezi obecné modifikace miRNA patří metylace [90], adenylace [91] a uridylace [92]. Vychází se z předpokladu, že tatáž miRNA je odolnější vůči působení RNáz v cirkulaci než v tkáni či buňce, a proto je možné, že je tato odolnost zajištěna právě zmíněnými modifikacemi. Nicméně modifikované cirkulující miRNA ještě nebyly prokázány [77].
miRNA jako hormony
V souvislosti s objevem cirkulujících miRNA bylo navrženo, že miRNA mohou fungovat jako hormony, tedy jako signální molekuly přenášející informace mezi donorovými a recipientními buňkami či tkáněmi. První důkaz o této funkci miRNA byl podán ve studii z roku 2007, kdy bylo zjištěno, že miRNA jsou transportovány v exozomech a že tyto exozomy jsou předávány mezi buňkami [80]. Koncept cirkulujících miRNA jako hormonů byl popsán v mnoha studiích, počínaje rokem 2008, kdy Hunter et al prokázali přítomnost miRNA v mikrovezikulech v periferní krvi u zdravých jedinců a navrhli, že by tyto miRNA mohly regulovat homeostázu krevních buněk a jejich metabolické dráhy [78]. Následně Skog et al pozorovali mikrovezikuly pocházející z glioblastomu. Tyto mikrovezikuly obsahovaly proteiny, mRNA a miRNA a byly transformovány normálními buňkami v signály podporující nádorové bujení [93]. miRNA se v cirkulaci vyskytují i v apoptických tělískách. Tento objev přinesl také zjištění, že se konkrétně miR-126 může podílet na inhibici rozvoje aterosklerózy u myší [81] a v souladu s tímto zjištěním byly pozorovány nižší hladiny miR-126 u pacientů s koronární aterosklerózou [94].
Další možností transportu miRNA v cirkulaci je vazba na proteiny. Tyto miRNA jsou cílovými buňkami z cirkulace zachycovány pomocí receptorů, které rozeznávají proteiny vázající RNA [85,95]. Zatím bylo popsáno působení miRNA jako hormonu pouze v komplexu s HDL, kdy po přijetí takového komplexu recipientní buňkou došlo k posttranskripční regulaci příslušné mRNA [85].
Mnoho dalších studií se zabývá mezibuněčnou komunikací pomocí cirkulujících miRNA, a to především ve veziklech, neboť tyto miRNA by mohly být potenciálně využity k terapeutickým účelům [96]. Tyto objevy značí, že cirkulující miRNA významně ovlivňují buňky a tkáně v různých částech těla, a to dvojím způsobem – mohou napomáhat progresi onemocnění (destruktivní efekt), nebo před poškozením organizmu chránit (protektivní efekt) [97]. Tyto poznatky představují nové možnosti využití cirkulujících miRNA, a proto je v této oblasti veden intenzivní výzkum.
Metodické aspekty detekce cirkulujících miRNA
I přes značné výhody cirkulujících miRNA zůstává jejich detekce náročná. Pro významné rozdíly ve výsledcích izolace, měření a kvantifikace miRNA prováděných různými metodami neexistuje jednotný postup, který by byl aplikovatelný pro klinické využití cirkulujících miRNA jako biomarkerů. Tyto rozdíly jsou dány především odlišným zpracováním vzorků, neefektivní izolací, hemolýzou ve vzorcích krve, ne vždy spolehlivou účinností reverzní transkripce a polymerázové řetězové reakce (polymerase chain reaction – PCR), které se ke kvantifikaci miRNA užívají, či nejednotností v používání referenčních genů [96,98].
Prvním krokem v detekci miRNA z tělních tekutin je nakládání se samotným vzorkem. V případě krevních vzorků je situace komplikována skutečností, že trombocyty, leukocyty [78,99] i erytrocyty [100] obsahují vlastní miRNA, a je proto možné, že uměle zvyšují hladiny extracelulárních miRNA. Bylo dokázáno, že koncentrace miRNA se měnily při dlouhodobém uchovávání trombocytů [101] i erytrocytů [102] při pokojové teplotě a že hladiny miRNA ovlivňuje i hemolýza erytrocytů [103]. Zvyšování počtů krevních elementů při některých patologických podmínkách může také měnit koncentrace extracelulárních miRNA [104], stejně jako se miRNA mohou vyplavovat z trombocytů při srážení krve za účelem získání séra [105]. S tím se pojí i výběr samotného koagulačního činidla. Bylo zjištěno, že nejlepší volbou je kyselina ethylendiamintetraoctová (ethylenediaminetetraacetic acid – EDTA), neboť citrát i heparin, které se pro koagulaci také využívají, mohou inhibovat kvantitativní PCR (qPCR) [106,107]. Faktory jako např. hemolýza nejsou v případě slin relevantní, nicméně kontaminace vzorku miRNA pocházejícími z buněčných elementů slin (např. epiteliální buňky dutiny ústní, leukocyty či trombocyty) problém představuje.
Izolaci miRNA z tělních tekutin, konkrétně z plazmy a séra, komplikuje více faktorů. Je mezi nimi i přítomnost proteinů, které mohou zhoršit výtěžnost izolace a následně i ovlivnit průběh PCR [108]. miRNA se také v tělních tekutinách vyskytují ve velmi nízkých koncentracích [109]. Pro izolaci miRNA se používají různé komerční kity, jejichž vhodnost porovnávalo již mnoho studií. Jednotlivé kity se v úspěšnosti izolace extracelulární RNA odlišují [110,111].
Dalším krokem je kvantifikace izolované miRNA, která probíhá nejčastěji třemi způsoby. Nejvíce používanou metodou je qPCR, dále sekvenování nové generace či hybridizační metody s použitím mikročipů. Každá z těchto metod poskytuje jisté výhody i nevýhody, jako jsou např. rozdíly v senzitivitě a specificitě, kapacitě přístrojů či ceně [112]. Faktory ovlivňující detekci volných miRNA ve slinách jsou shrnuty v tab. 1.
Tab. 1. Metodické aspekty ovlivňující diagnostické využití miRNA ve slinách. 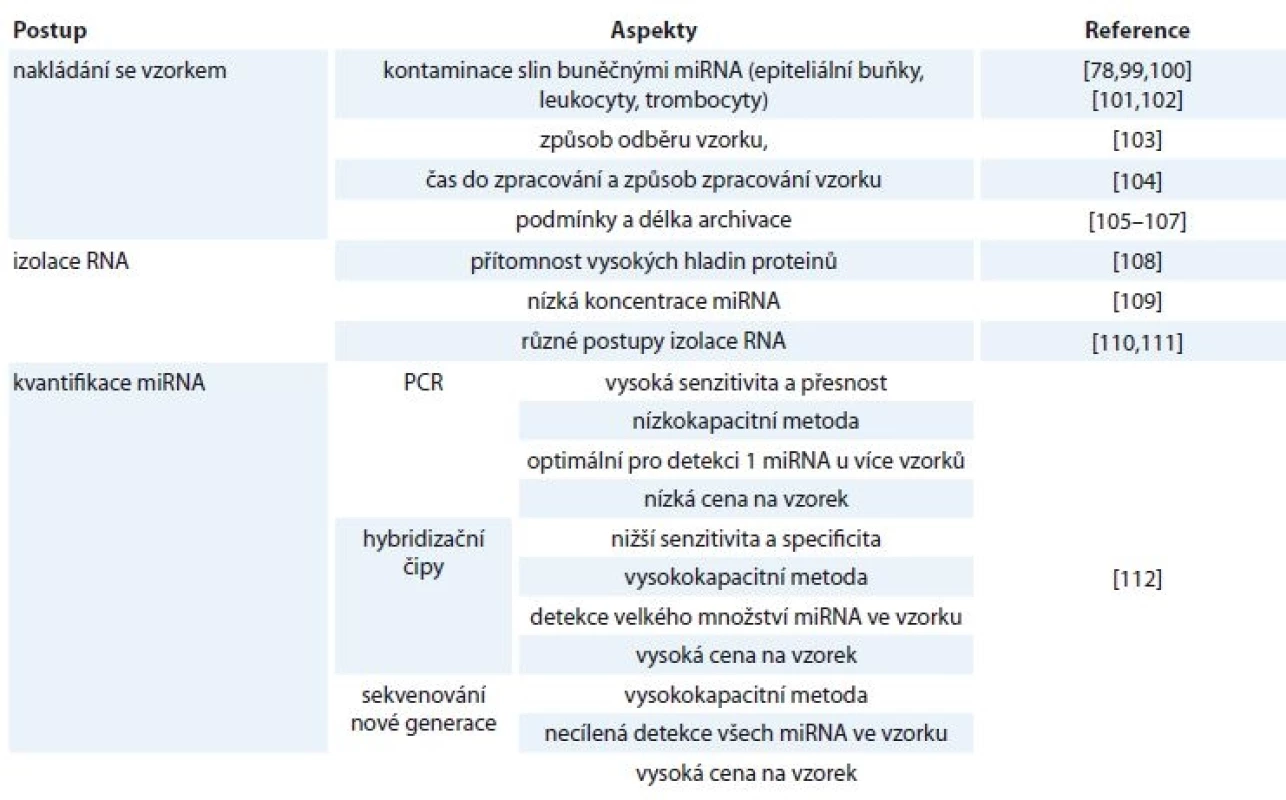
PCR – polymerázová řetězová reakce Diagnostické využití miRNA ve slinách
miRNA byly poprvé ve slinách detekovány roku 2009, kdy byla prokázána přítomnost miRNA ve slinách i ve slinném supernatantu [31]. miRNA byly následně ve slinách detekovány v exozomech [72], přičemž bylo zjištěno, že v této formě se vyskytují nejvíce [113]. Bylo navrženo, že by miRNA ve slinách mohly sloužit jako biomarkery nádorových a jiných onemocnění, především z oblasti hlavy a krku. Ideální biomarkery by měly být senzitivní a specifické, stabilní, měly by být produktem drah zapojených přímo do vzniku onemocnění, neovlivnitelné potravou, měřitelné v mezích detekce [114], dostupné neinvazivními metodami, specifické pro danou chorobu, měly by chorobu předpovídat ještě před nástupem klinických příznaků a biomarkery nalezené u modelových organizmů by měly být použitelné i u člověka [74]. Mezi RNA biomarkery řadíme i miRNA, a to tkáňové i extracelulární neboli cirkulující. V mnoha studiích byla popsána deregulace exprese miRNA při různých onemocněních vč. nádorových [115]. miRNA ve slinách se řadí mezi cirkulující miRNA. Tyto miRNA jsou vhodnými biomarkery nádorových onemocnění z mnoha důvodů. Jsou to méně komplexní molekuly bez posttranskripčních modifikací, jejich detekce a amplifikace je snadná a jejich sekvence jsou konzervované u člověka a u dalších modelových organizmů [74]. Změny jejich exprese se snadno detekují a jejich expresní profily jsou pro danou tělní tekutinu specifické [69]. miRNA ve slinách tyto podmínky splňují. Vyskytují se ve vysoce stabilní formě [116] a jejich exprese je u nádorových onemocnění deregulována [31]. Sliny samy o sobě jsou vhodným médiem pro využití v diagnostice onemocnění. Odběr slin je nenáročný, neinvazivní, vzorky se jednoduše zpracovávají, transportují i uchovávají [117].
Následující podkapitoly popisují jednotlivé studie zabývající se výskytem miRNA ve slinách ve spojitosti s nádorovými onemocněními. Souhrn všech deregulovaných miRNA poskytuje tab. 2.
Tab. 2. Přehled deregulovaných miRNA ve slinách u pacientů s nádorovými onemocněními. 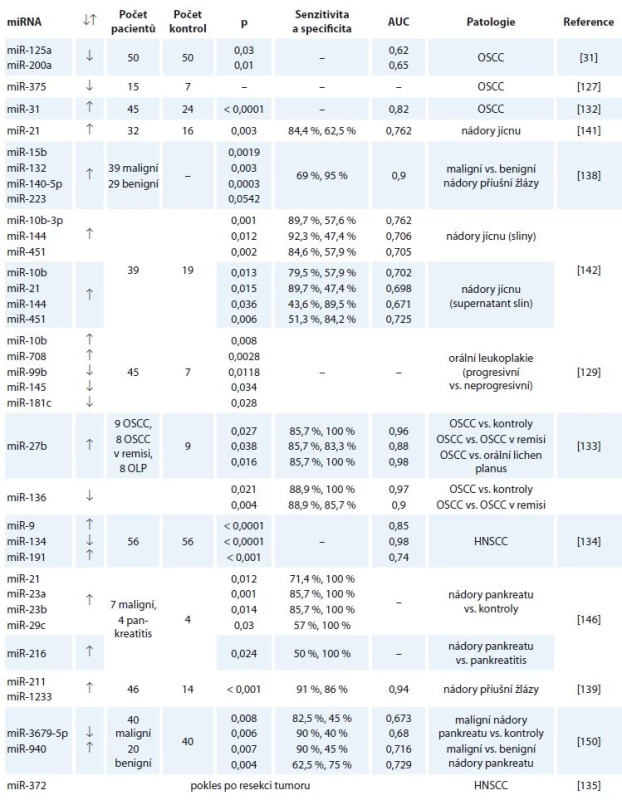
KO 2018/4 tab2b 
AUC – plocha pod křivkou, OSCC – dlaždicobuněčný karcinom dutiny ústní, OLP – orální lichen planus, HNSCC – dlaždicobuněčný karcinom hlavy a krku, TSCC – dlaždicobuněčný karcinom jazyka, CRC – kolorektální karcinom, ESCC – dlaždicobuněčný karcinom jícnu
↓↑ – snížená či zvýšená expreseNádory hlavy a krku
Nádory hlavy a krku zahrnují nádory dutiny ústní, faryngu (konkrétně nasofaryngu, orofaryngu a hypofaryngu), laryngu, dutiny nosní a vedlejších dutin nosních, velkých a malých slinných žláz [118]. Jedná se o velmi časté nádorové onemocnění, ročně je na celém světě diagnostikováno kolem 900 000 nových případů [119]. Nejčastějším buněčným typem je dlaždicobuněčný (spinocelulární) karcinom, který tvoří až 90 % všech nádorů hlavy a krku. Postihuje nejčastěji rty, dutinu ústní, hltan i hrtan [120]. Jedná se o maligní nádor vyvíjející se z dlaždicových buněk, které lemují dýchací a trávicí trakt a který může metastazovat lymfatickými cévami [121]. Protože je míra přežití dlaždicobuněčného karcinomu (squamous cell carcinoma – SCC velice nízká [120], hledají se nové způsoby časné detekce onemocnění a úspěšné léčby.
Spojitost mezi dlaždicobuněčným karcinomem hlavy a krku (head and neck squamous cell carcinoma – HNSCC) a miRNA byla objevena roku 2008, kdy byla tkáňová miR-21 analyzována jako onkogen podporující růst nádoru. Pro studii byli vybráni pacienti s nádory orofaryngu, dutiny ústní, laryngu a hypofaryngu [122]. miRNA ve slinách byla objevena o rok později, a to u pacientů s OSCC [31]. Od tohoto objevu se výzkumné týmy zaměřily na využití miRNA ve slinách jako biomarkerů pro diagnostiku HNSCC, zvláště pro OSCC, v jehož případě se sliny zdají být vhodnějším médiem než plazma či vzorky tkání, neboť karcinom přímo omývají [123]. Navíc ve slinách byly již pro diagnostiku OSCC biomarkery popsány, a to biomarkery proteinové [124], mRNA biomarkery [9] a DNA biomarkery [125].
OSCC tvoří až 90 % všech nádorů dutiny ústní. Jedná se o agresivní karcinom s vysokou mortalitou, nicméně při časné detekci onemocnění se míra přežití zvyšuje až na 90 % [126]. Při porovnání pacientů s OSCC se zdravými kontrolami byla poprvé zaznamenána odlišná exprese miRNA ve slinách roku 2009. Jednalo se o snížení hladin miR-200a a miR-125 u pacientů s OSCC. Bylo tedy navrženo, že by se tyto dvě miRNA mohly užívat jako neinvazivní a rychlé diagnostické markery v diagnostice OSCC [31]. Wiklund et al následně pozorovali aberantní expresi miR-375 a metylaci miR-200c-141 u OSCC jak ve tkáních, tak ve slinách [127].
OSCC se většinou vyvíjí z prekanceróz, mezi které patří např. leukoplakie. Časná detekce prekanceróz snižuje riziko rozvoje nádoru [128]. Byla proto zkoumána exprese miRNA ve slinách u prekanceróz dutiny ústní. První studie porovnávala deregulaci exprese miRNA ve slinách u progresivní a neprogresivní orální leukoplakie. Byla prokázána odlišná exprese 25 miRNA, přičemž signifikantní rozdíly mezi progresivními a neprogresivními leukoplakiemi vykazovaly miR-10b, miR-145, miR-708, miR-181c a miR-99b. Byla také srovnávána exprese miRNA ve tkáni pacientů s podobnými leukoplakiemi s expresí miRNA ve slinách a byla detekována deregulovaná exprese stejných miRNA, ačkoli hladiny miRNA ve slinách byly nižší [129]. Ve druhé studii bylo zjištěno, že zvýšená exprese miR-21, miR-145 a miR-184 může napomáhat časné diagnostice prekanceróz, které se maligně transformují v OSCC, přičemž pouze v případě miR-184 existuje statisticky signifikantní korelace [130]. Hung et al o rok později popsali, že hladiny miR-21a miR-31 jsou zvýšeny u pacientů s prekancerózami dutiny ústní [131], což je v souladu s výsledky studie z roku 2012, kdy byly zvýšené hladiny miR-31 ve slinách popsány u OSCC. V této studii bylo navíc pozorováno, že po excizi karcinomu hladiny miR-31 prudce poklesly [132].
Další poznatky poskytla rozsáhlá studie, ve které byly porovnávány vzorky slin od pacientů s OSCC, OSCC v remisi, orálním lichen planus a od zdravých kontrol. Nejdůležitějším nálezem byly zvýšené hladiny miR-27b u pacientů s OSCC oproti všem ostatním skupinám, což z miR-27b činí důležitý biomarker tohoto onemocnění. Byly pozorovány i snížené hladiny miR-136 u OSCC oproti zdravým kontrolám i OSCC v remisi [133].
Mezi další miRNA, které byly navrženy jako nové neinvazivní markery HNSCC, patří i miR-9, miR-191 a miR-134. Navíc byla popsána metoda pro izolaci velkého množství miRNA i z malého objemu slin [134]. Dále bylo objeveno, že miR-372 podporuje rozvoj HNSCC a že její hladiny po resekci tumoru klesly [135].
Mezi HNSCC, resp. OSCC, se řadí i dlaždicobuněčný karcinom jazyka (tongue squamous cell carcinoma – TSCC). Jazyk bývá postižen karcinomem ve 41 % případů karcinomů dutiny ústní [136]. Duz et al popsali snížené hladiny miR-193-5p u pacientů s TSCC oproti zdravým kontrolám. Nadto zjistili, že ve vzorcích slin odebraných po operaci tumoru se hladiny této miR vrátily k normálu, což by z miR-193-5p mohlo činit marker specifický pro TSCC [137].
První studie zkoumající deregulaci exprese miRNA ve slinách pacientů s karcinomy slinných žláz se zaměřila na karcinom příušní žlázy. V této studii byly popsány rozdíly exprese miRNA u benigních a maligních nádorů příušní žlázy. Největší specificitu, a tedy i možnost klinického využití, vykazovala kombinace navrhovaných markerů miR-140,miR-223, miR-15b a miR-132 [138]. Stejní autoři také sledovali rozdíly v expresi miRNA ve slinách u pacientů s různými histologickými typy nádorů příušní žlázy, a to benigními i maligními. Ve výsledku identifikovali sedm miRNA s vyšší expresí u pacientů s nádorem, z toho vybrali dvě miRNA, miR-1233 a miR-211, jako biomarkery nádorů příušní žlázy s 91% senzitivitou a 86% specificitou. V této studii bylo také zjištěno, že pět ze sedmi miRNA, které vykazovaly odlišnou expresi u pacientů s nádorem a u zdravých kontrol, nebylo exprimováno ve slinách získaných přímo z příušní žlázy. Z toho plyne, že by tyto miRNA neměly být specificky exprimovány či vylučovány postiženou příušní žlázou [139].
Nádory jícnu
Karcinom jícnu představoval v roce 2015 celosvětově jedenácté nejčastější nádorové onemocnění s incidencí 483 000 a šestou nejčastější příčinu úmrtí kvůli nádoru se 439 000 úmrtími za rok [119]. Nádory jícnu se dělí na dva hlavní typy – SCC vyvíjející se z epitelových buněk, a adenokarcinom, který mívá počátek v dolní části jícnu [140]. Z důvodu hledání alternativy k invazivním vyšetřením a nutnosti časné diagnostiky onemocnění bylo zkoumáno, zda by miRNA ve slinách nemohly představovat nové biomarkery karcinomu jícnu. První studie se zabývala expresí miRNA ve slinném supernatantu pacientů. Byly pozorovány zvýšené hladiny miR-21 [141]. V další studii byly analyzovány vzorky slin i supernatantu slin. Ve slinách byla zaznamenána zvýšená exprese miR-10b*, miR-144 a miR-451, v supernatantu kromě jmenovaných i exprese miR-21. Tyto miRNA byly tedy navrhnuty jako nové biomarkery karcinomu jícnu [142]. Další studie se zabývala miRNA ve slinách i tkáních u dlaždicobuněčného karcinomu jícnu (esophageal squamous cell carcinoma – ESCC). Byly pozorovány zvýšené hladiny miR-196a ve tkáních i ve slinách oproti zdravým kontrolám. Dále byla zkoumána funkce miR-196a v patogenezi ESCC. Byl vysloven názor, že se tato miRNA zapojuje do několika významných signálních drah, jako je např. regulace buněčného cyklu pomocí proteinu p53, a mohla by se proto podílet na inhibici apoptózy nádorových buněk [143].
Nádory slinivky břišní
Nádory slinivky břišní představovaly roku 2012 sedmou nejčastější příčinu úmrtí způsobenou nádorovým onemocněním [144]. Počet nových případů za rok 2015 se pohyboval okolo 425 000 [119]. Nejčastějším typem karcinomu pankreatu je duktální adenokarcinom se zastoupením 85 % [145]. Karcinom postupuje agresivně a neexistuje efektivní screening pro jeho časnou detekci. V důsledku toho se onemocnění až v 85 % případů dostane do pokročilých stadií, kdy je chirurgická léčba možná pouze u 15 % pacientů [146]. Pozdní stadia karcinomu nelze resekovat, protože již metastazuje ve vzdálenějších částech těla či vrůstá do cév [147]. Nalezení vhodného biomarkeru pro časnou detekci onemocnění by tedy mohlo zlepšit jeho prognózu.
První studie zabývající se vhodností miRNA jako biomarkerů nádorů pankreatu sledovaly a zaznamenaly změnu exprese miRNA v plazmě [148] a tkáni [149]. Pro četné výhody slin jako diagnostického média provedli Xie et al studii exprese miRNA ve slinách u pacientů s resekabilním karcinomem pankreatu a jako biomarkery vhodné pro diagnostiku onemocnění navrhli miR-3679-5p a miR-940. Rozdílná exprese těchto miRNA byla pozorována i při porovnání maligního karcinomu pankreatu s benigním [150]. Humeau et al u pacientů s neresekabilním adenokarcinomem pankreatu zaznamenali zvýšenou expresi miR-21, miR-23a, miR-23b a miR-29c oproti zdravým kontrolám. Kromě tohoto objevu navíc popsali miR-210 a let-7c jako biomarkery pankreatitidy a miR-216 jako miRNA, jejíž exprese odlišuje pankreatitidu od karcinomu, i když v tomto případě senzitivita dosahovala pouze 50 % [146].
Další studii zabývající se miRNA ve slinách u nádorů pankreatu publikovali Machida et al. Tato studie ovšem popisuje karcinomy pankreatobiliárního traktu, který zahrnuje kromě slinivky břišní i žlučník a mimojaterní žlučové cesty. Autoři se zaměřili na exozomální miRNA, přičemž vycházeli z předpokladu, že miRNA se ve slinách vyskytují převážně v exozomech [72,113] a že již byly popsány čtyři exozomální miRNA v séru u pacientů s karcinomem slinivky. Jednalo se o miR-1246, miR-4644, miR-3976 a miR-4306 [151]. Dvě z těchto exozomálních miRNA, miR-1246 a miR-4644, vykazovaly zvýšené hladiny exprese i ve vzorcích slin pacientů s pankreatobiliárním karcinomem. Bylo tedy navrženo, že tyto exozomální miRNA mají původ v postižené tkáni, neboť zvýšené hladiny miR-1246 u pacientů s pankreatobiliárním karcinomem byly pozorovány i ve tkáních [152], ze kterých se mohou vyplavovat do krve a z krve do slin. Nicméně limitací uvedené studie je fakt, že byli vybráni pouze pacienti s pokročilými stadii karcinomu. Aby byl screening karcinomu na podkladě miRNA biomarkerů efektivní, bylo by třeba provést další studie zkoumající expresní hladiny miRNA v časnějších stadiích vývoje karcinomu či u pacientů s chronickou pankreatitidou, ze které se karcinom často vyvíjí [153].
Nádory kolorekta
CRC představuje třetí nejčastější nádorové onemocnění na světě [119]. Souvislost mezi miRNA ve slinách a CRC popisuje v současnosti v anglické literatuře jediná studie, kdy byly prokázány zvýšené hladiny miR-21 u pacientů, a to nejen ve slinách, ale i v plazmě. Nicméně miR-21 ve slinách se jeví jako vhodnější biomarker pro screening CRC díky větší senzitivitě i specificitě, navíc sliny představují technicky jednodušší diagnostické médium [154].
Závěr
miRNA jsou důležité posttranskripční regulátory genové exprese a v tělních tekutinách, jako je krev, moč či sliny, představují důležité biomarkery stavu organizmu. V mnoha studiích bylo prokázáno, že se míra exprese miRNA u různých patologických stavů a onemocnění výrazně liší, přičemž nejvíce pozornosti je věnováno expresi miRNA u pacientů s nádorovými onemocněními. Porovnáváním expresních hladin miRNA u pacientů a zdravých kontrol lze identifikovat diagnostické biomarkery, porovnáním různých skupin pacientů pak biomarkery prognostické.
Využití cirkulujících miRNA ve slinách pro časnou diagnostiku a případně stanovení prognózy u pacientů s nádorovým onemocněním má nesporný potenciál pro zlepšení kvality života pacientů. U čtyř typů nádorových onemocnění, jejichž časná diagnostika je pro terapii klíčová, byla popsána významná deregulace exprese různých slinných miRNA. Zavedením metod využívajících detekce miRNA ve slinách jako biomarkerů těchto nádorových onemocnění by se tedy mohla urychlit diagnostika či příp. posoudit prognóza. Sliny navíc pro jednoduchost odběru, skladování i další zpracování představují vhodné neinvazivní diagnostické médium.
Nicméně aby slinné miRNA mohly být využívány v klinické praxi, je třeba zhodnotit vliv řady metodických aspektů způsobujících technologickou variabilitu při jejich detekci. Po nalezení optimálních analytických postupů a jejich standardizaci a po rozšíření studií na větší počet pacientů a nezávislých validacích výsledků by miRNA mohly sloužit jako užitečné biomarkery daných onemocnění.
Výsledky tohoto výzkumu byly získány v rámci projektu CEITEC 2020 (LQ1601) za fi nančního přispění Ministerstva školství, mládeže a tělovýchovy České republiky v rámci účelové podpory z prostředků Národního programu udržitelnosti II.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů
prof. RNDr. Ondřej Slabý, Ph.D.
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: on.slaby@gmail.com
Obdrženo: 8. 3. 2018
Přijato: 15. 5. 2018
Zdroje
1. Zhang CZ, Cheng XQ, Li JY et al. Saliva in the diagnosis of diseases. Int J Oral Sci 2016; 8 (3): 133–137. doi: 10.1038/ijos.2016.38.
2. Panat SR, Agarwal N, Kishore M et al. Sialochemistry – an emerging oral diagnostic tool. J Dent Sci Oral Rehabil 2013; 4 (1): 1–3.
3. Wong DT. Salivaomics. J Am Dent Assoc 2012; 143 (Suppl 10): 19S–24S.
4. Wang X, Kaczor-Urbanowicz KE, Wong DT. Salivary biomarkers in cancer detection. Med Oncol 2017; 34 (1): 7. doi: 10.1007/s12032-016-0863-4.
5. Mandel ID. Sialochemistry in diseases and clinical situations affecting salivary glands. Crit Rev Clin Lab Sci 1980; 12 (4): 321–366. doi: 10.3109/10408368009108733.
6. Streckfus CF, Bigler LR. Saliva as a diagnostic fluid. Oral Dis 2002; 8 (2): 69–76.
7. AlMoharib HS, AlMubarak A, AlRowis R et al. Oral fluid based biomarkers in periodontal disease: part 1. Saliva. J Int Oral Health 2014; 6 (4): 95–103.
8. Baughan LW, Robertello FJ, Sarrett DC et al. Salivary mucin as related to oral Streptococcus mutans in elderly people. Oral Microbiol Immunol 2000; 15 (1): 10–14.
9. Li Y, St John MA, Zhou X et al. Salivary transcriptome diagnostics for oral cancer detection. Clin Cancer Res 2004; 10 (24): 8442–8450. doi: 10.1158/1078-0432.CCR-04-1167.
10. St. John MA, Li Y, Zhou X et al. Interleukin 6 and interleukin 8 as potential biomarkers for oral cavity and oropharyngeal squamous cell carcinoma. Arch Otolaryngol Head Neck Surg 2004; 130 (8): 929–935. doi: 10.1001/archotol.130.8.929.
11. Streckfus C, Bigler L, Navazesh M et al. Cytokine concentrations in stimulated whole saliva among patients with primary Sjögren‘s syndrome, secondary Sjögren‘s syndrome, and patients with primary Sjögren‘s syndrome receiving varying doses of interferon for symptomatic treatment of the condition: a preliminary study. Clin Oral Investig 2001; 5 (2): 133–135.
12. Bonamico M, Nenna R, Montuori M et al. First salivary screening of celiac disease by detection of anti-transglutaminase autoantibody radioimmunoassay in 5000 Italian primary school children. J Pediatr Gastroenterol Nutr 2011; 52 (1): 17–20. doi: 10.1097/MPG.0b013e3181e6f2d0.
13. Floriano PN, Christodoulides N, Miller CS et al. Use of saliva-based nano-biochip tests for acute myocardial infarction at the point of care: a feasibility study. Clin Chem 2009; 55 (8): 1530–1538. doi: 10.1373/clinchem.2008.117713.
14. Rao PV, Reddy AP, Lu X et al. Proteomic identification of salivary biomarkers of type-2 diabetes. J Proteome Res 2009; 8 (1): 239–245. doi: 10.1021/pr8003776.
15. Amado LA, Villar LM, de Paula VS et al. Detection of hepatitis A, B, and C virus-specific antibodies using oral fluid for epidemiological studies. Mem Inst Oswaldo Cruz 2006; 101 (2): 149–155.
16. Viet CT, Schmidt BL. Methylation array analysis of preoperative and postoperative saliva DNA in oral cancer patients. Cancer Epidemiol Biomarkers Prev 2008; 17 (12): 3603–3611. doi: 10.1158/1055-9965.EPI-08-0507.
17. Sivadasan P, Gupta MK, Sathe GJ et al. Human salivary proteome-a resource of potential biomarkers for oral cancer. J Proteomics 2015; 127 (Pt A): 89–95. doi: 10.1016/j.jprot.2015.05.039.
18. Bigler LR, Streckfus CF, Copeland L et al. The potential use of saliva to detect recurrence of disease in women with breast carcinoma. J Oral Pathol Med 2002; 31 (7): 421–431.
19. Wu Y, Shu R, Luo LJ et al. Initial comparison of proteomic profiles of whole unstimulated saliva obtained from generalized aggressive periodontitis patients and healthy control subjects. J Periodontal Res 2009; 44 (5): 636–644. doi: 10.1111/j.1600-0765.2008.01172.x.
20. Sugimoto M, Wong DT, Hirayama A et al. Capillary electrophoresis mass spectrometry-based saliva metabolomics identified oral, breast and pancreatic cancer-specific profiles. Metabolomics 2010; 6 (1): 78–95. doi: 10.1007/s11306-009-0178-y.
21. Tsuruoka M, Hara J, Hirayama A et al. Capillary electrophoresis-mass spectrometry-based metabolome analysis of serum and saliva from neurodegenerative dementia patients. Electrophoresis 2013; 34 (19): 2865–2872. doi: 10.1002/elps.201300019.
22. Dewhirst FE, Chen T, Izard J et al. The human oral microbiome. J Bacteriol 2010; 192 (19): 5002–5017. doi: 10.1128/JB.00542-10.
23. Keijser BJ, Zaura E, Huse SM et al. Pyrosequencing analysis of the oral microflora of healthy adults. J Dent Res 2008; 87 (11): 1016–1020. doi: 10.1177/154405910808701104.
24. Liou TC, Chang TT, Young KC et al. Detection of HCV RNA in saliva, urine, seminal fluid, and ascites. J Med Virol 1992; 37 (3): 197–202.
25. Lazarevic V, Whiteson K, Gaïa N et al. Analysis of the salivary microbiome using culture-independent techniques. J Clin Bioinforma 2012; 2 : 4. doi: 10.1186/2043-9113-2-4.
26. Farrell JJ, Zhang L, Zhou H et al. Variations of oral microbiota are associated with pancreatic diseases including pancreatic cancer. Gut 2012; 61 (4): 582–588. doi: 10.1136/gutjnl-2011-300784.
27. Goodson JM, Groppo D, Halem S et al. Is obesity an oral bacterial disease? J Dent Res 2009; 88 (6): 519–523. doi: 10.1177/0022034509338353.
28. Li Y, Zhou X, St. John MA et al. RNA profiling of cell-free saliva using microarray technology. J Dent Res 2004; 83 (3): 199–203. doi: 10.1177/154405910408300303.
29. Zhang L, Farrell JJ, Zhou H et al. Salivary transcriptomic biomarkers for detection of resectable pancreatic cancer. Gastroenterology 2010; 138 (3): 949–957. e1–e7. doi: 10.1053/j.gastro.2009.11.010.
30. Zhang L, Xiao H, Zhou H et al. Development of transcriptomic biomarker signature in human saliva to detect lung cancer. Cell Mol Life Sci 2012; 69 (19): 3341–3350. doi: 10.1007/s00018-012-1027-0.
31. Park NJ, Zhou H, Elashoff D et al. Salivary microRNA: discovery, characterization, and clinical utility for oral cancer detection. Clin Cancer Res 2009; 15 (17): 5473–5477. doi: 10.1158/1078-0432.CCR-09-0736.
32. Hicks SD, Ignacio C, Gentile K et al. Salivary miRNA profiles identify children with autism spectrum disorder, correlate with adaptive behavior, and implicate ASD candidate genes involved in neurodevelopment. BMC Pediatr 2016; 16 : 52. doi: 10.1186/s12887-016-0586-x.
33. Alevizos I, Alexander S, Turner RJ et al. MicroRNA expression profiles as biomarkers of minor salivary gland inflammation and dysfunction in Sjögren‘s syndrome. Arthritis Rheum 2011; 63 (2): 535–544. doi: 10.1002/art.30131.
34. Machida T, Tomofuji T, Ekuni D et al. MicroRNAs in salivary exosome as potential biomarkers of aging. Int J Mol Sci 2015; 16 (9): 21294–21309. doi: 10.3390/ijms160921294.
35. Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell 1993; 75 (5): 843–854.
36. Wightman B, Ha I, Ruvkun G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell 1993; 75 (5): 855–862.
37. Reinhart BJ, Slack FJ, Basson M et al. The 21 nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature 2000; 403 (6772): 901–906. doi: 10.1038/35002607.
38. Pasquinelli AE, Reinhart BJ, Slack F et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature 2000; 408 (6808): 86–89. doi: 10.1038/35040556.
39. Lagos-Quintana M, Rauhut R, Lendeckel W et al. Identification of novel genes coding for small expressed RNAs. Science 2001; 294 (5543): 853–858. doi: 10.1126/science.1064921.
40. Lau NC, Lim LP, Weinstein EG et al. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans. Science 2001; 294 (5543): 858–862. doi: 10.1126/science.1065062.
41. Lee RC, Ambros V. An extensive class of small RNAs in Caenorhabditis elegans. Science 2001; 294 (5543): 862–864. doi: 10.1126/science.1065329.
42. Calin GA, Dumitru CD, Shimizu M et al. Frequent deletions and down-regulation of microRNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proc Natl Acad Sci U S A 2002; 99 (24): 15524–15529. doi: 10.1073/pnas.242606799.
43. Michael MZ, O’Connor SM, van Holst Pellekaan NG et al. Reduced accumulation of specific microRNAs in colorectal neoplasia. Mol Cancer Res 2003; 1 (12): 882–891.
44. Takamizawa J, Konishi H, Yanagisawa K et al. Reduced expression of the let-7 microRNAs in human lung cancers in association with shortened postoperative survival. Cancer Res 2004; 64 (11): 3753–3756. doi: 10.1158/0008-5472.CAN-04-0637.
45. Chan JA, Krichevsky AM, Kosik KS. MicroRNA-21 is an antiapoptotic factor in human glioblastoma cells. Cancer Res 2005; 65 (14): 6029–6033. doi: 10.1158/0008-5472.CAN-05-0137.
46. Iorio MV, Ferracin M, Liu CG et al. MicroRNA gene expression deregulation in human breast cancer. Cancer Res 2005; 65 (16): 7065–7070. doi: 10.1158/0008-5472.CAN-05-1783.
47. Kozomara A, Griffiths-Jones S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Res 2014; 42 (Database issue): D68–D73. doi: 10.1093/nar/gkt1181.
48. Lu J, Getz G, Miska EA et al. MicroRNA expression profiles classify human cancers. Nature 2005; 435 (7043): 834–838. doi: 10.1038/nature03702.
49. Calin GA, Liu CG, Sevignani C et al. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci U S A 2004; 101 (32): 11755–11760. doi: 10.1073/pnas.0404432101.
50. Calin GA, Ferracin M, Cimmino A et al. A MicroRNA signature associated with prognosis and progression in chronic lymphocytic leukemia. N Engl J Med 2005; 353 (17): 1793–1801. doi: 10.1056/NEJMoa050995.
51. Christopher AF, Kaur RP, Kaur G et al. MicroRNA therapeutics: Discovering novel targets and developing specific therapy. Perspect Clin Res 2016; 7 (2): 68–74. doi: 10.4103/2229-3485.179431.
52. Lee Y, Jeon K, Lee JT et al. MicroRNA maturation: stepwise processing and subcellular localization. EMBO J 2002; 21 (17): 4663–4670.
53. Lee Y, Ahn C, Han J et al. The nuclear RNase III Drosha initiates microRNA processing. Nature 2003; 425 (6956): 415–419. doi: 10.1038/nature01957.
54. Denli AM, Tops BB, Plasterk RH et al. Processing of primary microRNAs by the Microprocessor complex. Nature 2004; 432 (7014): 231–235. doi: 10.1038/nature03049.
55. Grishok A, Pasquinelli AE, Conte D et al. Genes and mechanisms related to RNA interference regulate expression of the small temporal RNAs that control C. elegans developmental timing. Cell 2001; 106 (1): 23–34.
56. Hammond SM, Bernstein E, Beach D et al. An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells. Nature 2000; 404 (6675): 293–296. doi: 10.1038/35005107.
57. Hutvágner G, Zamore PD. A microRNA in a multiple-turnover RNAi enzyme complex. Science 2002; 297 (5589): 2056–2060. doi: 10.1126/science.1073827.
58. Zeng Y, Yi R, Cullen BR. MicroRNAs and small interfering RNAs can inhibit mRNA expression by similar mechanisms. Proc Natl Acad Sci U S A 2003; 100 (17): 9779–9784. doi: 10.1073/pnas.1630797100.
59. Fire A, Xu S, Montgomery MK et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature 1998; 391 (6669): 806–811. doi: 10.1038/35888.
60. Elbashir SM, Lendeckel W, Tuschl T. RNA interference is mediated by 21 - and 22-nucleotide RNAs. Genes Dev 2001; 15 (2): 188–200.
61. Meister G, Landthaler M, Patkaniowska A et al. Human Argonaute2 mediates RNA cleavage targeted by miRNAs and siRNAs. Mol Cell 2004; 15 (2): 185–197. doi: 10.1016/j.molcel.2004.07.007.
62. Brennecke J, Hipfner DR, Stark A et al. Bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila. Cell 2003; 113 (1): 25–36.
63. Xu P, Vernooy SY, Guo M et al. The Drosophila microRNA Mir-14 suppresses cell death and is required for normal fat metabolism. Curr Biol 2003; 13 (9): 790–795.
64. Poy MN, Eliasson L, Krutzfeldt J et al. A pancreatic islet-specific microRNA regulates insulin secretion. Nature 2004; 432 (7014): 226–230. doi: 10.1038/nature03076.
65. van Rooij E, Sutherland LB, Liu N et al. A signature pattern of stress-responsive microRNAs that can evoke cardiac hypertrophy and heart failure. Proc Natl Acad Sci U S A 2006; 103 (48): 18255–18260. doi: 10.1073/pnas.0608791103.
66. Caudy AA, Myers M, Hannon GJ et al. Fragile X-related protein and VIG associate with the RNA interference machinery. Genes Dev 2002; 16 (19): 2491–2496. doi: 10.1101/gad.1025202.
67. Calin GA, Sevignani C, Dumitru CD et al. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proc Natl Acad Sci U S A 2004; 101 (9): 2999–3004. doi: 10.1073/pnas.0307323101.
68. Chim SS, Shing TK, Hung EC et al. Detection and characterization of placental microRNAs in maternal plasma. Clin Chem 2008; 54 (3): 482–490. doi: 10.1373/clinchem.2007.097972.
69. Chen X, Ba Y, Ma L et al. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res 2008; 18 (10): 997–1006. doi: 10.1038/cr.2008.282.
70. Lawrie CH, Gal S, Dunlop HM et al. Detection of elevated levels of tumour-associated microRNAs in serum of patients with diffuse large B-cell lymphoma. Br J Haematol 2008; 141 (5): 672–675. doi: 10.1111/j.1365-2141.2008.07077.x.
71. Mitchell PS, Parkin RK, Kroh EM et al. Circulating microRNAs as stable blood-based markers for cancer detection. Proc Natl Acad Sci U S A 2008; 105 (30): 10513–10518. doi: 10.1073/pnas.0804549105.
72. Michael A, Bajracharya SD, Yuen PS et al. Exosomes from human saliva as a source of microRNA biomarkers. Oral Dis 2010; 16 (1): 34–38. doi: 10.1111/j.1601-0825.2009.01604.x.
73. Hanke M, Hoefig K, Merz H et al. A robust methodology to study urine microRNA as tumor marker: microRNA-126 and microRNA-182 are related to urinary bladder cancer. Urol Oncol 2010; 28 (6): 655–661. doi: 10.1016/j.urolonc.2009.01.027.
74. Weber JA, Baxter DH, Zhang S et al. The microRNA spectrum in 12 body fluids. Clin Chem 2010; 56 (11): 1733–1741. doi: 10.1373/clinchem.2010.147405.
75. Kosaka N, Izumi H, Sekine K et al. MicroRNA as a new immune-regulatory agent in breast milk. Silence 2010; 1 (1): 7. doi: 10.1186/1758-907X-1-7.
76. Laterza OF, Lim L, Garrett-Engele PW et al. Plasma microRNAs as sensitive and specific biomarkers of tissue injury. Clin Chem 2009; 55 (11): 1977–1983. doi: 10.1373/clinchem.2009.131797.
77. Chen X, Zhang J, Zen K et al. MicroRNAs as blood-based biomarkers of cancer. In: Cho WCS (eds). MicroRNAs in Cancer Translational Research. Dordrecht: Springer Netherlands 2011 : 499–532.
78. Hunter MP, Ismail N, Zhang X et al. Detection of microRNA expression in human peripheral blood microvesicles. PloS One 2008; 3 (11): e3694. doi: 10.1371/journal.pone.0003694.
79. Zhang Y, Liu D, Chen X et al. Secreted monocytic miR-150 enhances targeted endothelial cell migration. Mol Cell 2010; 39 (1): 133–144. doi: 10.1016/j.molcel.2010.06.010.
80. Valadi H, Ekström K, Bossios A et al. Exosome-mediated transfer of mRNAs and microRNAs is a novel mechanism of genetic exchange between cells. Nat Cell Biol 2007; 9 (6): 654–659. doi: 10.1038/ncb1596.
81. Zernecke A, Bidzhekov K, Noels H et al. Delivery of microRNA-126 by apoptotic bodies induces CXCL12-dependent vascular protection. Sci Signal 2009; 2 (100): ra81. doi: 10.1126/scisignal.2000610.
82. Arroyo JD, Chevillet JR, Kroh EM et al. Argonaute2 complexes carry a population of circulating microRNAs independent of vesicles in human plasma. Proc Natl Acad Sci U S A 2011; 108 (12): 5003–5008. doi: 10.1073/pnas.1019055108.
83. Turchinovich A, Weiz L, Langheinz A et al. Characterization of extracellular circulating microRNA. Nucleic Acids Res 2011; 39 (16): 7223–7233. doi: 10.1093/nar/gkr254.
84. Wang K, Zhang S, Weber J et al. Export of microRNAs and microRNA-protective protein by mammalian cells. Nucleic Acids Res 2010; 38 (20): 7248–7259. doi: 10.1093/nar/gkq601.
85. Vickers KC, Palmisano BT, Shoucri BM et al. MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nat Cell Biol 2011; 13 (4): 423–433. doi: 10.1038/ncb2210.
86. Fendler A, Stephan C, Yousef GM et al. The translational potential of microRNAs as biofluid markers of urological tumours. Nat Rev Urol 2016; 13 (12): 734–752. doi: 10.1038/nrurol.2016.193.
87. Taylor DD, Gercel-Taylor C. MicroRNA signatures of tumor-derived exosomes as diagnostic biomarkers of ovarian cancer. Gynecol Oncol 2008; 110 (1): 13–21. doi: 10.1016/j.ygyno.2008.04.033.
88. Tsui NB, Ng EK, Lo YM. Stability of endogenous and added RNA in blood specimens, serum, and plasma. Clin Chem 2002; 48 (10): 1647–1653.
89. Köberle V, Pleli T, Schmithals C et al. Differential stability of cell-free circulating microRNAs: implications for their utilization as biomarkers. PLoS One 2013; 8 (9): e75184. doi: 10.1371/journal.pone.0075184.
90. Yu B, Yang Z, Li J et al. Methylation as a crucial step in plant microRNA biogenesis. Science 2005; 307 (5711): 932–935. doi: 10.1126/science.1107130.
91. Katoh T, Sakaguchi Y, Miyauchi K et al. Selective stabilization of mammalian microRNAs by 3‘ adenylation mediated by the cytoplasmic poly (A) polymerase GLD-2. Genes Dev 2009; 23 (4): 433–438. doi: 10.1101/gad.1761509.
92. Jones MR, Quinton LJ, Blahna MT et al. Zcchc11-dependent uridylation of microRNA directs cytokine expression. Nat Cell Biol 2009; 11 (9): 1157–1163. doi: 10.1038/ncb1931.
93. Skog J, Würdinger T, van Rijn S et al. Glioblastoma microvesicles transport RNA and proteins that promote tumour growth and provide diagnostic biomarkers. Nat Cell Biol 2008; 10 (12): 1470–1476. doi: 10.1038/ncb1800.
94. Fichtlscherer S, De Rosa S, Fox H et al. Circulating microRNAs in patients with coronary artery disease. Circ Res 2010; 107 (5): 677–684. doi: 10.1161/CIRCRESAHA.109.215566.
95. Chen X, Liang H, Zhang J et al. Secreted microRNAs: a new form of intercellular communication. Trends Cell Biol 2012; 22 (3): 125–132. doi: 10.1016/j.tcb.2011.12.001.
96. Hruštincová A, Votavová H, Dostálová Merkerová M. Circulating microRNAs: methodological aspects in detection of these biomarkers. Folia Biol 2015; 61 (6): 203–218.
97. Shah MY, Calin GA. The mix of two worlds: non-coding RNAs and hormones. Nucleic Acid Ther 2013; 23 (1): 2–8. doi: 10.1089/nat.2012.0375.
98. Moldovan L, Batte KE, Trgovcich J et al. Methodological challenges in utilizing miRNAs as circulating biomarkers. J Cell Mol Med 2014; 18 (3): 371–390. doi: 10.1111/jcmm.12236.
99. Landry P, Plante I, Ouellet DL et al. Existence of a microRNA pathway in anucleate platelets. Nat Struct Mol Biol 2009; 16 (9): 961–966. doi: 10.1038/nsmb.1651.
100. Chen SY, Wang Y, Telen MJ et al. The genomic analysis of erythrocyte microRNA expression in sickle cell diseases. PLoS One 2008; 3 (6): e2360. doi: 10.1371/journal.pone.0002360.
101. Kannan M, Mohan KV, Kulkarni S et al. Membrane array-based differential profiling of platelets during storage for 52 miRNAs associated with apoptosis. Transfusion 2009; 49 (7): 1443–1450. doi: 10.1111/j.1537-2995.2009.02140.x.
102. Kannan M, Atreya C. Differential profiling of human red blood cells during storage for 52 selected microRNAs. Transfusion 2010; 50 (7): 1581–1588. doi: 10.1111/j.1537-2995.2010.02585.x.
103. Kirschner MB, Kao SC, Edelman JJ et al. Haemolysis during sample preparation alters microRNA content of plasma. PLoS One 2011; 6 (9): e24145. doi: 10.1371/journal.pone.0024145.
104. Pritchard CC, Kroh E, Wood B et al. Blood cell origin of circulating microRNAs: a cautionary note for cancer biomarker studies. Cancer Prev Res 2012; 5 (3): 492–497. doi: 10.1158/1940-6207.CAPR-11-0370.
105. Wang K, Yuan Y, Cho JH et al. Comparing the microRNA spectrum between serum and plasma. PLoS One 2012; 7 (7): e41561. doi: 10.1371/journal.pone.0041561.
106. Holodniy M, Kim S, Katzenstein D et al. Inhibition of human immunodeficiency virus gene amplification by heparin. J Clin Microbiol 1991; 29 (4): 676–679.
107. García ME, Blanco JL, Caballero J et al. Anticoagulants interfere with PCR used to diagnose invasive aspergillosis. J Clin Microbiol 2002; 40 (4): 1567–1568.
108. Kroh EM, Parkin RK, Mitchell PS et al. Analysis of circulating microRNA biomarkers in plasma and serum using quantitative reverse transcription-PCR (qRT-PCR). Methods 2010; 50 (4): 298–301. doi: 10.1016/j.ymeth.2010.01.032.
109. Andreasen D, Fog JU, Biggs W et al. ImprovedmicroRNA quantification in total RNA from clinical samples. Methods 2010; 50 (4): S6–S9. doi: 10.1016/j.ymeth.2010.01.006.
110. Brunet-Vega A, Pericay C, Quílez ME et al. Variability in microRNA recovery from plasma: comparison of five commercial kits. Anal Biochem 2015; 488 : 28–35. doi: 10.1016/j.ab.2015.07.018.
111. Guo Y, Vickers K, Xiong Y et al. Comprehensive evaluation of extracellular small RNA isolation methods from serum in high throughput sequencing. BMC Genomics 2017; 18 (1): 50. doi: 10.1186/s12864-016-3470-z.
112. Chen M, Calin GA, Meng QH. Circulating microRNAs as promising tumor biomarkers. Adv Clin Chem 2014; 67 : 189–214. doi: 10.1016/bs.acc.2014.09.007.
113. Gallo A, Tandon M, Alevizos I et al. The majority of microRNAs detectable in serum and saliva is concentrated in exosomes. PLoS One 2012; 7 (3): e30679. doi: 10.1371/journal.pone.0030679.
114. Griffiths HR, Møller L, Bartosz G et al. Biomarkers. Mol Aspects Med 2002; 23 (1–3): 101–208.
115. Iorio MV, Croce CM. MicroRNA dysregulation in cancer: diagnostics, monitoring and therapeutics. A comprehensive review. EMBO Mol Med 2012; 4 (3): 143–159. doi: 10.1002/emmm.201100209.
116. Patel RS, Jakymiw A, Yao B et al. High resolution of microRNA signatures in human whole saliva. Arch Oral Biol 2011; 56 (12): 1506–1513. doi: 10.1016/j.archoralbio.2011.05.015.
117. Yoshizawa JM, Wong DT. Salivary microRNAs and oral cancer detection. Methods Mol Biol 2013; 936 : 313–324. doi: 10.1007/978-1-62703-083-0_24.
118. Rothenberg SM, Ellisen LW. The molecular pathogenesis of head and neck squamous cell carcinoma. J Clin Invest 2012; 122 (6): 1951–1957.
119. Global Burden of Disease Cancer Collaboration, Fitzmaurice C, Allen C et al. Global, regional, and national cancer incidence, mortality, years of life lost, years lived with disability, and disability-adjusted life-years for 32 cancer groups, 1990 to 2015: a systematic analysis for the global burden of disease study. JAMA Oncol 2017; 3 (4): 524–548. doi: 10.1001/jamaoncol.2016.5688.
120. Cooper JS, Porter K, Mallin K et al. National Cancer Database report on cancer of the head and neck: 10-year update. Head Neck 2009; 31 (6): 748–758. doi: 10.1002/hed.21022.
121. Leemans CR, Braakhuis BJ, Brakenhoff RH. The molecular biology of head and neck cancer. Nat Rev Cancer 2011; 11 (1): 9–22. doi: 10.1038/nrc2982.
122. Chang SS, Jiang WW, Smith I et al. MicroRNA alterations in head and neck squamous cell carcinoma. Int J Cancer 2008; 123 (12): 2791–2797. doi: 10.1002/ijc.23831.
123. Korostoff A, Reder L, Masood R et al. The role of salivary cytokine biomarkers in tongue cancer invasion and mortality. Oral Oncol 2011; 47 (4): 282–287. doi: 10.1016/j.oraloncology.2011.02.006.
124. Warnakulasuriya S, Soussi T, Maher R et al. Expression of p53 in oral squamous cell carcinoma is associated with the presence of IgG and IgA p53 autoantibodies in sera and saliva of the patients. J Pathol 2000; 192 (1): 52–57. doi: 10.1002/1096-9896 (2000) 9999 : 9999<:: AID-PATH669>3.0.CO; 2-C.
125. Liao PH, Chang YC, Huang MF et al. Mutation of p53 gene codon 63 in saliva as a molecular marker for oral squamous cell carcinomas. Oral Oncol 2000; 36 (3): 272–276.
126. Radhika T, Jeddy N, Nithya S et al. Salivary biomarkers in oral squamous cell carcinoma – an insight. J Oral Biol Craniofac Res 2016; 6 (Suppl 1): S51–S54. doi: 10.1016/j.jobcr.2016.07.003.
127. Wiklund ED, Gao S, Hulf T et al. MicroRNA alterations and associated aberrant DNA methylation patterns across multiple sample types in oral squamous cell carcinoma. PLoS One 2011; 6 (11): e27840. doi: 10.1371/journal.pone.0027840.
128. George A, Sreenivasan BS, Sunil S et al. Potentially malignant disorders of oral cavity. J Oral Maxillofac Pathol 2011; 2 (1): 95–100.
129. Yang Y, Li YX, Yang X et al. Progress risk assessment of oral premalignant lesions with saliva miRNA analysis. BMC Cancer 2013; 13 : 129. doi: 10.1186/1471-2407-13-129.
130. Zahran F, Ghalwash D, Shaker O et al. Salivary microRNAs in oral cancer. Oral Dis 2015; 21 (6): 739–747. doi: 10.1111/odi.12340.
131. Hung KF, Liu CJ, Chiu PC et al. MicroRNA-31 upregulation predicts increased risk of progression of oral potentially malignant disorder. Oral Oncol 2016; 53 : 42–47. doi: 10.1016/j.oraloncology.2015.11.017.
132. Liu CJ, Lin SC, Yang CC et al. Exploiting salivary miR-31 as a clinical biomarker of oral squamous cell carcinoma. Head Neck 2012; 34 (2): 219–224. doi: 10.1002/hed.21713.
133. Momen-Heravi F, Trachtenberg AJ, Kuo WP et al. Genomewide study of salivary microRNAs for detection of oral cancer. J Dent Res 2014; 93 (Suppl 7): 86S–93S. doi: 10.1177/0022034514531018.
134. Salazar C, Nagadia R, Pandit P et al. A novel saliva-based microRNA biomarker panel to detect head and neck cancers. Cell Oncol 2014; 37 (5): 331–338. doi: 10.1007/s13402-014-0188-2.
135. Yeh LY, Liu CJ, Wong YK et al. miR-372 inhibits p62 in head and neck squamous cell carcinoma in vitro and in vivo. Oncotarget 2015; 6 (8): 6062–6075. doi: 10.18632/oncotarget.3340.
136. Patel RS, Clark JR, Dirven R et al. Prognostic factors in the surgical treatment of patients with oral carcinoma. ANZ J Surg 2009; 79 (1–2): 19–22. doi: 10.1111/j.1445-2197.2008.04791.x.
137. Duz MB, Karatas OF, Guzel E et al. Identification of miR-139-5p as a saliva biomarker for tongue squamous cell carcinoma: a pilot study. Cell Oncol 2016; 39 (2): 187–193. doi: 10.1007/s13402-015-0259-z.
138. Matse JH, Yoshizawa J, Wang X et al. Discovery and prevalidation of salivary extracellular microRNA biomarkers panel for the noninvasive detection of benign and malignant parotid gland tumors. Clin Cancer Res 2013; 19 (11): 3032–3038. doi: 10.1158/1078-0432.CCR-12-3505.
139. Matse JH, Yoshizawa J, Wang X et al. Human salivary micro-RNA in patients with parotid salivary gland neoplasms. PLoS One 2015; 10 (11): e0142264. doi: 10.1158/1078-0432.CCR-12-3505.
140. David S, Meltzer S. MicroRNA involvement in esophageal carcinogenesis. Curr Opin Pharmacol 2011; 11 (6): 612–616. doi: 10.1016/j.coph.2011.09.006.
141. Xie ZJ, Chen G, Zhang XC et al. Saliva supernatant miR-21: a novel potential biomarker for esophageal cancer detection. Asian Pac J Cancer Prev 2012; 13 (12): 6145–6149.
142. Xie Z, Chen G, Zhang X et al. Salivary microRNAs as promising biomarkers for detection of esophageal cancer. PLoS One 2013; 8 (4): e57502. doi: 10.1371/journal.pone.0057502.
143. Fendereski M, Zia MF, Shafiee M et al. MicroRNA-196a as a potential diagnostic biomarker for esophageal squamous cell carcinoma. Cancer Invest 2017; 35 (2): 78–84. doi: 10.1080/07357907.2016.1254228.
144. Ferlay J, Soerjomataram I, Dikshit R et al. Cancer incidence and mortality worldwide: sources, methods and major patterns in GLOBOCAN 2012. Int J Cancer 2015; 136 (5): E359–386. doi: 10.1002/ijc.29210.
145. Howlader N, Noone AM, Krapcho M (eds). SEER Cancer Statistics Review, 1975–2013. [online]. Available from: https: //seer.cancer.gov/csr/1975_2013/.
146. Humeau M, Vignolle-Vidoni A, Sicard F et al. Salivary microRNA in pancreatic cancer patients. PLoS One 2015; 10 (6): e0130996. doi: 10.1371/journal.pone.0130996.
147. Callery MP, Chang KJ, Fishman EK et al. Pretreatment assessment of resectable and borderline resectable pancreatic cancer: expert consensus statement. Ann Surg Oncol 2009; 16 (7): 1727–1733. doi: 10.1245/s10434-009-0408-6.
148. Wang J, Chen J, Chang P et al. MicroRNAs in plasma of pancreatic ductal adenocarcinoma patients as novel blood-based biomarkers of disease. Cancer Prev Res 2009; 2 (9): 807–813. doi: 10.1158/1940-6207.CAPR-09-0094.
149. Roldo C, Missiaglia E, Hagan JP et al. MicroRNA expression abnormalities in pancreatic endocrine and acinar tumors are associated with distinctive pathologic features and clinical behavior. J Clin Oncol 2006; 24 (29): 4677–4684. doi: 10.1200/JCO.2005.05.5194.
150. Xie Z, Yin X, Gong B et al. Salivary microRNAs show potential as a noninvasive biomarker for detecting resectable pancreatic cancer. Cancer Prev Res 2015; 8 (2): 165–173. doi: 10.1158/1940-6207.CAPR-14-0192.
151. Madhavan B, Yue S, Galli U et al. Combined evaluation of a panel of protein and miRNA serum-exosome biomarkers for pancreatic cancer diagnosis increases sensitivity and specificity. Int J Cancer 2015; 136 (11): 2616–2627. doi: 10.1002/ijc.29324.
152. Ali S, Saleh H, Sethi S et al. MicroRNA profiling of diagnostic needle aspirates from patients with pancreatic cancer. Br J Cancer 2012; 107 (8): 1354–1360. doi: 10.1038/bjc.2012.383.
153. Machida T, Tomofuji T, Maruyama T et al. miR 1246 and miR 4644 in salivary exosome as potential biomarkers for pancreatobiliary tract cancer. Oncol Rep 2016; 36 (4): 2375–2381. doi: 10.3892/or.2016.5021.
154. Sazanov AA, Kiselyova EV, Zakharenko AA et al. Plasma and saliva miR-21 expression in colorectal cancer patients. J Appl Genet 2017; 58 (2): 231–237. doi: 10.1007/s13353-016-0379-9.
Štítky
Dětská onkologie Chirurgie všeobecná Onkologie
Článek Precizovaná onkologie
Článek vyšel v časopiseKlinická onkologie
Nejčtenější tento týden
2018 Číslo 4- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejlepší kůže je zdravá kůže: 3 úrovně ochrany v moderní péči o stomii
- Nejasný stín na plicích – kazuistika
- Metamizol v léčbě různých bolestivých stavů – kazuistiky
-
Všechny články tohoto čísla
- Precizovaná onkologie
- Využití mikroRNA ve slinách pro diagnostiku nádorových onemocnění
- Expresní analýza OIP5-AS1 u nemalobuněčného karcinomu plic
- Kombinovaný bioinformatický a literární přístup k identifikaci dlouhých nekódujících molekul RNA, které modulují signalizaci přes receptor vitaminu D u karcinomu prsu
- Monoklonální gamapatie nejasného významu (MGUS)
- Metastáza do lymfatických uzlín na krku pri neznámom primárnom tumore
- Výskyt a antibiotická rezistence enterobakterií izolovaných z klinického materiálu od pacientů s akutní leukemií
- Malígne melanómy kože vyrastajúce na nohe
- Metastázy karcinomu prsu do baze lební
- Primární branchiogenní karcinom
- Fludarabín v liečbe indolentného lymfómu s chylothoraxom
- Terapeutické účinky specifických inhibitorů CDK4/6 při léčbě pokročilých HR-pozitivních a HER2-negativních pokročilých karcinomů prsu
- Nové techniky IGRT – sledování povrchu těla pacienta (SIGRT)
- Klinická onkologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Monoklonální gamapatie nejasného významu (MGUS)
- Metastáza do lymfatických uzlín na krku pri neznámom primárnom tumore
- Výskyt a antibiotická rezistence enterobakterií izolovaných z klinického materiálu od pacientů s akutní leukemií
- Využití mikroRNA ve slinách pro diagnostiku nádorových onemocnění
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Autoři: MDDr. Eleonóra Ivančová, PhD., MHA
Autoři: prof. MUDr. Eva Kubala Havrdová, DrSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



