-
Články
- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Aplikace metody SNP array v prenatální diagnostice
Application of SNP array method in prenatal diagnosis
Objectives:
SNP array (array method using Single Nucleotide Polymorphisms) enables to detect cytogenetically undetectable submicroscopic alterations (microdeletions, microduplications), which could be also causative for ultrasonographic anomalies of fetus. This article describes the principle, advantages, disadvantages and application possibilities of the SNP array method in prenatal diagnosis. The ten month experience with SNP array use in prenatal diagnosis is presented.Design:
Prospective study.Settings:
Gennet, Prague.Material and methods:
During the period from April 2010 to January 2011 we performed 110 SNP array analyses of fetal DNA: 14 chorionic villi samples (CVS), 88 amniotic fluid samples (AMC), 1 cord blood sample and 7 miscarriage samples. Laboratory tests were carried out on DNA from both cultured and uncultured fetal cells. Examinations were performed in fetuses with sonographic abnormal findings having normal karyotype. In addition 14 fetal cytogenetic abnormalities were solved. SNP array analysis was performed using Illumina InfiniumHD HumanCytoSNP-12 chip. All data were analysed by Illumina KaryoStudio and GenomeStudio software.Results:
SNP array analysis was performed in 108 fetuses (only 2 examination failures, 1.8%). In total, we detected CNV (copy number variation) in 29 samples (29/108 = 27%). 15% (16/108) of fetuses with abnormal ultrasound findings were found to carry clinically relevant CNV. Probably benign CNVs were found in 8 samples (8/108 = 7%) and in additional 5 CNVs parental samples have not been analysed yet. Excluding karyotypically abnormal cases clinically relevant CNVs were found in 10% of fetuses (9/94). In all cases with de novo chromosomal aberration the clinical relevancy was clarified (imbalances in 50%).Conclusion:
Our data suggest that SNP array analysis is a relevant and useful technique in prenatal diagnosis.Key words:
SNP array, CNV, prenatal diagnosis, ultrasound diagnosis.
Autoři: V. Bečvářová 1; M. Hynek 1; M. Putzová 1; I. Soldátová 1; J. Horáček 1; D. Smetanová 1; E. Kulovaný 1; M. Matoušková 1; V. Krutílková 1; E. Hlavová 1; D. Rašková 1; M. Hejtmánková 1; K. Čutka 2; D. Čutka 2; D. Stejskal 1; R. Mihalová 3; M. Trková 1
Působiště autorů: Gennet, Centrum lékařské genetiky a reprodukční medicíny, Praha, vedoucí lékař MUDr. D. Stejskal 1; Centrum lékařské genetiky, České Budějovice, vedoucí lékař MUDr. K. Čutka 2; Ústav biologie a lékařské genetiky 1. LF UK a VFN, Praha, přednostka doc. MUDr. M. Kohoutová, CSc. 3
Vyšlo v časopise: Ceska Gynekol 2011; 76(4): 261-267
Souhrn
Cíl studie:
Metoda SNP array (využívající k diagnostice polymorfismy v 1 nukleotidu – single nucleotide polymorphism) umožňuje odhalit v karyotypu nedetekovatelné submikroskopické změny (mikrodelece, mikroduplikace), jež mohou mít kauzální souvislost s patologickým ultrazvukovým nálezem u plodu. V článku je stručně popsán princip, výhody, nevýhody a možnosti použití metody SNP array v prenatální diagnostice. Jsou prezentovány zkušenosti za 10 měsíců používání této metody v prenatální diagnostice.Typ studie:
Prospektivní studie.Pracoviště:
Gennet, Praha.Metodika:
Vyšetření SNP array bylo v období duben 2010 až leden 2011 provedeno u 110 vzorků DNA získané z kultivovaných nebo nekultivovaných buněk plodů po biopsii choria (CVS, n=14), amniocentéze (AMC, n=88), kordocentéze (n=1) a z tkáně z abortů (n=7). Vyšetření byla indikována na základě patologického nálezu na ultrazvuku s normálním karyotypem plodu, případně k dořešení patologického cytogenetického nálezu vzniklého de novo. Zpracování DNA pro chip Illumina InfiniumHD HumanCytoSNP-12v2.1, jeho příprava a skenování byly provedeny podle standardních protokolů firmy Illumina. Data byla analyzována pomocí softwaru Illumina KaryoStudio a GenomeStudio.Výsledky:
Metodou SNP array bylo úspěšně vyhodnoceno 108 vzorků (neúspěch vyšetření byl pouze ve 2 případech, 1,8 %). Odchylky od normálního profilu (CNV-Copy Number Variation) byly zachyceny u 29 případů (29/108 = 27 %), z nichž 16 bylo klinicky významných (16/108=15 %). Jako pravděpodobně nepatogenní byly CNV u 8 případů a u 5 případů nebyl zatím původ nalezené CNV ověřen u rodičů. Po odečtení případů ověřujících de novo patologii v karyotypu plodu byla klinicky významná CNV nalezena v 9 případech (9/94 = 10 %). Nález klinicky významné CNV byl nejčastěji v kategorii ultrazvukových nálezů srdečních vad, vad centrálního nervového systému (CNS) a mnohočetných anomálií. Ve všech 14 případech de novo chromozomální přestavby v karyotypu byla jasně specifikována závažnost přestavby (patologie u 7/14 = 50 %).Závěr:
Výsledky studie ukazují, že vedle jednoznačného přínosu při posuzování patogenity de novo chromozomálních aberací prokazuje SNP array i jednoznačný přínos pro záchyt dosud skrytých submikroskopických změn.Klíčová slova:
SNP array, CNV, prenatální diagnostika, ultrazvuková diagnostika.ÚVOD
Současná prenatální péče představuje komplexní spolupráci ultrazvukové, biochemické a genetické diagnostiky. V indikovaných případech jsou za účelem diagnostiky vrozených vad a chorob plodu prováděna vyšetření metodami cytogenetickými (konvenční karyotyp), molekulárně genetickými (QF-PCR – kvantitativní fluorescenční PCR pro rychlé potvrzení či vyloučení nejčastějších trizomií, i přímá a nepřímá diagnostika předem známých chorob) a molekulárně cytogenetickými (FISH – fluorescenční in situ hybridizace). Všechny používané metody mají své přednosti a současně omezení daná principem každé metody. Na jedné straně je to rychlost provedení vyšetření, na druhé straně nutná cílenost a omezený rozsah zjistitelných odchylek [9]. Proto dochází často ke kombinaci více typů vyšetření. Přesto zůstává určitá část gravidit s patologickým ultrazvukovým (UZ) nálezem (často s infaustní prognózou) a s normálním výsledkem ve všech uvedených vyšetřeních.
Možnou alternativou pro zjištění genetické příčiny a stanovení diagnózy u výše popisovaných případů je metoda SNP array. Je to jedna z technologií celogenomového screeningu (podobně jako metoda array CGH – komparativní genomová hybridizace), která při 1000krát vyšší rozlišovací schopnosti než klasický karyotyp detekuje nebalancované chromozomální abnormity. Vyšetřovacím principem metody SNP array je využití statisticky vybraných konkrétních polymorfismů v 1 nukleotidu mezi 2 alelami (formami téhož genového lokusu) vyskytujících se v genomu každého jedince (single nucleotid polymorphism – SNP).
Metodou SNP array můžeme zachytit chromozomální aberace pod rozlišovací schopností G-pruhovaných chromozomů v konvenčním karyotypu (10 kb versus 5–10 Mb) a současně doplnit standardní cytogenetickou metodu v případě nálezu některých typů chromozomálních aberací (např. určení původu marker chromozomů, derivovaných chromozomů apod.). Výhody a nevýhody jednotlivých cytogenetických metod jsou shrnuty v tabulce 1.
Tab. 1. Srovnání diagnostických možností metod užívaných v prenatální diagnostice 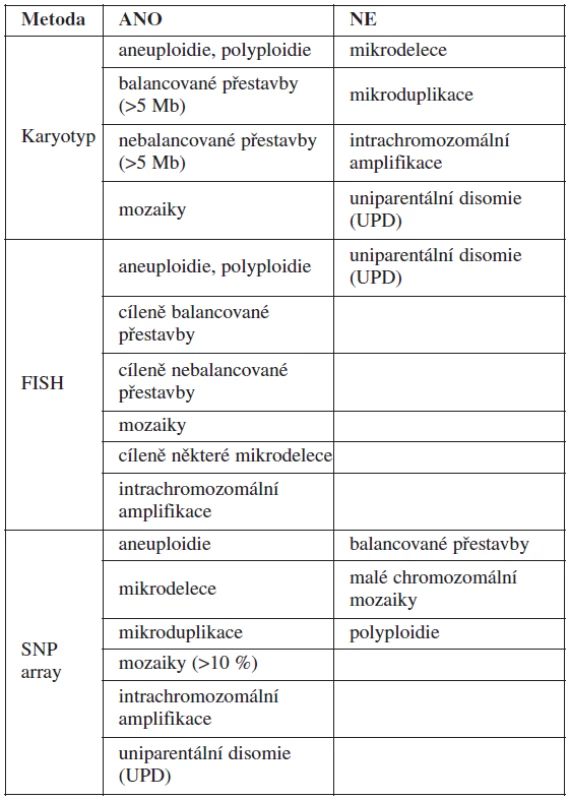
V našich vyšetřeních byla metoda SNP array použita u plodů se závažným UZ nálezem a současně normálním výsledkem konvenčního karyotypu jako metoda doplňková. Dále byla array technologie použita také pro jednoznačné posouzení závažnosti abnormalit nalezených v karyotypu (marker chromozomy, de novo „balancované“ chromozomální aberace apod.).
SOUBOR A METODIKA
Do souboru vyšetřovaných pacientek (plodů) bylo v období duben 2010 až leden 2011 zařazeno 110 případů, u kterých bylo primárně indikováno cytogenetické vyšetření plodu na základě abnormálního nálezu při ultrazvukovém vyšetření: 14 vyšetření choriové biopsie v 1. trimestru těhotenství (CVS), 88 vyšetření plodové vody ve druhém trimestru těhotenství (AMC) a 1 kordocentéza. Bylo provedeno i vyšetření u 7 abortů. Přehled typů vyšetřovaných vzorků je uveden v tabulce 2. Před provedením SNP array vyšetření byly známy výsledky QF-PCR a karyotypu plodu. Dovyšetření a jasné určení chromozomální abnormity nalezené v karyotypu bylo provedeno u 14 vzorků. Všechny pacientky i jejich partneři byli při klinickogenetické konzultaci informováni o provedení a možnostech SNP array vyšetření plodu. Souhlas s ním potvrdili podpisem informovaného souhlasu k vyšetření a dobrovolným odevzdáním vzorku periferní krve v případě nutnosti srovnání SNP array profilů plodu a rodičů. Vzorky plodů s cytogeneticky nebo molekulárně geneticky potvrzenou jasnou patologií (např. trizomie, triploidie apod.) nebyly metodou SNP array vyšetřovány.
DNA byla izolována z buněk plodů po biopsii choria, amniocentéze, kordocentéze a z tkáně potracených plodů (tab. 2). Přibližně u 75 % případů byly před izolací DNA fetální buňky kultivovány, v průměru 15 dní. Pro izolaci DNA byl použit kit QIAamp DNA Mini Kit Qiagen (viz QIAamp DNA Mini Kit Handbook 2003), umožňující izolaci kvalitní DNA ze všech typů výše uvedených vzorků. Zpracování, aplikace na vyšetřovací chip, hybridizace a barvení bylo provedeno podle standardního protokolu firmy Illumina pro chip typu InfiniumHD HumanCytoSNP-12. Skenování chipu bylo provedeno na Illumina Bead Array Reader (servisní laboratoř Ústavu molekulární genetiky AV ČR). K vyhodnocení dat byly použity programy Illumina KaryoStudio1.3 a Illumina GenomeStudioV2010.2. Oba programy pracují se dvěma základními statistickými veličinami. Tyto veličiny jsou v případě Illumina SNP array virtuálně srovnávány softwarem se statisticky vybraným reprezentativním souborem 200 kontrolních vzorků zdravých jedinců. Z toho vyplývá, že vlastní laboratorní příprava každého chipu musí být provedena za striktního dodržení podmínek, za kterých byly tyto referenční vzorky zpracovávány.
Tab. 2. Přehled prenatálních vyšetření z hlediska původu vyšetřovaného materiálu 
První vyšetřovanou veličinou je „normalizovaná hodnota intenzity fluorescence“ – pojem log R ratio. Ta charakterizuje, stejně jako u běžné CGH, zvýšení (např. duplikace) nebo snížení (delece) počtu kopií (CNV – copy number variation) daného úseku v konkrétním vzorku vyšetřované DNA. Oproti běžné CGH odpadá díky počítačové analýze nutnost přípravy standardní referenční DNA. Druhou užívanou veličinou je „frekvence alely B“ – pojem B allele frequency. Díky výpočtu této hodnoty je možné zjistit, jaký je genotyp vzorku v místě daného vyšetřeného polymorfismu a lze detekovat potenciálně patogenní místa neutrální z hlediska počtu kopií (ztrátu heterozygozity, uniparentální dizomii). To běžná array-CGH neumožňuje.
V případě nálezu odlišností v SNP array profilu od standardní statistické normy (Illumina KaryoStudio a GenomeStudio) byla zjištěná data porovnávána s veřejně dostupnými genomickými databázemi pro populační varianty CNV a pro popsané patologie [14, 15, 16]. Za účelem potvrzení původu CNV byl takový profil plodu porovnáván s profily obou rodičů, aby se vyloučila možnost familiárního polymorfismu. Hodnocení se řídí mezinárodními doporučeními Cytogenetic European Quality Assessment (CEQA) a nomenklaturou International System of Cytogenetic Nomenclature (ISCN).
Časové schéma metody zahrnovalo 4 dny laboratorní práce, několik hodin skenování a dobu nutnou pro samotné vyhodnocení profilu (liší se podle UZ nálezu a zjištěného CNV – 1 až 3 dny). U kultivovaných buněk je časové schéma prodlouženo o dobu kultivace. Tj. u vzorků bez kultivace od dodání vzorku k odevzdání výsledku dohromady přibližně 7 dní, při kultivaci fetálních buněk před vlastním vyšetřením je to 15 až 20 dnů.
Výstupem z laboratoře je protokol o molekulárně cytogenetickém vyšetření předaný klinickému genetikovi nebo gynekologovi s genetickou specializací. Protokol obsahuje popis nálezu a komentář. V případě patologického nálezu jsou přiloženy články, abstrakta, případně odkazy na další zdroje informací týkající se daného nálezu.
VÝSLEDKY
Z celkového počtu 110 vzorků bylo vyšetření neúspěšné ve dvou případech, tj. 1,8 %, kdy se nepodařilo zajistit dostatečné množství kvalitní DNA (plodová voda s nízkou koncentrací amniocytů, kontaminace mateřskou krví) a výsledný array profil byl nehodnotitelný. Většina získaného biologického materiálu obou vzorků byla přednostně zpracována pro vyšetření konvenčního ka-ryotypu.
Přehled výsledků je uveden v tabulce 3. Z úspěšně vyšetřených 108 vzorků jsme našli odlišnost od standardního SNP array profilu (CNV) ve 29 případech (29/108 = 27 %). Porovnáním s veřejně dostupnými databázemi CNV u zdravých kontrol, případně vyšetřením rodičů plodu, bylo 16 z nich (16/108 = 15 %) potvrzeno jako patogenní (související s patologickým UZ nálezem). Dalších 8 nalezených CNV (8/108 = 7 %) bylo vyhodnoceno jako pravděpodobně nepatogenní a dalších 5 CNV nebylo dosud ověřeno (5/108 = 5 %). Pokud odečteme 14 případů, kde byla řešena závažnost abnormálního karyotypu plodu, z celkového počtu 94 vyšetřených plodů byl záchyt klinicky významných CNV v 9 případech (10 %). V jednom případě (tab. 4 – pacientka 8) bylo z důvodu vysokého týdne těhotenství (23. týden) provedeno array vyšetření z nekultivovaných buněk dříve, než byl znám výsledek karyotypu. Byla nalezena terminální delece na krátkých raménkách chromozomu 7 velikosti přibližně 3,4 Mb (megabase – 106 bazí). Tento nález byl posléze verifikován v karyotypu plodu, kde byla aberace na hranici mikroskopické rozlišitelnosti.
Tab. 3. Přehled výsledků prenatálních vyšetření metodou SNP array 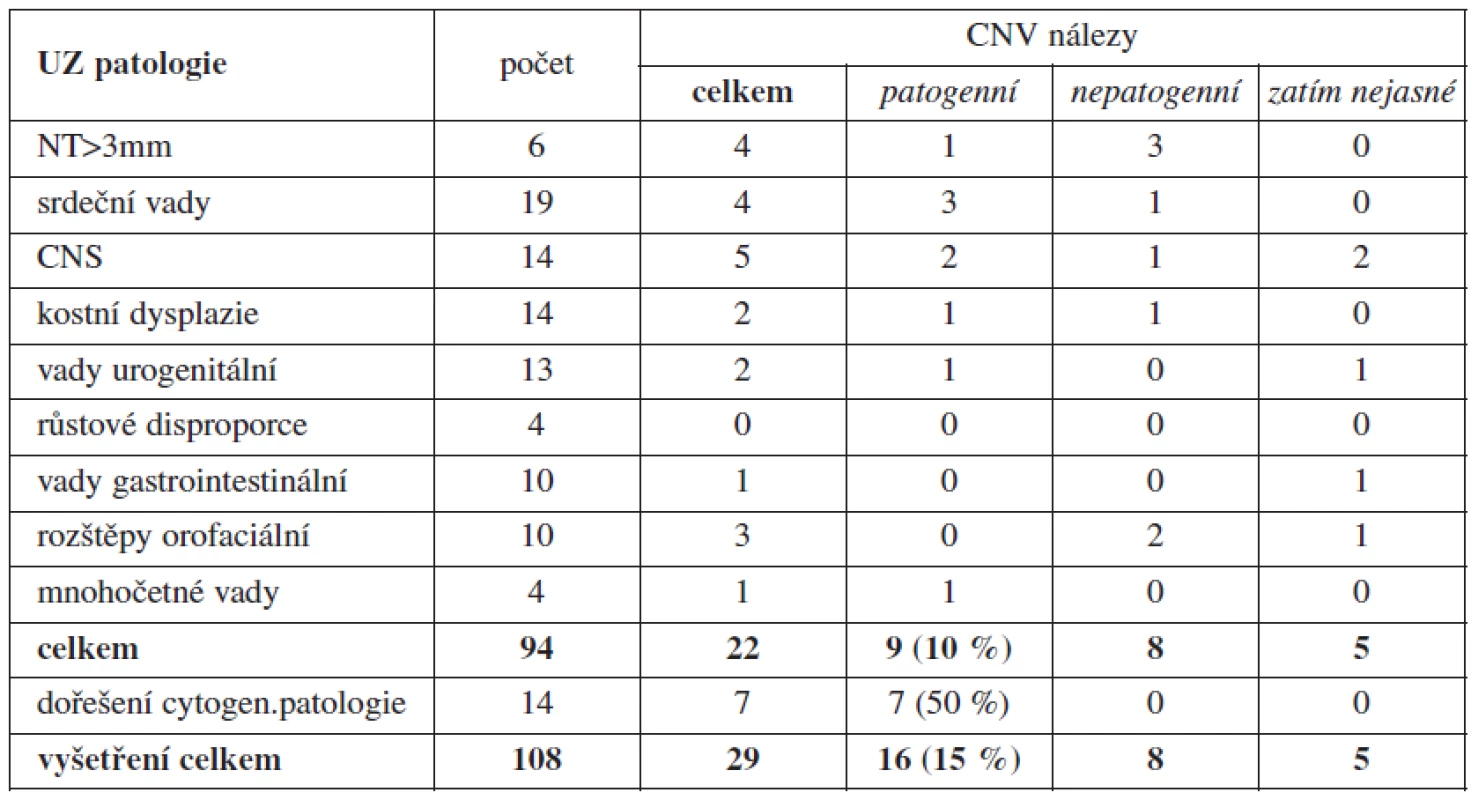
Rozdělení UZ patologií dle Kleeman et al. Use of array CGH for prenatal diagnosis of fetuses with sonographic anomalies [3] Tab. 4. Přehled klinicky významných nálezů u plodů s ultrazvukovými anomáliemi v 1. nebo 2. trimestru 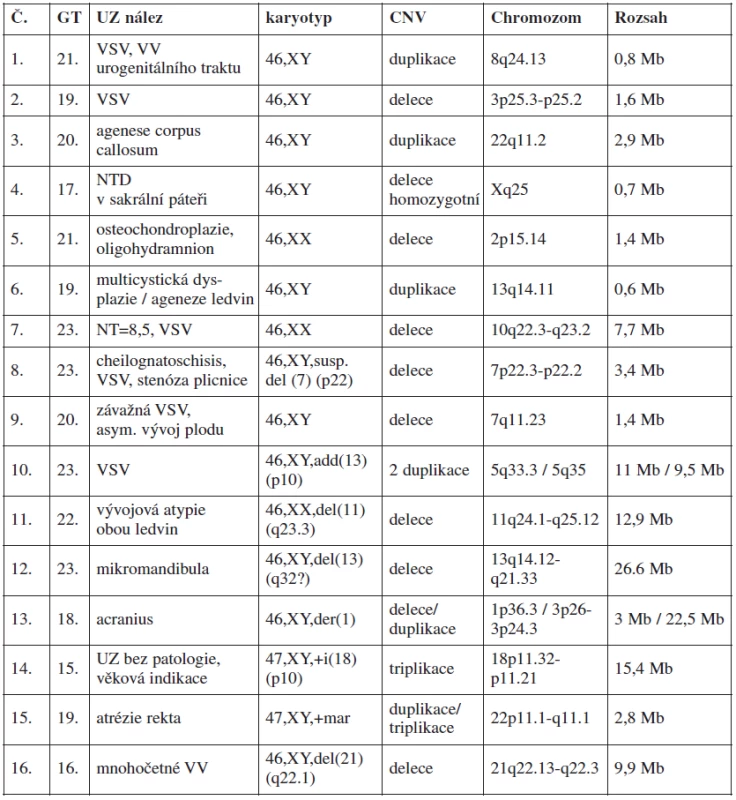
Vysvětlivky: Č - číslo pacienta; GT - gestační týden; CNV - Copy Number Variation; VSV - vrozená srdeční vada; NT - nuchální translucence; Mb - megabase (106 bází) Cytogenetické zápisy podle aktuální verze International System of Cytogenetic Nomenclature (ISCN 2009) p - krátké raménko chromozomu; q - dlouhé raménko chromozomu; číslo před "p" (resp. "q") označuje číslo chromozomu; číselné údaje za písmenem p (resp. q) udávají přesnou polohu aberantního místa na konkrétním chromozomu; mar - marker chromozom; add - na chromozomu přítomen nadbytečný neznámý chromozomální materiál; del - delece; i - isochromozom; der - derivovaný chromozom (změna na chromozomu dále cytogeneticky neidentifikovatelná) V jednotlivých UZ indikacích byl nejvyšší záchyt klinicky relevantní patogenní CNV u anomálií centrálního nervového systému (2/14 a 2 další zatím nedořešeny) a srdečních vad (3/19). Nejvíce dalších nálezů klinicky významných CNV bylo u indikace s patologií nuchální translucence (1/6), mnohočetných ultrazvukových vad (1/4), urogenitálních anomálií (1/13, 1 zatím nedořešena) a kostních dysplazií (1/14). Nedořešené případy se týkají těhotenství, která byla ukončena a rodiče zatím neposkytli vzorek periferní krve ke srovnání jejich SNP array profilů s nálezem u plodu. U prenatálně zjištěných vad typu růstové retardace, vad gastrointestinálního traktu a orofaciálních rozštěpů (které nejsou asociovány s jiným UZ nálezem) nebyl dosud žádný záchyt patologické CNV.
Jednoznačné posouzení závažnosti cytogenetického nálezu se podařilo ve všech vyšetřovaných případech; u 50 % (7/14) byla prokázána patogenita (nevyvážená chromozomální výbava). U dalších 7 plodů byly SNP array profily v normě, což umožnilo uzavřít prenatální vyšetření s příznivou prognózou. Např. u 2 našich pacientek byly v karyotypu nalezeny komplexní translokace zahrnující 5 (chromozomy 1, 6, 8, 18 a 21) resp. 3 chromozomy (chromozomy 3, 9 a 4). Obě translokace se cytogeneticky v karyotypu jevily jako balancované. Po provedení vyšetření SNP array byl tento závěr v obou případech potvrzen ve smyslu zachování normálního počtu kopií ve všech vyšetřovaných SNP markerech. První gravidita (translokace s 5 chromozomy) byla v lednu 2011 úspěšně završena porodem fyziologického novorozence, druhá gravidita pokračuje s normálním ultrazvukovým nálezem.
V tabulce 4 je uveden přehled 16 kauzálních aberací s UZ nálezy. Příklady patologických profilů u 2 plodů jsou uvedeny v obr. 1 a 2.
Obr. 1. Výsledek vyšetření metodou SNP array – mikrodelece na chromozomu 7 (případ č. 9, tab. 4). Mikrodelece 7q11.23 (okrový blok) o velikosti 1.4 Mb u plodu se závažnou srdeční vadou a vývojovou asymetrií. Mikrodelece překrývá oblast pro Williamsův-Beurenův syndrom. 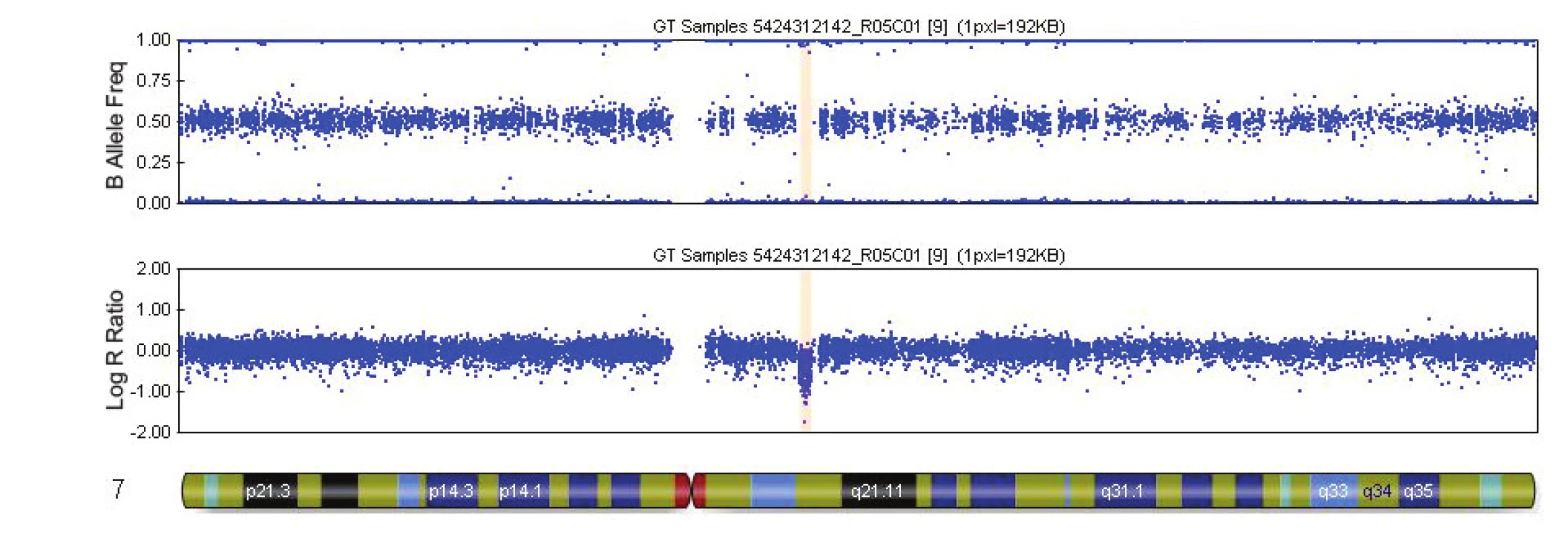
Obr. 2. Výsledek vyšetření metodou SNP array - mikroduplikace na chromozomu 22 (případ č. 3, tab. 4). Duplikace 22q11.21 (modrý blok) o velikosti 2.9 Mb u plodu s agenezí corpus callosum. Duplikace překrývá oblast pro tzv.22q11.21 mikroduplikační syndrom. Zdroj: Illumina GenomeStudioV2010.2 / Chromosome Browser 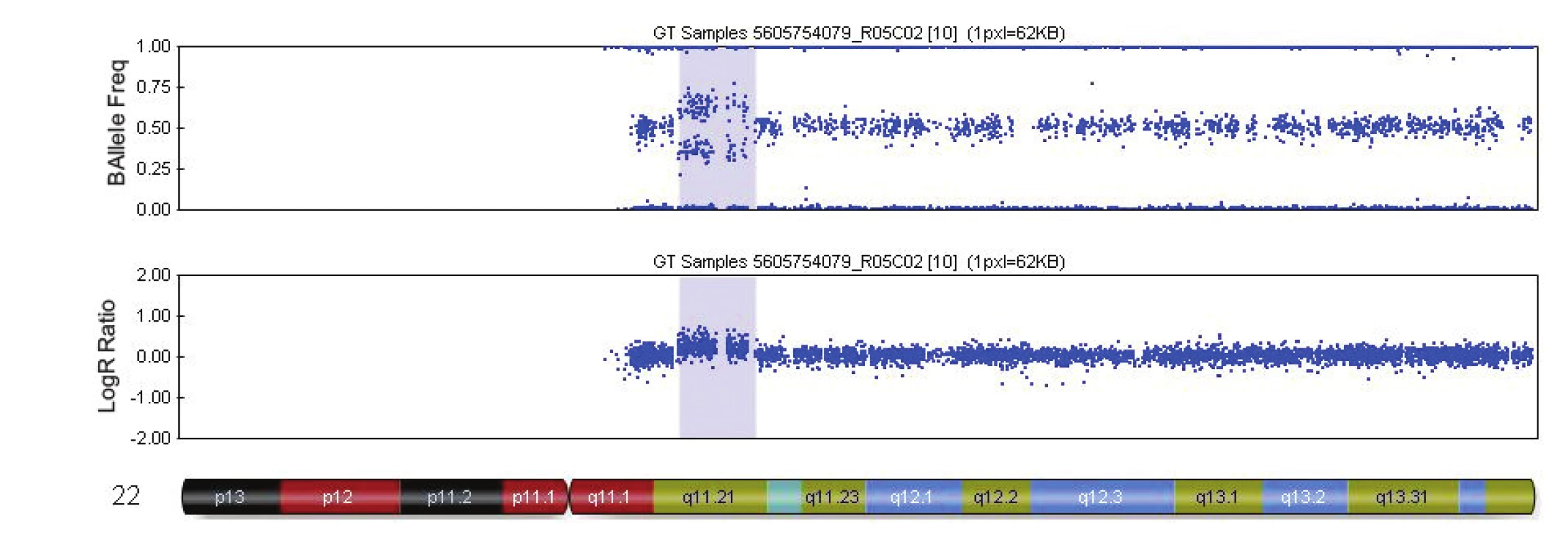
DISKUSE
Lidská populace je z hlediska CNV (mikrodelecí a mikroduplikací) velmi variabilní a v každém vyšetřovaném místě existuje poměrně velká pravděpodobnost variant [10]. Za účelem shromažďování dat o těchto variantách, jejich klinické významnosti, eventuálně spojitosti se známými postiženími a geneticky podmíněnými chorobami vznikly volně dostupné databáze [14, 15, 16], které jsou stále aktualizovány. Zavedení metody SNP array v prenatální diagnostice následovalo v naší laboratoři až po období, kdy byla tato metoda užívána pouze pro postnatální vyšetření. Jednak k ověření cytogeneticky známých aberací a poté pro pacienty s různým stupněm geneticky podmíněného postižení, kde nebyla jinými metodami stanovena příčina postižení. Bez ohledu na původ vzorku vyšetřované DNA se laboratorní provedení i hodnocení řídí v podstatě shodnými pravidly. Vzhledem k tomu, že nepatogenní CNV různého rozsahu se nalézají i u zdravých kontrol jako běžné varianty, byla také u každé zachycené CNV u plodu prověřena závažnost nálezu porovnáním s databází známých variant [15]. Pro posouzení klinické relevance musí být dále prokázán původ CNV vyšetřením rodičů. Uvádí se, že 90-97 % zjištěných odlišností totiž může být familiárních [1, 3, 5]. Teprve až po porovnání nalezené CNV plodu s databází známých a popsaných mikrodelecí / mikroduplikací, ověření případného parentálního původu CNV, vyloučení běžné varianty a po prostudování genů lokalizovaných v duplikované nebo deletované oblasti, byly CNV považovány za klinicky významné. Je-li to metodicky dostupné, doporučuje se nález paralelně vyšetřit některou jinou laboratorní metodou (FISH, MLPA-Multiple Ligation-dependent Probe Amplification) [13].
Jak je patrno z tabulky 3, byl v jednotlivých UZ indikacích nejúčinnější záchyt klinicky relevantní CNV u anomálií CNS (2/14, 2 zatím nedořešeny) a srdečních vad (3/19). Více nálezů klinicky významných CNV bylo u indikace s patologií nuchální translucence (1/6), mnohočetných ultrazvukových vad (1/4), urogenitálních anomálií (1/13, 1 zatím nedořešena) a kostních dysplazií (1/14). Žádný záchyt patologické CNV zatím nebyl u prenatálně zjištěných vad typu růstové retardace, vad gastrointestinálního traktu a orofaciálních rozštěpů (které nejsou asociovány s jiným UZ nálezem). Výsledky jsou srovnatelné s výsledky podobných studií používajících některou z metod array (array CGH nebo SNP array) v prenatální diagnostice: Tyreman a kol. našli patogenní CNV u 10 % plodů s abnormálním UZ nálezem [11]; Faas a kol. našli de novo klinicky relevantní CNV u 16 % plodů s patologickým UZ a normálním karyotypem [2].
Stejně tak i výsledky jednotlivých UZ nálezů odpovídají údajům srovnatelných studií, kde byl nejvyšší záchyt klinicky relevantních CNV u srdečních vad, vad CNS, urogenitálních a skeletálních vad [3, 11]. Ukazuje se, že v případě některých UZ abnormalit (izolované vady gastrointestinálního traktu a izolované orofaciální rozštěpy) array vyšetření zřejmě nepřispívá k objasnění etiologie.
Jednoznačný přínos pro prenatální diagnostiku představuje array technologie při upřesňování rozsahu nebo závažnosti aberací vzniklých de novo. Vermeesh ve své studii udává, že 6–10 % zdánlivě balancovaných translokací je spojeno s následnými kongenitálními anomáliemi, což nepochybně souvisí s mikrodelecemi nebo mikroduplikacemi v oblasti zlomů [12]. Velký přínos představuje metoda SNP array v případech, kdy je v karyotypu plodu nalezen de novo marker chromozom (nadpočetný, dále cytogeneticky nezařaditelný genetický materiál). Je možné rozlišit, zda je v marker chromozomu obsažena kódující DNA, včetně určení popsaných genů, nebo zda jde pravděpodobně o heterochromatinový nekódující materiál, bez fenotypických následků pro plod. Vyšetření SNP array tak poskytuje cenné informace klinickému genetikovi a významně přispívá k rozhodování rodičů o osudu gravidity. Příkladem vyšetření marker chromozomu v našem souboru je případ 15 (viz tab. 4).
Naprosto unikátní je SNP array v identifikaci ztráty heterozygozity, a tím identifikace uniparentální disomie. Je známo, že potomci nosičů některých typů chromozomálních přestaveb, například některých robertsonských translokací, mají zvýšené riziko uniparentální disomie (izodisomie nebo heterodisomie), jejímž důsledkem je projev genetického onemocnění při zdánlivě normálním karyotypu [4, 6]. Vyšetřením plodu a rodičů dítěte touto metodou lze rozlišit uniparentální disomii na jakémkoli chromozomu či jeho části.
Hlavním úskalím použití array technologie v prenatální diagnostice je omezená možnost korelovat nalezenou mikroduplikaci nebo mikrodeleci s fenotypem. Na rozdíl od postnatálního vyšetření pacientů s psychomotorickou retardací nebo stigmatizací, kde se staly metody molekulární cytogenetiky vyšetřením první volby [1, 7], je array v prenatální diagnostice zatím metodou doplňkovou. Přibývající studie v tomto oboru přinesou dosud chybějící poznatky pro stanovení souvislostí mezi patologickými ultrazvukovými nálezy u plodu a jejich molekulárním základem [3, 11, 12].
Vyšetření SNP array je nutné plánovat i s ohledem na gestační týden. Z kapacitních a ekonomických důvodů není možné provádět takové vyšetření denně. Jako optimální a časově dostačující se ukazuje počkat na zaplnění alespoň dvou chipů (24 vzorků). Stávající perioda v naší laboratoři je 2–3 týdny. Pro maximální zkrácení doby vyšetření se podařilo vypracovat protokol izolace kvalitní DNA (minimální koncentrace 50 ng/μl) přímo z nekultivovaných buněk. Díky tomu se doba trvání vyšetření zkrátila o dobu kultivace. Výsledky SNP array profilů při použití DNA z kultivovaných a nekultivovaných buněk jsou zcela srovnatelné.
ZÁVĚR
Celogenomový screening submikroskopických změn metodou SNP array má v prenatální diagnostice význam díky svému vysokému rozlišení. Jejím použitím dochází k záchytu mnoha klinicky významných mikrodelecí a mikroduplikací, jež by nebyly jinými genetickými metodami odhaleny. Metoda SNP array tak může objasnit příčinu závažných ultrazvukových nálezů a dát relevantní informaci pro genetické poradenství u případné další gravidity. Současně může pomoci při rozhodování o osudu gravidity tam, kde při abnormálním nálezu na ultrazvuku všechna ostatní vyšetření vykazují normální výsledky. Nepopíratelný význam má v případech identifikace de novo aberace nejasného významu v karyotypu plodu. V této souvislosti se již objevily i odkazy na význam stanovení abnormálních CNV ve vzorcích plodů pocházejících z opakovaných potratů, k nimž dochází u některých párů bez zjevné příčiny [8].
Poděkování
Chceme poděkovat kolektivu Laboratoře molekulární genetiky Gennet za vstřícnost a spolupráci při izolaci DNA včetně vylepšení protokolu izolace z nekultivovaných buněk. Dále děkujeme všem pracovníkům Cytogenetické laboratoře Gennet za vedení kultivací a za přípravu buněk pro izolaci. Děkujeme za ochotnou spolupráci kolektivu Oddělení fetální medicíny a Oddělení lékařské genetiky. V neposlední řadě také pracovníkům IT oddělení Gennet.
Naše poděkování patří i všem spolupracujícím gynekologům a klinickým genetikům, kteří nechápou abnormální těhotenský nález izolovaně, ale v širších medicínských souvislostech. Tím zprostředkovaně přispívají k rozšíření poznatků o souvislostech abnormálních těhotenských nálezů s jejich možnou genetickou příčinou.
Ing. Věra Bečvářová
Gennet
Centrum lékařské genetiky a reprodukční medicíny
Kostelní 292/9
170 00 Praha 7
Korespondence
Ing Věra Bečvářová
e-mail: vera.becvarova@gennet.cz
RNDr. Marie Trková, Ph.D.
e-mail: marie.trkova@gennet.cz
Zdroje
1. Ahn, J., Mann, K., Walsh, S., et al. Validation and implementation of array comparative genomic hybridisation as a first line test in place of postnatal karyotyping for genome imbalance. Molecular Cytogen, 2010, 3, p. 9.
2. Faas, BHW., de Burgt, I., Kooper, AJA., et al. Identification of clinically significant submicroscopic chromosome alterations and UPD in fetuses with ultrasound anomalies using genome-wide 250k SNP array analysis. J Med Genet, 2010, 47, p. 586.
3. Kleeman, L., Bianchi, DW., Shaffer, LG., et al. Use of array comparative genomic hybridization for prenatal diagnosis of fetuses with sonographic anomalies and normal metaphase karyotype. Prenatal Diagnosis, 2009, 29, p. 1213.
4. Kotzot, D. Complex and segmental uniparental disomy updated. J Med Genet, 2008, 45, p. 545.
5. LaFramboise, T. Single nucleotide polymorphism arrays: a decade of biological, computational and technological advances. Nucleic Acids Res, 2009, 37, p. 4181.
6. Liehr, T. Cytogenetic contribution to uniparental disomy (UPD). Molecular Cytogen, 2010, 3, p. 8.
7. Miller, DT., Adam, MP., Aradhya, S., et al. Consensus Statement: Chromosomal microarray is a first-tier clinical diagnostic test for individuals with developmental disabilities or congenital anomalies. Am J Hum Genet, 2010, 86, p. 749.
8. Rajcan-Separović, E., Diego-Alvarez, D., Robinson, WP., et al. Identification of copy number variants in miscarriages from couples with idiopathic recurrent pregnancy loss. Human Reprod, 2010, 25, p. 2913.
9. Shaffer, LG., Bui, TH. Molecular cytogenetic and rapid aneuploidy detection methods in prenatal diagnosis. Am J Med Genet Part C, 2007, 145C, p. 87.
10. Shaikh, TH., Gai, X., Perin, JC., et al. High-resolution mapping and analysis of copy number variations in the human genome: A data resource for clinical and research applications. Genome Res, 2009, 19, p. 1682.
11. Tyreman, M., Abbott, KM., Willatt, LR., et al. High resolution array analysis: diagnosing pregnancies with abnormal ultrasound findings. J Med Genet, 2009, 46, p. 531.
12. Vermeesch, JR., Fiegler, H., de Leeuw, N., et al. Guidelines for molecular karyotyping in constitutional genetic diagnosis. Eur J Hum Genet, 2007, 15, p. 1105.
13. Vissers, LELM., de Vries, BBA., Veltman, JA. Genomic microarrays in mental retardation: from copy number variation to gene, from research to diagnosis. J Med Genet, 2010, 49, p. 289.
Web stránky(veřejně dostupné databáze):
14. Database of Chromosomal Imbalance and Phenotype in Humans using Ensembl Resources(DECIPHER), http://decipher.sanger. ac.uk/application/, verze v 5.1, GR Ch37
15. Database of Genomic Variants(DGV), http://projects.tcag.ca/ variation/, verze Human genome assembly hg.19, Human (GR Ch37).
16. UCSC Genome Bioinformatics Site, http://genome.ucsc.edu, verze Feb. 2009 (GR Ch37/hg19).
Štítky
Dětská gynekologie Gynekologie a porodnictví Reprodukční medicína
Článek Abstrakt
Článek vyšel v časopiseČeská gynekologie
Nejčtenější tento týden
2011 Číslo 4- Alergie na antibiotika u žen s infekcemi močových cest − poznatky z průřezové studie z USA
- Horní limit denní dávky vitaminu D: Jaké množství je ještě bezpečné?
- Magnosolv a jeho využití v neurologii
- Moje zkušenosti s Magnosolvem podávaným pacientům jako profylaxe migrény a u pacientů s diagnostikovanou spazmofilní tetanií i při normomagnezémii - MUDr. Dana Pecharová, neurolog
- Isoprinosin je bezpečný a účinný v léčbě pacientů s akutní respirační virovou infekcí
-
Všechny články tohoto čísla
- Doporučené postupy – závazné, nebo nezávazné?
- Transabdominální ultrazvukové vyšetření v gynekologii
- Minimálně invazivní výkony pod ultrazvukovou kontrolou v onkogynekologii
- Aplikace metody SNP array v prenatální diagnostice
- Vplyv exogénneho luteinizačného hormónu na parametre IVF/ICSI cyklov
- Metylácia vybraných tumor-supresorických génov v benígnych a malígnych ovariálnych nádoroch
- Současná klasifikace zhoubných nádorů v onkogynekologii – část I
- Biochemické aspekty fetálnej hypoxie
- Nová metoda screeningu Downova syndromu v I. trimestru: One-Stop-Clinic for Assessment of Risk (OSCAR)
- Trikuspidální regurgitace u plodu
- Je třeba revidovat doporučení prevence trombembolické nemoci u těhotných?
- Riziko vzniku prolapsu „de novo“ v primárne nepostihnutom kompartmente pri použití syntetického implantátu v rámci operačnej liečby prolapsu panvových orgánov
- Profesor MUDr. Vojtěch Šnaid, DrSc.Významná osobnost české gynekologie a porodnictví21. 9. 1911 – 25. 6. 2001
- Abstrakt
- Postřehy z ENTOG exchange Manchester, Velká Británie
- Entog exchange ve Velké Británii 2. 5.–7. 5. 2011
- Česká gynekologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Trikuspidální regurgitace u plodu
- Aplikace metody SNP array v prenatální diagnostice
- Transabdominální ultrazvukové vyšetření v gynekologii
- Současná klasifikace zhoubných nádorů v onkogynekologii – část I
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Autoři: MDDr. Eleonóra Ivančová, PhD., MHA
Autoři: prof. MUDr. Eva Kubala Havrdová, DrSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



