-
Články
- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Chromotripse – rozsáhlé chromozomové přestavby a jejich význam u onkologických onemocnění
Chromothripsis – Extensive Chromosomal Rearrangements and Their Significance in Cancer
Background:
Chromosome rearrangements play an important role in cancer pathophysiology. Recently, chromothripsis has been proposed among the mechanisms leading to their formation. Chromothripsis leads to fragmentation of chromosomes and their reconstitution with tens to hundreds of rearrangements clustered in small genome regions. In contrast to the traditional concept of malignant transformation, abnormalities caused by chromothripsis are not accumulated gradually but arise during a single event. The resulting structural variants are extensive and often cause oncogene activation or tumor suppressor inactivation. Chromothripsis is associated with many tumor types, especially with brain and bone tumors. Besides that, it has been described also in congenital disorders. The exact mechanism of chromothripsis origin has not been clarified yet; however, several hypotheses have been prosed, among which DNA damage in micronucleus seems to be most likely. Similarly, an impact of chromothripsis on cellular processes has not been fully understood, yet its association with unfavorable prognosis has been observed.
Purpose:
The purpose of this article is to summarize the current knowledge about chromothripsis and to present gathered pieces of information in a structured way. We focused on describing the basic features of chromothripsis, potential mechanisms of its origin, its impact on cellular processes and providing an overview of diseases where chromothripsis has been noted, with particular attention to cancer. Finally, we suggest a potential use of current knowledge about chromothripsis in the optimization of personalized treatment.
Supported by Ministry of Health of the Czech Republic, grant no. 15-31834A. All rights reserved.
The authors declare they have no potential conflicts of interest concerning drugs, products, or services used in the study.
The Editorial Board declares that the manuscript met the ICMJE recommendation for biomedical papers.
Submitted: 31. 12. 2018
Accepted: 19. 3. 2019
Keywords:
chromothripsis – Chromosomes – DNA – Mutation – genomic structural variation – DNA end-joining repair
Autoři: K. Závacká 1; K. Plevová 1,2; M. Jarošová 1; Š. Pospíšilová 1,2
Působiště autorů: Centrum molekulární bio logie a genové terapie, Interní hematologická a onkologická klinika LF MU a FN Brno 1; Centrum molekulární medicíny, CEITEC – Středoevropský technologický institut, Masarykova univerzita, Brno 2
Vyšlo v časopise: Klin Onkol 2019; 32(2): 101-108
Kategorie: Přehled
doi: https://doi.org/10.14735/amko2019101Souhrn
Východiska:
Chromozomové přestavby hrají významnou roli v patofyziologii nádorů. Mezi mechanizmy vedoucí k jejich vzniku byla v nedávné době zařazena chromotripse. Ta způsobuje rozpad chromozomů a jejich opětovné seskupení s desítkami až stovkami přestaveb. Na rozdíl od klasického schématu nádorové transformace se při chromotripsi aberace nehromadí postupně, ale vznikají během jediné události. Vzniklé přestavby bývají rozsáhlé a často zapříčiňují aktivaci onkogenů či inaktivaci nádorových supresorů. Chromotripse je spojována s mnoha typy nádorů, především s nádory mozku a kostí. Byla však popsána nejen u nádorových onemocnění, ale i u vrozených poruch. Přesný mechanizmus vzniku nebyl dosud objasněn, existuje však několik modelů, z nichž nejpravděpodobnější se jeví vznik přestaveb následkem poškození DNA v tzv. mikrojádře. Stejně tak důsledky chromotripse pro průběh buněčných pochodů nejsou zatím plně pochopeny. U onkologických pacientů obecně je pozorována souvislost mezi chromotripsí a nepříznivou prognózou.
Cíl:
Záměrem tohoto článku je shrnout dosavadní poznatky o chromotripsi a předložit je čtenáři v přehledné podobě. Zaměřili jsme se na popsání základních rysů chromotripse, pravděpodobných mechanizmů jejího vzniku, důsledků na buněčné procesy a dále na poskytnutí přehledu onemocnění, u kterých se s ní můžeme setkat, přičemž pozornost je věnována zejména onkologickým onemocněním. Na závěr je uveden výhled pro využití dosavadních poznatků o chromotripsi při optimalizaci léčebných postupů u onkologických pacientů.
Klíčová slova:
chromotripse – chromozomy – DNA – mutace – strukturní varianta genomu – oprava poškození DNA
Úvod
Nádorová transformace je podmíněna vznikem aberací a hromaděním mutací v lidském genomu, k nimž může docházet při každém buněčném cyklu. Zajímavým objevem v oblasti nádorové biologie je nedávno popsaná chromotripse – proces zahrnující vznik komplexních genomových přestaveb velkého rozsahu. Chromotripse byla v různé míře pozorována u mnoha typů nádorů [1,2], ale také u některých vrozených poruch [3] a několik případů bylo zaznamenáno i u zdravých jedinců [4].
Pojem chromotripse je odvozen z řeckých výrazů „chromos“ pro chromozom a „thripsis“ pro rozpad na kousky. Během chromotripse totiž dochází k rozpadu chromozomů nebo jejich částí na malé fragmenty. Ty jsou následně prostřednictvím nehomologního spojování konců DNA (non-homologous end joining – NHEJ) či procesů založených na mikrohomologii [1,5] sestaveny zpět, většinou ovšem nepřesně oproti původní struktuře (schéma 1). Vznikají tak četné delece, inverze, inzerce, amplifikace a translokace. Mnohé experimenty naznačují, že ke vzniku těchto přestaveb dochází během jediného buněčného dělení [1].
Schéma 1. Schéma vzniku chromotripse. 
Vedle chromotripse byly popsány také příbuzné mechanizmy, jako jsou chromoanasyntéza a chromoplexe (souhrnně bývají všechny tři procesy označovány jako chromoanageneze), které se od chromotripse liší strukturou chromozomových přestaveb [6].
Hlavní rysy chromotripse
Existuje několik základních charakteristik chromotripse, které lze pozorovat pomocí cytogenetických a genomických metod, k nimž patří např. mnohobarevná fluorescenční in situ hybridizace, čipy pro detekci jednonukleotidových polymorfizmů či čipy pro komparativní genomovou hybridizaci. Nejpřesnější charakterizaci však poskytuje celogenomové sekvenování.
Typickým znakem chromotripse je vznik desítek až stovek chromozomových přestaveb mezi několika málo chromozomy, ale i v rámci jednoho chromozomu, jeho ramene či pouze konkrétního regionu. Obvykle se přestavby vyskytují ve shlucích, tzv. klastrech. Dlouhé úseky neporušené DNA mohou střídat úseky cca 50 kb zahrnující 5–10 zlomů [1].
Vznik nebalancovaných aberací lze pozorovat pomocí čipových technologií jako změnu počtu kopií DNA. Počet kopií je alternativním označením pro počet alel, standardně je charakterizován počtem dva. Dojde-li ovšem k nebalancované aberaci, hodnota se změní. V případě delece dochází ke ztrátě heterozygotnosti (ztrátě jedné alely), a je tedy přítomna pouze jedna kopie DNA. Při duplikaci se naopak počet kopií zvýší na tři. V genomu zasaženém chromotripsí počet kopií DNA obvykle kolísá mezi jednou až třemi [1]. Tento znak nejvíce podporuje myšlenku, že k přestavbám provázejícím chromotripsi dochází naráz během jednoho buněčného dělení. Pokud by k přestavbám docházelo postupně, změny by byly různorodější.
Opětovné spojování fragmentů DNA probíhá při chromotripsi náhodně, a proto je náhodná i jejich posloupnost a orientace [1]. Každý volný fragment má dva konce, které se označují jako „head“ (5‘ konec) a „tail“ (3‘ konec). Jejich náhodným spojením dochází ke vzniku čtyř typů spojů – head-to-tail (vznik duplikace), tail-to-head (vznik delece), head-to-head a tail-to-tail (vznik inverze), a to v téměř stejném poměru (schéma 2).
Schéma 2. Nehomologní spojování volných konců DNA. 
dsDNA – dvouvláknová DNA V některých případech při chromotripsi dochází k formování malých kruhovitých útvarů, tzv. double-minute chromozomů [1,5]. Vznikají duplikací části chromozomu a často nesou onkogeny. Replikují se nezávisle na buněčném cyklu a v buňce jsou udržovány, jelikož představují selekční výhodu pro růst a přežití.
Modelové mechanizmy vzniku chromotripse
Dosud nepanuje jednotný názor na to, jakým způsobem k chromotripsi dochází. Bylo popsáno několik modelových mechanizmů.
V současnosti nejvíce přijímaný je založen na pozorování mimojaderných struktur, tzv. mikrojader (schéma 3A). Tyto struktury vznikají během mitózy odpojením chromozomů nebo jejich částí od hlavního jádra při přechodu buňky z metafáze do anafáze. Oddělené chromozomy v mikrojádře se replikují pomaleji než chromozomy v hlavním buněčném jádře. Dosud se předpokládalo, že v mikrojádře nedochází k aktivaci kontrolních bodů buněčného cyklu, odpojené chromozomy předčasně kondenzují a rozpadají se [7]. Jedna z nejnovějších studií však poukazuje na to, že je rozpad chromozomů zapříčiněn defektním jaderným obalem, který je tvořen pouze nejnutnějšími proteiny. Je mnohem křehčí než normální jaderný obal, není schopen tvořit póry, a tudíž neumožňuje transport důležitých proteinů zajišťujících integritu genomu [8]. Fragmenty chromozomů se poté mohou z mikrojádra vyčlenit a vytvořit tzv. double-minute chromozomy (viz předchozí kapitola), nebo se náhodně pospojují a vznikne derivovaný chromozom. Ten může v mikrojádře existovat po několik dalších buněčných cyklů a v mnoha případech se z dceřiných buněk zcela vytrácí. S vysokou frekvencí ale dochází k jeho začlenění zpět do hlavního jádra [9]. Buněčným dělením pak vznikají dceřiné buňky s různě přestavěným genomem.
Schéma 3. Nejvíce přijímané mechanizmy vzniku chromotripse v průběhu buněčného cyklu. A. Formování mikrojádra. 
B. Breakage-fusion-bridge cyklus. Jiná hypotéza předpokládá, že existuje vztah mezi chromotripsí a zkracováním telomer, jelikož charakteristické přestavby často zasahují až do oblasti telomer [1]. V případě abraze telomer buňka spouští tzv. breakage-fusion-bridge cyklus, který se opakuje tak dlouho, dokud konce chromozomů nezískají novou telomeru, obvykle translokací z jiného chromozomu (schéma 3B) [10].
Další hypotézy považují za možné příčiny chromotripse exogenní vlivy poškozující DNA, jako je ionizující záření nebo volné radikály. V jejich důsledku totiž vznikají náhodné zlomy DNA, a tím se aktivují mnohé opravné mechanizmy [11]. Uvažuje se také, že chromotripse může být způsobena replikačním stresem, který vede k zastavení replikační vidlice a vzniku dvouvláknových zlomů DNA [12].
Zajímavý pohled přináší myšlenka, že by některé buňky mohly být schopny unikat apoptóze (programované buněčné smrti) i v případech poškození DNA, např. při nedostatku kyslíku. Místo toho, aby podstoupily apoptózu, by takové buňky byly opraveny reparačními mechanizmy za vzniku chyb, což by mohlo mít za následek vznik chromozomových přestaveb [13]. Mechanizmus úniku před buněčnou smrtí však zatím nebyl dostatečně prozkoumán.
Výskyt chromotripse u nádorů
Chromotripse bývá asociována s mnoha typy nádorů. Vzhledem k pozorovaným rysům by se dala považovat za spouštěč nebo jeden z prvních zásahů při nádorové transformaci. Jsou známy případy, kdy byla chromotripse prokázána prakticky ve všech buňkách jednoho nádoru, což by odpovídalo teorii, že chromotripse vzniká v relativně brzkém stadiu onemocnění [1,5]. Zároveň ovšem existují ojedinělé případy, kdy byla chromotripse po léčbě reverzibilní, což by znamenalo, že se nepodílela na rozvoji nádoru, ani na jeho následném relapsu [14]. Výsledky jiných studií naopak ukazují, že se chromotripse vyskytuje v počátku onemocnění subklonálně a může docházet k její pozdější expanzi, např. až v relapsu [15]. V těchto případech by chromotripse byla důsledkem klonální evoluce.
Co se týče výskytu chromotripse u jednotlivých typů nádorů, rozdílné malignity vykazují odlišnou četnost [16]. Chromotripse byla popsána u melanomů, sarkomů, gliomů i hematoonkologických onemocnění. Podle dostupných analýz se její výskyt u všech typů nádorových onemocnění pohybuje mezi 2–3 % [1], skutečná incidence však bude pravděpodobně mnohem vyšší, než se původně předpokládalo, a to především u agresivnějších typů nádorů. Dostupné údaje o frekvenci výskytu chromotripse u jednotlivých onkologických diagnóz jsou uvedeny v tab. 1. Ne vždy jsou uvedené četnosti přesné, jelikož jednotlivé studie používají odlišné detekční metody. Stejně tak definice a analyzované soubory pacientů nejsou vždy reprezentativní. Velkým nedostatkem je obvykle malý vzorek testovaných pacientů.
Tab. 1. Výskyt chromotripse u jednotlivých typů nádorů dle dostupných publikovaných dat. 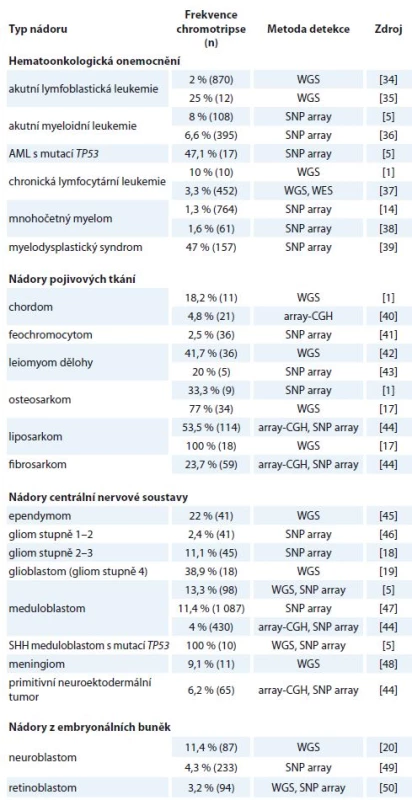

AML – akutní myeloidní leukemie, SHH meduloblastom – meduloblastom s aktivovanou Sonic-Hedgehog signalizací, WGS – celogenomové sekvenování, SNP – detekce jednonukleotidových polymorfizmů, WES – celoexomové sekvenování, CGH – komparativní genomová hybridizace Genetické změny asociované s chromotripsí
Mezi patologické změny, které mohou být příčinou nebo doprovodným jevem chromotripse, patří mimo jiné inaktivace nádorových supresorů. K potlačení jejich funkce obvykle dochází prostřednictvím delece. Nejčastěji mutovaným nádorovým supresorem spojovaným s chromotripsí je gen TP53, popsány však byly také mutace genů jako ATM, PTEN, BRCA1, BRCA2, APC a SMAD4 [5,17,18].
Chromotripse je také spojena s amplifikací typických onkogenů, jako je např. CCND1, CDK4, MDM2, SETDB1, ERBB3, ERBB2, MYC a MYCN [17,19,20]. K jejich amplifikaci často dochází prostřednictvím double-minute chromozomů. Zajímavé poznatky přinesla studie z roku 2018 hodnotící vliv dávky mutantního genu KRAS na biologii a fenotypové projevy duktálního adenokarcinomu pankreatu [21]. KRAS patří mezi protoonkogeny rodiny RAS, které jsou prostřednictvím mutací a genových duplikací aktivovány přibližně u 30 % nádorů. V uvedené studii byla pozorována synergie mezi zvýšenou genovou dávkou mutantního KRAS a inaktivací genů TP53, CDKN2A a TGFβ. Kombinace těchto abnormalit byla asociována se vznikem chromotripse a velmi agresivním fenotypem duktálního karcinomu.
U všech typů nádorů platí, že s vyšší četností mutací TP53 stoupá i pravděpodobnost výskytu chromotripse [22]. Gen TP53 se účastní regulace mnoha procesů a hraje významnou roli v zajištění stability genomu. Mutace genu TP53 mohou mít různé následky, mezi něž patří potlačení funkce reparačních mechanizmů reagujících na poškození DNA, deregulace kontroly průchodu buněčným cyklem či narušení údržby telomer. Všechny tyto události mohou přispívat ke vzniku chromotripse. S vysokou frekvencí je chromotripse pozorována u pacientů s vrozenými mutacemi genu TP53, jako jsou např. pacienti s Li-Fraumeniho syndromem [5]. Tito pacienti mají predispozice ke vzniku rekurentních nádorů, mimo jiné se u nich tvoří meduloblastomy. V případě meduloblastomů s aktivovanou Sonic-Hedgehog signalizací (tzv. SHH-meduloblastomů) byla chromotripse pozorována ve 100 % případů. Výskyt chromotripse byl však prokázán i u pacientů se somatickými mutacemi genu TP53, které vznikly v průběhu života, a to např. u akutní myeloidní leukemie [5,23].
Kromě inaktivace nádorových supresorů a aktivace onkogenů může v důsledku chromotripse také docházet k fúzi genů či jejich částí prostřednictvím translokací. Díky tomu vznikají fúzní transkripty, které buď nejsou přepisovány do proteinů, nebo naopak kódují proteiny s novými funkcemi.
Chromotripse a klinický průběh onemocnění
Mnohé studie potvrdily, že existuje spojitost mezi výskytem chromotripse a špatným průběhem onemocnění [1,5,24]. Přítomnost chromozomálních abnormalit, jako jsou hyperdiploidie nebo přestavby chromozomů, má význam pro stanovení prognózy. Při chromotripsi se takové abnormality nehromadí v nádorových buňkách postupně a náhodně v celém genomu, ale vznikají všechny současně za ovlivnění funkce mnoha genů, které často hrají roli v regulaci buněčného cyklu či dysfunkci telomer [23]. To vysvětluje, proč mají buňky poškozené chromotripsí obvykle agresivnější fenotyp [21]. Vzhledem k tomu, že tak rozsáhlé poškození nevede k zániku postižených buněk, lze předpokládat, že vzniklé defekty poskytují buňkám selekční výhodu a umožňují další proliferaci nádorového klonu.
S nepříznivou prognózou pacientů úzce souvisí i rezistence k protinádorové terapii, neboť některé geny ovlivněné chromotripsí mohou být spojeny s rezistencí na konkrétní druhy léčiv [25]. Byly rovněž pozorovány rozdíly mezi jednotlivými subtypy onemocnění, např. u pacientů s karcinomem prsu chromotripse zhoršuje prognózu pouze v případě subtypů s exprimovaným estrogenovým receptorem nebo triple negativních subtypů [26]. Jak již také bylo zmíněno, k chromotripsi může docházet až po léčbě. Takto byla pozorována rezistence na terapii v důsledku pozdější selekce chromotripse u pacientů s mnohočetným myelomem [27].
Význam chromotripse v klinické praxi
Ačkoli je nutné provést rozsáhlejší studie, dostupná data poukazují na skutečnost, že chromotripse je markerem nepříznivého průběhu nádorových onemocnění. Proto by mohla její detekce přispět ke stanovení prognózy, stagingu pacientů či volbě individuální cílené terapie. Například pacienti s glioblastomy s chromotripsí by mohli profitovat z léčby cílené na receptory epidermálních růstových faktorů a jiné tyrozinkinázové receptory, které bývají v důsledku chromotripse amplifikovány [28]. Dalším příkladem je léčba cílená na protoonkogen PIM1 u pacientů s akutní myeloidní leukemií, který má funkci serin/treoninové kinázy a podílí se na buněčné proliferaci a u akutní myeloidní leukemie je nejčastěji deregulovaným genem v důsledku chromotripse [23].
Kromě toho bylo zjištěno, že v důsledku přestaveb způsobených chromotripsí mohou vznikat potenciální neoantigeny, které vážou molekuly hlavního histokompatibilního komplexu a umožňují expanzi T buněčných klonů [29]. Na základě těchto poznatků je možné vyvíjet nové imunoterapeutické postupy a lépe vyhodnotit, kterým pacientům imunoterapii indikovat.
Některé studie také navrhují, že by exprese fúzních genů vzniklých v důsledku chromotripse mohla napomoci zvýšení citlivosti buněk na určité druhy léčiv [30]. Jako příklad lze uvést fúzní transkript ETV6-PDGFRB identifikovaný u pacienta s myelodysplastickým syndromem [31]. Podobné fúzní varianty vzniklé následkem chromotripse by mohly posloužit jako potenciální terapeutický cíl – např. konkrétně přestavby genu PDGFRB jsou dle klasifikace Světové zdravotnické organizace charakteristickým znakem subtypu myeloidních neoplazií, které jsou senzitivní vůči tyrozikinázovým inhibitorům.
Dalo by se využít i poznatku, že nádorové buňky s chromotripsí obvykle mívají poškozený mitotický aparát a narušenou funkci systému odpovědi buňky na poškození DNA (DNA damage response – DDR). Systém DDR zahrnuje signalizační a opravné procesy, které vedou k zastavení buněčného cyklu a opravě poškozené DNA. V případě, že je poškození tak rozsáhlé, že není možná oprava, buňka spouští apoptózu. DDR tedy působí jako protinádorová bariéra, zatímco mutace v genech, které se účastní DDR, se podílejí na progresi nádoru. Jelikož se může více opravných mechanizmů překrývat a navzájem zastupovat, nabízí se cílit léčbu na ty mechanizmy, na nichž je buňka s určitým poškozením DDR závislá. Jako příklad lze uvést glioblastomy, u nichž dochází k posílení funkce DDR [32]. Tyto nádory jsou rezistentní vůči konvenční terapii a je vhodné zvolit léčbu pomocí inhibitorů cílených na jednotlivé faktory DDR. Vhodnými cílovými molekulami mohou být např. serin/threoninové kinázy Chk1 a Chk2, které se významně podílejí na zajištění opravy poškozené DNA.
Ve velmi výjimečných případech může chromotripse vést také k vyléčení závažné choroby, jak tomu bylo v kazuistice zaznamenané v roce 2015 [33]. Pacient trpěl imunodeficiencí způsobenou vrozenou hyperaktivitou chemokinového receptoru CXCR4. Prostřednictvím chromotripse v hematopoetické kmenové buňce u něj došlo k deleci abnormální kopie genu kódujícího tento receptor, čímž byla obnovena normální funkce jeho imunitního systému.
Závěr
Od popsání chromotripse v roce 2011 bylo provedeno a publikováno mnoho navazujících studií tohoto zajímavého jevu. Kromě snahy objasnit mechanizmy vzniku chromotripse a její důsledky v patofyziologii buňky se do středu zájmu dostalo i potenciální uplatnění získaných poznatků v klinické oblasti. Aby to však bylo možné, bude potřeba provést prospektivní analýzu asociace mezi výskytem chromotripse a průběhem onemocnění. Pro využití v oblasti cílené nádorové terapie bude také nutné definovat diagnostická doporučení pro její vyšetřování u jednotlivých diagnóz.
Podpořeno z programového projektu Ministerstva zdravotnictví ČR s reg. č. 15-31834A.
Veškerá práva podle předpisů na ochranu duševního vlastnictví jsou vyhrazena.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
Mgr. Karla Plevová, Ph.D.
Centrum molekulární biologie a genové terapie Interní hematologická a onkologická klinika LF MU a FN Brno
Černopolní 9 625 00 Brno
e-mail: plevova.karla@fnbrno.cz
prof. RNDr. Šárka Pospíšilová, Ph.D.
Centrum molekulární biologie a genové terapie Interní hematologická a onkologická klinika LF MU a FN Brno
Černopolní 9 625 00 Brno
e-mail: pospisilova.sarka@fnbrno.cz
Obdrženo: 31. 12. 2018
Přijato: 19. 3. 2019
Zdroje
1. Stephens PJ, Greenman CD, Fu B et al. Massive genomic rearrangement acquired in a single catastrophic event during cancer development. Cell 2011; 144 (1): 27–40. doi: 10.1016/j.cell.2010.11.055.
2. Kloosterman WP, Koster J, Molenaar JJ. Prevalence and clinical implications of chromothripsis in cancer genomes. Curr Opin Oncol 2014; 26 (1): 64–72. doi: 10.1097/CCO.0000000000000038.
3. Kloosterman WP, Guryev V, van Roosmalen M et al. Chromothripsis as a mechanism driving complex de novo structural rearrangements in the germline. Hum Mol Genet 2011; 20 (10): 1916–1924. doi: 10.1093/hmg/ ddr073.
4. de Pagter MS, van Roosmalen MJ, Baas AF et al. Chromothripsis in healthy individuals affects multiple protein-coding genes and can result in severe congenital abnormalities in offspring. Am J Hum Genet 2015; 96 (4): 651–656. doi: 10.1016/j.ajhg.2015.02.005.
5. Rausch T, Jones DT, Zapatka M et al. Genome sequencing of pediatric medulloblastoma links catastrophic DNA rearrangements with TP53 mutations. Cell 2012; 148 (1–2): 59–71. doi: 10.1016/j.cell.2011.12.013.
6. Holland AJ, Cleveland DW. Chromoanagenesis and cancer: mechanisms and consequences of localized, complex chromosomal rearrangements. Nat Med 2012; 18 (11): 1630–1638. doi: 10.1038/nm.2988.
7. Crasta K, Ganem NJ, Dagher R et al. DNA breaks and chromosome pulverization from errors in mitosis. Nature 2012; 482 (7383): 53–58. doi: 10.1038/nature10802.
8. Liu S, Kwon M, Mannino M et al. Nuclear envelope assembly defects link mitotic errors to chromothripsis. Nature 2018; 561 (7724): 551–555. doi: 10.1038/s41586-018-0534-z.
9. Huang Y, Jiang L, Yi Q et al. Lagging chromosomes entrapped in micronuclei are not “lost” by cells. Cell Res 2012; 22 (5): 932–935. doi: 10.1038/cr.2012.26.
10. Artandi SE, Chang S, Lee SL et al. Telomere dysfunction promotes non-reciprocal translocations and epithelial cancers in mice. Nature 2000; 406 (6796): 641–645.
11. Shim G, Ricoul M, Hempel WM et al. Crosstalk between telomere maintenance and radiation effects: a key player in the process of radiation-induced carcinogenesis. Mutat Res Rev Mutat Res 2014; S1383–S5742 (14): 00002-7. doi: 10.1016/j.mrrev.2014.01.001.
12. Halazonetis TD, Gorgoulis VG, Bartek J. An oncogene-induced DNA damage model for cancer development. Science 2008; 319 (5868): 1352–1355. doi: 10.1126/science.1140735.
13. Tubio JM, Estivill X. Cancer: when catastrophe strikes a cell. Nature 2011; 470 (7335): 476–477. doi: 10.1038/470476a.
14. Bassaganyas L, Beà S, Escaramís G et al. Sporadic and reversible chromothripsis in chronic lymphocytic leukemia revealed by longitudinal genomic analysis. Leukemia 2013; 27 (12): 2376–2379. doi: 10.1038/leu.2013.127.
15. Federer-Gsponer JR, Quintavalle C, Müller DC et al. Delineation of human prostate cancer evolution identifies chromothripsis as a polyclonal event and FKBP4 as a potential driver of castration resistence. J Pathol 2018; 245 (1): 74–84. doi: 10.1002/path.5052.
16. Jones MJ, Jallepalli PV. Chromothripsis: chromosomes in crisis. Dev Cell 2012; 23 (5): 908–917. doi: 10.1016/j.devcel.2012.10.010.
17. Cortés-Ciriano I, Lee JK, Xi R et al. Comprehensive analysis of chromothripsis in 2,658 human cancers using whole-genome sequencing. [online]. Available from: https: //www.biorxiv.org/content/biorxiv/early/2018/05/30/333617.full.pdf.
18. Cohen A, Sato M, Aldape K et al. DNA copy number analysis of Grade II–III and Grade IV gliomas reveals differences in molecular ontogeny including chromothripsis associated with IDH mutation status. Acta Neuropathol Commun 2015; 3 : 34. doi: 10.1186/s40478-015-0213-3.
19. Malhotra A, Lindberg M, Faust GG et al. Breakpoint profiling of 64 cancer genomes reveals numerous complex rearrangements spawned by homology-independent mechanisms. Genome Res 2013; 23 (5): 762–776. doi: 10.1101/gr.143677.112.
20. Molenaar JJ, Koster J, Zwijnenburg DA et al. Sequencing of neuroblastoma identifies chromothripsis and defects in neuritogenesis genes. Nature 2012; 483 (7391): 589–593. doi: 10.1038/nature10910.
21. Mueller S, Engleitner T, Maresch R et al. Evolutionary routes and KRAS dosage define pancreatic cancer phenotypes. Nature 2018; 554 (7690): 62–68. doi: 10.1038/nature25459.
22. Zhang CZ, Leibowitz ML, Pellman D. Chromothripsis and beyond: rapid genome evolution from complex chromosomal rearrangements. Genes Dev 2013; 27 (23): 2513–2530. doi: 10.1101/gad.229559.113.
23. Rücker FG, Dolnik A, Blätte TJ et al. Chromothripsis is linked to TP53 alteration, cell cycle impairment, and dismal outcome in acute myeloid leukemia with complex karyotype. Hematologica 2018; 103 (1): e17–e20. doi: 10.3324/haematol.2017.180497.
24. Magrangeas F, Avet-Loiseau H, Munshi NC et al. Chromothripsis identifies a rare and aggressive entity among newly diagnosed multiple myeloma patients. Blood 2011; 118 (3): 675–678. doi: 10.1182/blood-2011-03-344069.
25. Forment JV, Kaidi A, Jackson SP. Chromothripsis and cancer: causes and consequences of chromosome shattering. Nat Rev Cancer 2012; 12 (10): 663–670. doi: 10.1038/nrc3352.
26. Przybytkowski E, Lenkiewicz E, Barrett MT et al. Chromosome-breakage genomic instability and chromothripsis in breast cancer. BMC Genomics 2014; 15 : 579. doi: 10.1186/1471-2164-15-579.
27. Lee KJ, Lee KH, Yoon KA et al. Chromothripsis in treatment resistance in multiple myeloma. Genomics Inform 2017; 15 (3): 87–97. doi: 10.5808/GI.2017.15.3.87.
28. Furgason JM, Koncar RF, Michelhaugh SK et al. Whole genome sequence analysis links chromothripsis to EGFR, MDM2, MDM4, and CDK4 amplification in glioblastoma. Oncoscience 2015; 2 (7): 618–628.
29. Mansfield AS, Peikert T, Smadbeck JB et al. Neoantigenic potential of complex chromosomal rearrangements in mesothelioma. J Thorac Oncol 2019; 14 (2): 276–287. doi: 10.1016/j.jtho.2018.10.001.
30. Nijhawan D, Zack TI, Ren Y et al. Cancer vulnerabilities unveiled by genomic loss. Cell 2012; 150 (4): 842–854. doi: 10.1016/j.cell.2012.07.023.
31. Singh ZN, Richards S, El Chaer F et al. Cryptic ETV6-PDGFRB fusion in a highly complex rearrangement of chromosomes 1, 5 and 12 due to a chromothripsis-like event in a myelodysplastic syndrome/myeloproliferative neoplasm. Leuk Lymphoma 2018; 11 : 1–4. doi: 10.1080/10428194.2018.1480774.
32. Bao S, Wu Q, McLendon RE et al. Glioma stem cells promote radioresistance by preferential activation of the DNA damage response. Nature 2006; 444 (7120): 756–760.
33. McDermott DH, Gao JL, Liu Q et al. Chromothriptic cure of WHIM syndrome. Cell 2015; 160 (4): 686–699. doi: 10.1016/j.cell.2015.01.014.
34. Li Y, Schwab C, Ryan S et al. Constitutional and somatic rearrangement of chromosome 21 in acute lymphoblastic leukaemia. Nature 2014; 508 (7494): 98–102. doi: 10.1038/nature13115.
35. Zhang J, Ding L, Holmfeldt L et al. The genetic basis of early T-cell precursor acute lymphoblastic leukaemia. Nature 2012; 481 (7380): 157–163. doi: 10.1038/ nature10725.
36. Fontana MC, Marconi G, Feenstra JD et al. Chromothripsis in acute myeloid leukemia: biological features and impact on survival. Leukemia 2018; 32 (7): 1609–1620. doi: 10.1038/s41375-018-0035-y.
37. Puente XS, Beà S, Valdés-Mas R et al. Non-coding recurrent mutations in chronic lymphocytic leukaemia. Nature 2015; 526 (7574): 519–524. doi: 10.1038/nature14666.
38. Stevens-Kroef M, Weghuis DO, Croockewit S et al. High detection rate of clinically relevant genomic abnormalities in plasma cells enriched from patients with multiple myeloma. Genes Chromosomes Cancer 2012; 51 (11): 997–1006. doi: 10.1002/gcc.21982.
39. Zemanova Z, Michalova K, Buryova H et al. Involvement of deleted chromosome 5 in complex chromosomal aberrations in newly diagnosed myelodysplastic syndromes (MDS) is correlated with extremely adverse prognosis. Leuk Res 2014; 38 (5): 537–544. doi: 10.1016/j.leukres.2014.01.012.
40. Le LP, Nielsen GP, Rosenberg AE et al. Recurrent chromosomal copy number alterations in sporadic chordomas. PLoS One 2011; 6 (5): e18846. doi: 10.1371/journal.pone.0018846.
41. Flynn A, Benn D, Clifton-Bligh R et al. The genomic landscape of phaeochromocytoma. J Pathol 2015; 236 (1): 78–89. doi: 10.1002/path.4503.
42. Mehine M, Kaasinen E, Mäkinen N et al. Characterization of uterine leiomyomas by whole-genome sequencing. N Engl J Med 2013; 369 (1): 43–53.
43. Holzmann C, Markowski DN, Koczan D et al. Cytogenetically normal uterine leiomyomas without MED12-mutations – a source to identify unknown mechanisms of the development of uterine smooth muscle tumors. Mol Cytogenet 2014; 7 (1): 88. doi: 10.1186/s13039-014-0088-1.
44. Cai H, Kumar N, Bagheri HC et al. Chromothripsis-like patterns are recurring but heterogeneously distributed features in a survey of 22,347 cancer genome screens. BMC Genomics 2014; 15 : 82. doi: 10.1186/1471-2164-15-82.
45. Parker M, Mohankumar KM, Punchihewa C et al. C11orf95-RELA fusions drive oncogenic NF-κB signalling in ependymoma. Nature 2014; 506 (7489): 451–455. doi: 10.1038/nature13109.
46. Lhotská H, Zemanová Z, Kramář F et al. Molekulárně cytogenetická analýza chromozomových aberací v buňkách nízkostupňových gliomů a její přínos pro klasifikaci nádoru. Klin Onkol 2014; 27 (3): 183–191.
47. Northcott PA, Shih DJ, Peacock J et al. Subgroup-specific structural variation across 1,000 medulloblastoma genomes. Nature 2012; 488 (7409): 49–56. doi: 10.1038/nature11327.
48. Brastianos PK, Horowitz PM, Santagata S et al. Genomic sequencing of meningiomas identifies oncogenic SMO and AKT1 mutations. Nat Genet 2013; 45 (3): 285–289. doi: 10.1038/ng.2526.
49. Ambros IM, Brunner C, Abbasi R et al. Ultra-high density SNParray in neuroblastoma molecular diagnostics. Front Oncol 2014; 4 : 202. doi: 10.3389/fonc.2014. 00202.
50. McEvoy J, Nagahawatte P, Finkelstein D et al. RB1 gene inactivation by chromothripsis in human retinoblastoma. Oncotarget 2014; 5 (2): 438–450. doi: 10.18632/oncotarget.1686.
51. Nones K, Waddell N, Wayte N et al. Genomic catastrophes frequently arise in esophageal adenocarcinoma and drive tumorigenesis. Nat Commun 2014; 5 : 5224. doi: 10.1038/ncomms6224.
52. Bass AJ, Lawrence MS, Brace LE et al. Genomic sequencing of colorectal adenocarcinomas identifies a recurrent VTI1A-TCF7L2 fusion. Nat Genet 2011; 43 (10): 964–968. doi: 10.1038/ng.936.
53. Kloosterman WP, Hoogstraat M, Paling O et al. Chromothripsis is a common mechanism driving genomic rearrangements in primary and metastatic colorectal cancer. Genome Biol 2011; 12 (10): R103. doi: 10.1186/gb-2011-12-10-r103.
54. Fernandez-Banet J, Lee NP, Chan KT et al. Decoding complex patterns of genomic rearrangement in hepatocellular carcinoma. Genomics 2014; 103 (2–3): 189–203. doi: 10.1016/j.ygeno.2014.01.003.
55. Jiang Z, Jhunjhunwala S, Liu J et al. The effects of hepatitis B virus integration into the genomes of hepatocellular carcinoma patients. Genome Res 2012; 22 (4): 593–601. doi: 10.1101/gr.133926.111.
56. Fraser M, Sabelnykova VY, Yamaguchiet TN et al. Genomic hallmarks of localized, non-indolent prostate cancer. Nature 2017; 541 (7637): 359–364. doi: 10.1038/nature20788.
57. Lapuk AV, Wu C, Wyatt AW et al. From sequence to molecular pathology, and a mechanism driving the neuroendocrine phenotype in prostate cancer. J Pathol 2012; 227 (3): 286–297. doi: 10.1002/path.4047.
58. Nik-Zainal S, Van Loo P, Wedge DC et al. The life history of 21 breast cancers. Cell 2012; 149 (5): 994–1007. doi: 10.1016/j.cell.2012.04.023.
59. Morrison CD, Liu P, Woloszynska-Read A et al. Whole-genome sequencing identifies genomic heterogeneity at a nucleotide and chromosomal level in bladder cancer. Proc Natl Acad Sci U.S.A. 2014; 111 (6): E672–E681. doi: 10.1073/pnas.1313580111.
60. Govindan R, Ding L, Griffith M et al. Genomic landscape of non-small cell lung cancer in smokers and never-smokers. Cell 2012; 150 (6): 1121–1134. doi: 10.1016/j.cell.2012.08.024.
61. Poaty H, Coullin P, Peko JF et al. Genome-wide high-resolution aCGH analysis of gestational choriocarcinomas. PLoS One 2012; 7 (1): e29426. doi: 10.1371/journal.pone.0029426.
62. Hirsch D, Kemmerling R, Davis S et al. Chromothripsis and focal copy number alterations determine poor outcome in malignant melanoma. Cancer Res 2013; 73 (5): 1454–1460. doi: 10.1158/0008-5472.CAN-12-0928.
63. Akbani R, Akdemir KC, Aksoy BA et al. Genomic classification of cutaneous melanoma. Cell 2015; 161 (7): 1681–1696. doi: 10.1016/j.cell.2015.05.044.
64. van Engen-van Grunsven AC, Baar MP, Pfundt R et al. Whole-genome copy-number analysis identifies new leads for chromosomal aberrations involved in the oncogenesis and metastastic behavior of uveal melanomas. Melanoma Res 2015; 25 (3): 200–209. doi: 10.1097/CMR.0000000000000152.
Štítky
Dětská onkologie Chirurgie všeobecná Onkologie
Článek vyšel v časopiseKlinická onkologie
Nejčtenější tento týden
2019 Číslo 2- Metamizol jako analgetikum první volby: kdy, pro koho, jak a proč?
- Nejlepší kůže je zdravá kůže: 3 úrovně ochrany v moderní péči o stomii
- Nejasný stín na plicích – kazuistika
- Metamizol v léčbě různých bolestivých stavů – kazuistiky
-
Všechny články tohoto čísla
- Chromotripse – rozsáhlé chromozomové přestavby a jejich význam u onkologických onemocnění
- Vysoká hladina cirkulujících mikropartikulí u pacientů s BCR/ABL negativními myeloproliferativními chorobami – pilotní studie
- Gastrointestinální stromální tumory rekta – hodnocení dat národního registru s ohledem na využití v klinické praxi
- Gorlinov-Goltzov syndróm
- Extrémny prípad syndrómu rastúceho teratómu retroperitonea testikulárneho pôvodu – klinický management a efekt liečby
- Postižení jater po jedné dávce nivolumabu – kazuistika a přehled literatury
- Leptomeningeálne metastázy karcinómu prsníka liečené dvoma líniami intratekálnej chemoterapie – kazuistika
- IgG4 sklerozující cholangitida – zánět imitující nádor hlavy pankreatu a cholangiokarcinom
- Aktuality z odborného tisku
- Mezinárodní klasifikace nemocí pro onkologii integrovaná s histogenetickým modelem melanomu
- Přizpůsobení nutričních intervencí molekulární patofyziologii nádorové kachexie – jak vyřešit dlouhodobý problém
- Onkologie v obrazech
- Když pomáháme imunitnímu systému
- Léčba relabující/refrakterní akutní lymfoblastické leukemie dnes a zítra
- Oligometastatický karcinom prostaty
- Klinická onkologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Chromotripse – rozsáhlé chromozomové přestavby a jejich význam u onkologických onemocnění
- Gorlinov-Goltzov syndróm
- Oligometastatický karcinom prostaty
- Postižení jater po jedné dávce nivolumabu – kazuistika a přehled literatury
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Autoři: MDDr. Eleonóra Ivančová, PhD., MHA
Autoři: prof. MUDr. Eva Kubala Havrdová, DrSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



