-
Medical journals
- Career
Interakce onkogenních virových proteinů s proteiny rodiny p53
Authors: V. Brychtová; V. Hrabal; B. Vojtěšek
Authors‘ workplace: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno
Published in: Klin Onkol 2019; 32(Supplementum 3): 72-77
Category: Review
doi: https://doi.org/10.14735/amko20193SOverview
Východiska: Buněčná transformace způsobená virovou infekcí je komplexní proces, kterého se účastní virové molekuly, hostitelská buňka a faktory okolního prostředí. Viry nejsou schopné samostatné reprodukce, a proto využívají signální, proteosyntetické a metabolické dráhy hostitelské buňky. Jedním z cílů virových molekul je nádorový supresor p53. Proteiny onkogenních virů jsou schopné funkčně inaktivovat p53, a tím deregulovat expresi mnoha genů zúčastněných při apoptóze, proliferaci a buněčné odpovědi na poškození DNA. Proteiny HbX viru hepatitidy B a NS2 a NS5A viru hepatitidy C interagují s p53, znemožňují jeho lokalizaci do jádra, a tím snižují jeho transkripční aktivitu. Dalším způsobem inaktivace p53 je zvýšení jeho degradace, čehož využívá virus Epstein-Barrové prostřednictvím proteinu BZLF1, virus Kaposiho sarkomu skrze protein LANA a lidské papilomaviry za účasti proteinu E6. Velký T antigen lidského polyomaviru Merkelových buněk přímo neinteraguje s p53, ale snižuje úroveň transkripce zprostředkované p53. Tax protein lidského T buněčného lymfotropního viru 1 posttranslačně modifikuje p53 a brání jeho interakci s transkripčními kofaktory. Protein p53 je jedním z nejprostudovanějších proteinů kvůli své nádorové supresorové funkci a úloze při udržování integrity genomu buňky. Díky tomu jsou také intenzivně studovány jeho evolučně příbuzné homology p63 a p73, které hrají roli při vývoji organizmu. Jejich úloha v onkogenezi ještě nebyla úplně objasněna.
Cíl: Tento přehledový článek popisuje dosud známé interakce proteinů p53, p63 a p73 s proteiny onkogenních virů.
Klíčová slova:
viry – virové proteiny – p53 – p63 – p73 – tumorigeneze
Úvod
Nádorová onemocnění zahrnují komplexní skupinu nemocí s různou etiologií. Jedním z faktorů podporujících nádorovou transformaci buněk jsou infekční agens zahrnující bakterie, viry a parazity [1]. Příspěvek virů k nádorové transformaci není v některých případech zcela jasný, na druhou stranu přítomnost některých virů jako např. lidského papilomaviru (human papillomavirus – HPV) byla celosvětově prokázána u více než 90 % případů invazivního karcinomu děložního hrdla [2]. Je známo, že samotná virová infekce není dostačující a nepůsobí jako kompletní karcinogen pro vznik nádoru, ale její úloha spočívá spíše v iniciaci nádorové transformace a podpoře nádorové progrese.
Lidské onkogenní viry (onkoviry) zahrnují širokou skupinu druhů, které využívají odlišné strategie. Mechanizmy, kterými zásadně přispívají ke vzniku nádorů, mají však společné rysy a ovlivňují stejné dráhy zapojené do kontroly buněčného cyklu [3]. Je známo, že virus v průběhu latentní infekce způsobuje změny na úrovni hostitelského genomu vznikem mutací a chromozomálních změn. Kromě genetických změn se v infikovaných buňkách kumulují i epigenetické změny vedoucí k aberantní metylaci hostitelských genů [4]. Cílem virových proteinů jsou především hostitelské proteiny regulující proliferaci, apoptózu, genomovou stabilitu, diferenciaci a rozpoznání imunitním systémem [5,6]. Z hlediska nádorové biologie jsou významné interakce virových proteinů s nádorovými supresory p53 a Rb, přičemž onkogenní potenciál virů spočívá především v jejich schopnosti potlačovat aktivitu nádorového supresoru p53 [7].
TP53 je nádorový supresorový gen – hlavní člen p53 rodiny proteinů, který se významně podílí na udržení genomové stability buněk. Protein p53 funguje jako kontrolní systém, který reaguje na různé stresové signály v buňkách a reguluje geny reagující na poškození DNA, hypoxii, apoptózu, senescenci a další [8,9]. Množství onkogenních virů kóduje onkoproteiny, které interagují s proteiny p53, p63 a p73 a různými mechanizmy ovlivňují jejich aktivitu. U nádorů spojených s virovou infekcí se zřídka vyskytují mutace nádorových supresorových genů a p53 je většinou inaktivován přímou interakcí s virovým proteinem nebo nepřímo skrze ovlivnění aktivity dalších signálních molekul.
Kromě onkogenních virů s p53 interaguje také další skupina virů, které nemají onkogenní potenciál a jimiž způsobená infekce nevede ke vzniku nádoru. Mezi tyto viry patří např. Zika virus, virus západonilské horečky, HIV-1 virus a další. Přehledně shrnuto v Aloni-Grinstein et al [10].
Onkogenní viry a nádorový supresor p53
Onkogenní viry přispívají k procesu neoplastické transformace buněk. V průběhu koevoluce se svými hostiteli si vyvinuly různé mechanizmy pro zajištění reprodukce a k úniku před imunitním systémem hostitele [11]. V roce 1911 prováděl Peyton Rous experimenty na kuřatech, v rámci nichž zdravá kuřata infikoval filtrátem z tumoru kuřat se sarkomem. U infikovaných kuřat se následně začaly objevovat novotvary. Tyto experimenty vedly k objevu prvního zvířecího onkogenního viru, viru Rousova sarkomu [12]. První lidský onkogenní virus byl objeven až v roce 1964 Epsteinem a Barrovou. Pomocí elektronové mikroskopie pozorovali virové částice v buňkách Burkittova lymfomu [13]. Doposud bylo objeveno nejméně sedm virů, které jsou spojeny s tvorbou malignit u lidí. Patří mezi ně lidský polyomavirus Merkelových buněk (Merkel cell polyomavirus – MCPyV), viry z čeledi Herpesviridae: virus Epstein-Barrové (Epstein-Barr virus – EBV) a virus Kaposiho sarkomu (Kaposi sarcoma-associated virus – KSHV), virus hepatitidy B (hepatitis B virus – HBV), HPV, virus hepatitidy C (hepatitis C virus – HCV), lidský T buněčný lymfotropní virus typu I (human T-lymphotropic virus type 1 – HTLV-1); charakteristiky virů jsou přehledně shrnuty v tab. 1 [14].
Table 1. Charakteristika jednotlivých onkogenních virů vč. jejich taxonomického zařazení, typu a velikosti genomu, buněčného tropizmu, nádorových onemocnění, se kterými jsou spojeny, a virových molekul ovlivňujících aktivitu p53. 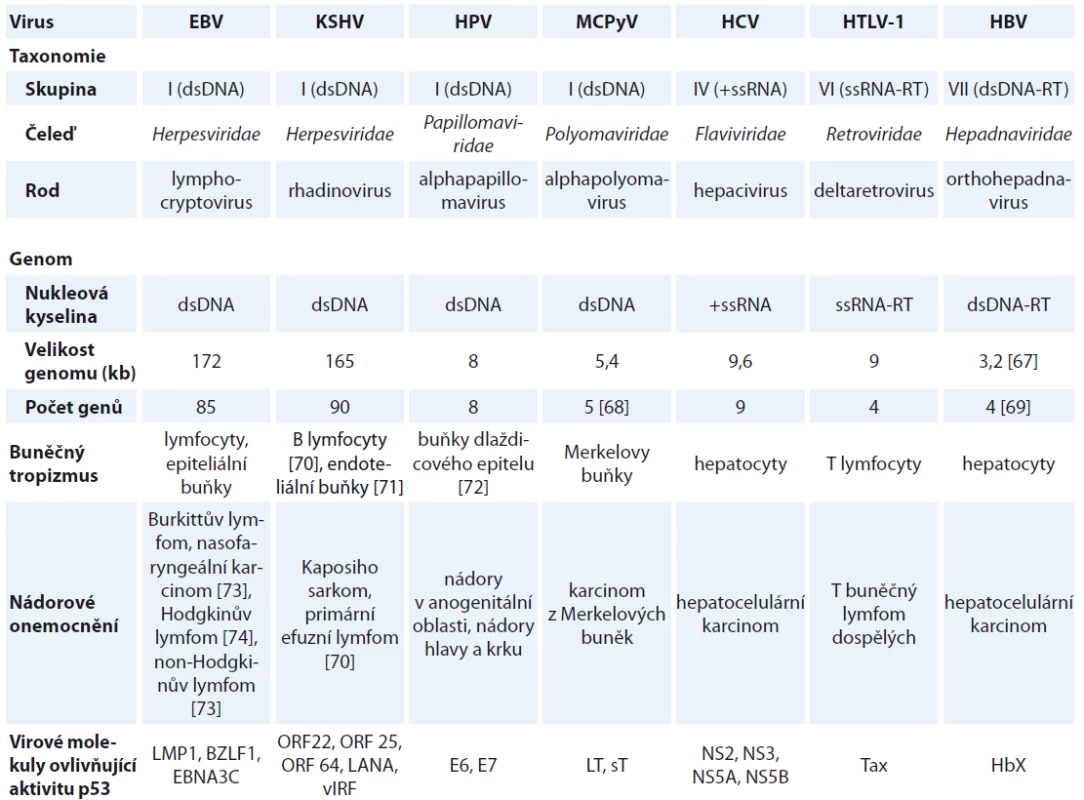
EBV – virus Epstein-Barrové, KSHV – virus Kaposiho sarkomu, HPV – lidský papilomavirus, MCPyV – polyomavirus Merkelových buněk, HCV – virus hepatitidy C, HTLV-1 – lidský T buněčný lymfotropní virus typu I, HBV – virus hepatitidy B, dsDNA – dvouvláknová DNA, ssRNA – jednovláknová RNA, RT – se zpětnou transkripcí Lidský polyomavirus Merkelových buněk
MCPyV je polyomavirus asociovaný s karcinomem z Merkelových buněk, velmi agresivním neuroendokrinním kožním nádorem. Poprvé byly virové sekvence izolovány z karcinomu z Merkelových buněk v roce 2008 [15]. MCPyV je malý neobalený virus s genomem v podobě cirkulární dvouřetězcové molekuly DNA o velikosti přibližně 5,4 kb [16]. Genom MCPyV kóduje proteiny rané fáze exprese: malý T antigen (small T antigen – sT), velký T antigen (large T antigen – LT), 57kT antigen a proteiny pozdní fáze exprese VP1, VP2 [15,17,18].
Velký T antigen se přímo neváže na p53, ale pravděpodobně spolu nepřímo interagují. V luciferázovém reportérovém systému v buňkách H1299 transfekovaných různým titrem velkého T antigenu byla pozorována inhibice transaktivace reportérového konstruktu proteinem p53 [19]. Malý T antigen funguje jako aktivátor transkripce MDM2 a CK1α, který následně ve spolupráci s MDM4 inhibuje funkci p53 v buňkách karcinomu z Merkelových buněk [20].
Virus Epstein-Barrové
EBV je gamma herpesvirus 4 spojený s řadou malignit B lymfocytů, T lymfocytů, NK (natural killer) buněk a epiteliálních karcinomů [5]. Genom EBV je tvořen lineární dvouřetězcovou DNA o délce 172 kb [21] a kóduje přibližně 85 genů [22].
Latentní membránový protein 1 v epiteliálních buňkách indukuje expresi TNFα indukovaného proteinu 3, který zabraňuje apoptóze zprostředkované proteinem p53 [23]. Virový protein BZLF1 funguje jako adaptor Elongin B/ C-Cul2/ 5-SOCS-box protein ubiquitin ligázového komplexu, který označuje p53 pro degradaci, a tím snižuje jeho hladinu v buňce [24]. Jaderný antigen 3C viru Epstein-Barrové podporuje tvorbu komplexu s p53 a Gemin3 in vivo, a tím následně inhibuje transaktivační funkci p53 a apoptózu buňky [25].
Virus Kaposiho sarkomu
KSHV neboli lidský herpesvirus 8 je gamma herpesvirus a je jednou z příčin vzniku Kaposiho sarkomu [26]. Genom HHV8 představuje lineární dvouřetězcová DNA o délce přibližně 165 kb [27]. Kóduje přibližně 90 genů a několik nekódujících RNA vč. mikroRNA (miRNA) [27,28].
Virové strukturní proteiny glykoprotein H (ORF22), hlavní kapsidový protein (ORF25) a tegumentový protein (ORF64) inhibují apoptózu zprostředkovanou proteinem p53. Všechny tři proteiny snižují úroveň exprese Bax a PIG3 [29], které jsou regulované proteinem p53 na transkripční úrovni [30,31]. Bax je aktivátor mitochondriální cesty apoptózy [32] a PIG3 se účastní buněčné odpovědi na poškození DNA [31]. S latencí asociovaný jaderný antigen (latency associated nuclear antigen – LANA) interaguje s p53 vazbou na jeho DNA-vazebnou doménu a zvyšuje úroveň polyubiquitinace a následné degradace p53 v proteazomu [33]. Virový interferon regulující faktor (viral interferon regulatory factor – vIRF) interaguje s p53 a snižuje úroveň fosforylace a acetylace p53, což inhibuje transkripční aktivaci proapoptotických proteinů p21 a Bax zprostředkovanou p53 [34].
Virus hepatitidy B
HBV je malý obalený virus, který vykazuje preferenční tropizmus k hepatocytům. Epidemiologické studie ukazují souvislost mezi chronickou infekcí HBV a rozvojem hepatocelulárního karcinomu. Bylo popsáno osm genotypů a genom HBV je co do velikosti nejmenším lidským DNA virem.
Zásadní roli v transformaci hraje virový protein HbX (HBV kódovaný protein X; 16,5 kDa) kódovaný genem X. Jedná se o protein exprimovaný v průběhu akutní i chronické infekce, který se podílí na replikaci viru in vivo jako transaktivátor virových genů i genů hostitelské buňky. Protein HbX je sám o sobě slabým onkogenem, avšak na myších a buněčných modelech bylo ukázáno, že je schopný aktivovat signální dráhy, jako je MAP kinázová, Ras a Src a podporovat tak maligní transformaci buněk [35].
Jednou z hlavních nádorově supresorových funkcí proteinu p53 je schopnost indukovat apoptózu. Řada publikací ukazuje inaktivaci p53 v důsledku jeho interakce s proteinem HbX, který funguje jako transkripční kotransaktivátor. Z hlediska nádorové transformace je významná schopnost proteinu HbX způsobovat defekty v průběhu mitotického dělení. Vazbou na proteiny BubR1 a HBXIP narušuje jejich funkci při tvorbě mikrotubulů a dělicího vřeténka a podporuje tak chromozomální nestabilitu.
Při infekci jaterních buněk dochází k integraci virové DNA do DNA hostitelských buněk, a to jak v repetitivních oblastech, tak v oblastech aktivně přepisovaných genů. Často tak dochází ke vzniku chimérních virově-buněčných proteinů.
Z hlediska inaktivace p53 byly u hepatocelulárního karcinomu popsány dva hlavní mechanizmy účinku onkovirů. Jedním z nich je přímá interakce proteinu HbX s proteinem p53 prostřednictvím jeho C-konce, která brání vstupu p53 do jádra [36]. Druhým je inhibice p53 prostřednictvím interakce s vysoce exprimovanou (v důsledku HbX) dlouhou nekódující RNA HUR1 (lnc HUR1), která inhibuje transkripční aktivaci p21 a Bax [37].
Lidský papilomavirus
HPV je malý neobalený virus tvořený dvouřetězcovou DNA o velikosti přibližně 8 kb, která kóduje osm genů vedoucích ke vzniku strukturních (L1, L2) a nestrukturních proteinů (E1, E2, E4, E5, E6 a E7). Z hlediska nádorové transformace jsou významné virové onkogeny E5, jejichž exprese vede k transformaci hostitelských buněk. Proteiny E6 a E7 interagují s nádorovými supresory p53 a pRb [38]. HPV zahrnují více než 200 typů rozdělených do pěti rodů, přičemž v rámci rodu alfa se na základě jejich onkogenního potenciálu rozlišují low-risk a high-risk kmeny [39]. Typy HPV-16, HPV-18 a HPV-31, HPV-33 a HPV-42 patří do high-risk skupiny a vedou ke vzniku nádorů v anogenitální oblasti a v oblasti hlavy a krku [40]. Typy HPV-6 a HPV-11 patří do skupiny low-risk a infekce jimi má za následek benigní proliferaci buněk [39].
Rozdíl mezi high-risk a low-risk typy HPV spočívá v lokalizaci a afinitě vazby HPV E6 s p53. U high-risk typů mechanizmus degradace vyžaduje nejprve vazbu E6 na krátký motiv LxxLL E6-asociovaného proteinu (E6AP) ubiquitin ligázy, při které dochází k vytvoření vazebné kapsy potřebné pro interakci s p53 [26]. Následuje polyubiquitinace a degradace p53 ubiquitin-proteazomovou dráhou. High-risk E6 touto cestou podporuje proliferaci nádorových buněk [41]. High-risk E6 má dvě vazebná místa pro p53 – jedno v centrální oblasti, které se podílí na tvorbě heterotrimerního komplexu a degradaci p53, a druhé na C-konci p53. Vazby na C-konci se neúčastní E6AP a není relevantní z hlediska degradace p53 [42].
Low-risk HPV E6 také váže p53 v C-oblasti, ale tato interakce nijak neovlivňuje degradaci proteinu p53. Obecně se udává, že low-risk E6 interaguje s p53 s mnohem nižší afinitou [43].
Kromě uvedeného mají high-risk E6 i E7 množství dalších funkcí jako inhibice diferenciace, apoptózy a imunitní odpovědi [44].
Virus hepatitidy C
Podobně jako u HBV je chronická infekce HCV častou příčinou vzniku hepatocelulárního karcinomu. Ačkoliv není zcela zřejmé, zdali je mechanizmus vedoucí ke vzniku hepatocelulárního karcinomu vlivem HCV přímý, nebo nepřímý, mnoho prací poukazuje na epigenetické změny v hepatocytech infikovaných HCV [45].
HCV je malý obalený RNA virus, jehož genom tvořený jednořetězcovou RNA o velikosti přibližně 9,6 kb kóduje polyprotein o velikosti přibližně 3 000 aminokyselin. Virový polyprotein je po translaci štěpen virovými i hostitelskými proteázami na 10 menších strukturních a nestrukturních proteinů (C, E1, E2, p7, NS2, NS3, NS4A, NS4B, NS5A a NS5B), z nichž většina je multifunkčních. Replikace viru probíhá v cytoplazmě, a nedochází tak k integraci virových sekvencí do genomu hostitelské buňky [46,47].
Virové proteiny NS2, NS3 a NS5A jsou zmiňovány v souvislosti s kancerogenezí, přičemž mezi jejich cíle v hostitelských buňkách patří i protein p53. Dráha proteinu p53 se zřejmě stala cílem virových proteinů z důvodu snahy o zablokování apoptózy nebo zastavení růstu infikovaných buněk, které umožňují přežití virů [48 – 50].
Virová cystein-proteáza NS2 vede ke změně lokalizace p53 v buňkách a brání buněčné odpovědi na poškození DNA. Přímá interakce mezi p53 a NS2 ani změna na úrovni regulace exprese p53 řízených genů však nebyla pozorována [48]. Na rozdíl od NS2 interaguje virová serin-proteáza NS3 svým N-koncem s C-koncovou oblastí proteinu p53 v oblasti oligomerizační domény, a je tedy možné, že tak brání tetramerizaci p53 [49]. Dalším z virových proteinů je NS5A, který přímo interaguje s proteinem p53 na jeho N-konci a zadržuje jej v perinukleárním prostoru buněk. Následkem toho klesá hladina proteinu p53 v jádře a je snížena exprese p53 regulovaných genů [50,51].
Některé práce ukazují, že HCV infekce může ovlivňovat aktivitu proteinu p53 nepřímo. NS5B interaguje s RNA helikázou DDX5 (p68), která je transkripčním koaktivátorem p53. Relokalizací DDX5 do cytoplazmy dochází k omezení její aktivity v jádře a snížení exprese p53 [52].
Lidský T buněčný lymfotropní virus typu I
HTLV-1 je první identifikovaný retrovirus, u kterého byla prokázána souvislost s rozvojem nádorového onemocnění, a to T buněčnou lymfocytární leukemií/ lymfomem dospělých (ATLL). Kromě uvedeného způsobuje infekce virem HTLV-1 také další onemocnění, jako je s HTLV-1 spojená myelopatie/ tropická spastická paraparéza, uveitidy a chronické neurovegetativní poruchy. Udává se, že až u 5 % infikovaných jedinců se po době latence, která může trvat řádově desítky let, rozvíjí ATLL [53].
HTLV-1 kóduje virový protein Tax a jeho exprese uděluje hostitelským buňkám imortalizovaný fenotyp a vede ke vzniku nádorů. Cílem proteinu Tax v buňkách jsou klíčové molekuly řídící proliferaci, buněčný cyklus a další významné signální dráhy, mezi jinými i nádorový supresor p53 [54].
Ukazuje se, že většina buněk infikovaných HTLV-1 nese wild type p53, který je ale funkčně inaktivovaný proteinem Tax. Bylo popsáno množství mechanizmů vedoucích k transkripční inaktivaci p53, které nepřímo ovlivňují aktivitu p53. Ve většině případů protein Tax brání interakci transkripčních faktorů s jejich kofaktory, čímž reguluje jejich transkripční aktivitu. Dochází především k hyperfosforylaci p53 na Ser15 a Ser392, která brání interakci s transkripčním faktorem TFIID a transkripčně znevýhodňuje p53 [55], a dále k inhibici p53 fosfatázou Wip1 zprostředkovanou modulací jaderných faktorů p300/ CBP [53].
Onkogenní viry a proteiny p63 a p73
Proteiny p53, p63 a p73 jsou evolučně příbuzné, vykazují vysokou sekvenční homologii a podobnou doménovou strukturu. Obsahují tři hlavní funkční domény: N-koncovou transaktivační doménu (TA), centrální DNA-vazebnou doménu a oligomerizační doménu [56 – 58]. Proteiny p63 a p73 byly popsány roku 1998 [57], resp. 1997 [59], tedy až téměř 20 let po objevu p53 [60].
Latentní membránový protein 1 viru EBV indukuje expresi ΔNp73 v maligních B buňkách [61]. Izoforma ΔNp73 je dominantní negativní inhibitor p53 [62].
Jaderný antigen 3C viru Epstein-Barrové interaguje s p73 a v buněčných liniích SAOS-2 a HCTp53-/ -, které neexprimují p53, inhibuje apoptózu indukovanou doxorubicinem [63].
Jaderný protein viru HCV koimunoprecipituje s izoformami p73α a p73β v buněčných liniích HepG2 a SAOS-2.Interakce jaderného proteinu HCV s oběma izoformami p73 vede k jeho translokaci do jádra buňky. Nízká úroveň exogenní exprese jaderného proteinu HCV v buněčné linii SAOS-2 na reportérovém plazmidu zvyšuje úroveň aktivace promotoru p21 zprostředkované p73α a p73β. Naopak vysoká úroveň exogenní exprese snižuje schopnost p73α aktivovat promotor p21, ale na schopnost aktivace proteinem p73β vliv nemá [64].
V HeLa buňkách vystavených působení cykloheximidu a kotransfekovaných s HPV18 E6 a zvlášť p53, TAp63β nebo ΔNp63β byla pozorována zvýšená rychlost degradace TAp63β ve srovnání s ΔNp63β. Degradace p53 pak probíhala nejrychleji. Izoforma TAp63β hraje úlohu při formaci fokálních adhezí v epiteliálních buňkách. Snížení stability TAp63β vlivem E6 může přispívat k buněčné transformaci indukcí růstu nezávislého na ukotvení buňky [65].
High-risk HPV E7 onkoprotein indukuje expresi proteinu p63, který hraje důležitou roli při opravě poškození DNA v buněčných liniích odvozených od karcinomu děložního čípku. High-risk HPV E7 indukovaná exprese p63 by tedy mohla být zásadním faktorem ovlivňujícím senzitivitu buněk karcinomu děložního čípku k radioterapii [66].
Závěr
Virová onkogeneze je komplexní proces, který závisí na interakci virů s hostitelskými proteiny. Onkoviry kódují regulační proteiny, které mají schopnost pozměňovat buněčnou signalizaci a ovlivňovat tak proliferaci hostitelských buněk. Tumorigeneze související s virovou infekcí spočívá v oslabení aktivity nádorového supresoru p53, přičemž mechanizmus spočívá v přímé či nepřímé interakci. Důsledkem těchto interakcí je snížení stability p53, inhibice transkripce cílových genů p53 nebo zabránění translokace p53 do jádra, kde plní funkci jako transkripční faktor. Kromě interakcí s nádorovým supresorem p53 byly popsány i interakce s příbuznými proteiny p63 a p73, jejichž funkce jsou virovými proteiny také ovlivněny.
Práce byla podpořena projektem MŠMT – NPU I – LO1413.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
Mgr. Veronika Brychtová, Ph.D.
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: vebrychtova@mou.cz
Obdrženo/Submitted: 12. 6. 2019
Přijato/Accepted: 12. 8. 2019
Sources
1. de Martel C, Ferlay J, Franceschi S et al. Global burden of cancers attributable to infections in 2008: a review and synthetic analysis. Lancet Oncol 2012; 13(6): 607 – 615. doi: 10.1016/ S1470-2045(12)70137-7.
2. Walboomers JM, Jacobs MV, Manos MM et al. Human papillomavirus is a necessary cause of invasive cervical cancer worldwide. J Pathol 1999; 189(1): 12 – 19. doi: 10.1002/ (SICI)1096-9896(199909)189 : 1<12::AID-PATH431>3.0.CO;2-F.
3. McLaughlin-Drubin ME, Munger K. Viruses associated with human cancer. Biochim Biophys Acta 2008; 1782(3): 127 – 150. doi: 10.1016/ j.bbadis.2007.12.005.
4. Fernandez AF, Esteller M. Viral epigenomes in human tumorigenesis. Oncogene 2010; 29(10): 1405 – 1420. doi: 10.1038/ onc.2009.517.
5. Mesri EA, Feitelson MA, Munger K. Human viral oncogenesis: a cancer hallmarks analysis. Cell Host Microbe 2014; 15(3): 266 – 282. doi: 10.1016/ j.chom.2014.02.011.
6. Tornesello ML, Annunziata C, Tornesello AL et al. Human oncoviruses and p53 tumor suppressor pathway deregulation at the origin of human cancers. Cancers (Basel) 2018; 10(7): E213. doi: 10.3390/ cancers10070213.
7. Tornesello ML, Buonaguro L, Buonaguro FM. An overview of new biomolecular pathways in pathogen-related cancers. Future Oncol 2015; 11(11): 1625 – 1639. doi: 10.2217/ fon.15.87.
8. Lane DP. Cancer. p53, guardian of the genome. Nature 1992; 358(6381): 15 – 16. doi: 10.1038/ 358015a0.
9. Levine AJ, Oren M. The first 30 years of p53: growing ever more complex. Nat Rev Cancer 2009; 9(10): 749 – 758. doi: 10.1038/ nrc2723.
10. Aloni-Grinstein R, Charni-Natan M, Solomon H et al. p53 and the viral connection: back into the future (double dagger). Cancers (Basel) 2018; 10(6): E178. doi: 10.3390/ cancers10060178.
11. Moore PS, Chang Y. Why do viruses cause cancer? Highlights of the first century of human tumour virology. Nat Rev Cancer 2010; 10(12): 878 – 889. doi: 10.1038/ nrc2961.
12. Rous P. A Sarcoma of the fowl transmissible by an agent separable from the tumor cells. J Exp Med 1911; 13(4): 397 – 411. doi: 10.1084/ jem.13.4.397.
13. Burkitt D. A children’s cancer dependent on climatic factors. Nature 1962; 194 : 232 – 234. doi: 10.1038/ 194232a0.
14. Bouvard V, Baan R, Straif K et al. A review of human carcinogens – part B: biological agents. Lancet Oncol 2009; 10(4): 321 – 322.
15. Feng H, Shuda M, Chang Y et al. Clonal integration of a polyomavirus in human Merkel cell carcinoma. Science 2008; 319(5866): 1096 – 1100. doi: 10.1126/ science.1152586.
16. MacDonald M, You J. Merkel cell polyomavirus: a new DNA virus associated with human cancer. Adv Exp Med Biol 2017; 1018 : 35 – 56. doi: 10.1007/ 978-981-10-5765-6_4.
17. Shuda M, Arora R, Kwun HJ et al. Human Merkel cell polyomavirus infection I. MCV T antigen expression in Merkel cell carcinoma, lymphoid tissues and lymphoid tumors. Int J Cancer 2009; 125(6): 1243 – 1249. doi: 10.1002/ ijc.24510.
18. Shuda M, Feng H, Kwun HJ et al. T antigen mutations are a human tumor-specific signature for Merkel cell polyomavirus. Proc Natl Acad Sci USA 2008; 105(42): 16272 – 16277. doi: 10.1073/ pnas.0806526105.
19. Borchert S, Czech-Sioli M, Neumann F et al. High-affinity Rb binding, p53 inhibition, subcellular localization, and transformation by wild-type or tumor-derived shortened Merkel cell polyomavirus large T antigens. J Virol 2014; 88(6): 3144 – 3160. doi: 10.1128/ JVI.02916-13.
20. Park DE, Cheng J, Berrios C et al. Dual inhibition of MDM2 and MDM4 in virus-positive Merkel cell carcinoma enhances the p53 response. Proc Natl Acad Sci USA 2019; 116(3): 1027 – 1032. doi: 10.1073/ pnas.1818798116.
21. Baer R, Bankier AT, Biggin MD et al. DNA sequence and expression of the B95-8 Epstein-Barr virus genome. Nature 1984; 310(5974): 207 – 211. doi: 10.1038/ 310207a0.
22. de Jesus O, Smith PR, Spender LC et al. Updated Epstein-Barr virus (EBV) DNA sequence and analysis of a promoter for the BART (CST, BARF0) RNAs of EBV. J Gen Virol 2003; 84(Pt 6): 1443 – 1450. doi: 10.1099/ vir.0.19054-0.
23. Fries KL, Miller WE, Raab-Traub N. Epstein-Barr virus latent membrane protein 1 blocks p53-mediated apoptosis through the induction of the A20 gene. J Virol 1996; 70(12): 8653 – 8659.
24. Sato Y, Kamura T, Shirata N et al. Degradation of phosphorylated p53 by viral protein-ECS E3 ligase complex. PLoS Pathog 2009; 5(7): e1000530. doi: 10.1371/ journal.ppat.1000530.
25. Cai Q, Guo Y, Xiao B et al. Epstein-Barr virus nuclear antigen 3C stabilizes Gemin3 to block p53-mediated apoptosis. PLoS Pathog 2011; 7(12): e1002418. doi: 10.1371/ journal.ppat.1002418.
26. Chang Y, Cesarman E, Pessin MS et al. Identification of herpesvirus-like DNA sequences in AIDS-associated Kaposi’s sarcoma. Science 1994; 266(5192): 1865 – 1869. doi: 10.1126/ science.7997879.
27. Russo JJ, Bohenzky RA, Chien MC et al. Nucleotide sequence of the Kaposi sarcoma-associated herpesvirus (HHV8). Proc Natl Acad Sci USA 1996; 93(25): 14862 – 14867. doi: 10.1073/ pnas.93.25.14862.
28. Samols MA, Hu J, Skalsky RL et al. Cloning and identification of a microRNA cluster within the latency-associated region of Kaposi’s sarcoma-associated herpesvirus. J Virol 2005; 79(14): 9301 – 9305. doi: 10.1128/ JVI.79.14.9301-9305.2005.
29. Chudasama P, Konrad A, Jochmann R et al. Structural proteins of Kaposi’s sarcoma-associated herpesvirus antagonize p53-mediated apoptosis. Oncogene 2015; 34(5): 639 – 649. doi: 10.1038/ onc.2013.595.
30. Miyashita T, Reed JC. Tumor suppressor p53 is a direct transcriptional activator of the human bax gene. Cell 1995; 80(2): 293 – 299. doi: 10.1016/ 0092-8674(95)90412-3.
31. Polyak K, Xia Y, Zweier JL et al. A model for p53-induced apoptosis. Nature 1997; 389(6648): 300 – 305. doi: 10.1038/ 38525.
32. Gross A, Jockel J, Wei M et al. Enforced dimerization of BAX results in its translocation, mitochondrial dysfunction and apoptosis. EMBO J 1998; 17(14): 3878 – 3885. doi: 10.1093/ emboj/ 17.14.3878.
33. Suzuki T, Isobe T, Kitagawa M et al. Kaposi’s sarcoma-associated herpesvirus-encoded LANA positively affects on ubiquitylation of p53. Biochem Biophys Res Commun 2010; 403(2): 194 – 197. doi: 10.1016/ j.bbrc.2010.11.004.
34. Nakamura H, Li M, Zarycki J et al. Inhibition of p53 tumor suppressor by viral interferon regulatory factor. J Virol 2001; 75(16): 7572 – 7582. doi: 10.1128/ JVI.75.16.7572-7582.2001.
35. Andrisani OM, Barnabas S. The transcriptional function of the hepatitis B virus X protein and its role in hepatocarcinogenesis (Review). Int J Oncol 1999; 15(2): 373 – 379. doi: 10.3892/ ijo.15.2.373.
36. Elmore LW, Hancock AR, Chang SF et al. Hepatitis B virus X protein and p53 tumor suppressor interactions in the modulation of apoptosis. Proc Natl Acad Sci USA 1997; 94(26): 14707 – 14712. doi: 10.1073/ pnas.94.26.14707.
37. Liu N, Liu Q, Yang X et al. Hepatitis B virus-upregulated LNC-HUR1 promotes cell proliferation and tumorigenesis by blocking p53 activity. Hepatology 2018; 68(6): 2130 – 2144. doi: 10.1002/ hep.30098.
38. Vousden KH, Doniger J, DiPaolo JA et al. The E7 open reading frame of human papillomavirus type 16 encodes a transforming gene. Oncogene Res 1988; 3(2): 167 – 175.
39. Doorbar J, Egawa N, Griffin H et al. Human papillomavirus molecular biology and disease association. Rev Med Virol 2015; 25 (Suppl 1): 2 – 23. doi: 10.1002/ rmv.1822.
40. Zheng ZM, Baker CC. Papillomavirus genome structure, expression, and post-transcriptional regulation. Front Biosci 2006; 11 : 2286 – 2302.
41. Crook T, Tidy JA, Vousden KH. Degradation of p53 can be targeted by HPV E6 sequences distinct from those required for p53 binding and trans-activation. Cell 1991; 67(3): 547 – 556. doi: 10.1016/ 0092-8674(91)90529-8.
42. Li X, Coffino P. High-risk human papillomavirus E6 protein has two distinct binding sites within p53, of which only one determines degradation. J Virol 1996; 70(7): 4509 – 4516.
43. Oh ST, Longworth MS, Laimins LA. Roles of the E6 and E7 proteins in the life cycle of low-risk human papillomavirus type 11. J Virol 2004; 78(5): 2620 – 2626. doi: 10.1128/ jvi.78.5.2620-2626.2004.
44. Yugawa T, Kiyono T. Molecular mechanisms of cervical carcinogenesis by high-risk human papillomaviruses: novel functions of E6 and E7 oncoproteins. Rev Med Virol 2009; 19(2): 97 – 113. doi: 10.1002/ rmv.605.
45. Hamdane N, Juhling F, Crouchet E et al. HCV-induced epigenetic changes associated with liver cancer risk persist after sustained virologic response. Gastroenterology 2019; 156(8): 2313 – 2329. doi: 10.1053/ j.gastro.2019.02.038.
46. Bühler S, Bartenschlager R. Promotion of hepatocellular carcinoma by hepatitis C virus. Dig Dis 2012; 30(5): 445 – 452. doi: 10.1159/ 000341688.
47. McGivern DR, SM Lemon. Virus-specific mechanisms of carcinogenesis in hepatitis C virus associated liver cancer. Oncogene 2011; 30(17): 1969 – 1983. doi: 10.1038/ onc.2010.594.
48. Bittar C, Shrivastava S, Bhanja Chowdhury J et al. Hepatitis C virus NS2 protein inhibits DNA damage pathway by sequestering p53 to the cytoplasm. PLoS One 2013; 8(4): e62581. doi: 10.1371/ journal.pone.0062581.
49. Ishido S, Hotta H. Complex formation of the nonstructural protein 3 of hepatitis C virus with the p53 tumor suppressor. FEBS Lett 1998; 438(3): 258 – 262. doi: 10.1016/ s0014-5793(98)01312-x.
50. Lan KH, Sheu ML, Hwang SJ et al. HCV NS5A interacts with p53 and inhibits p53-mediated apoptosis. Oncogene 2002; 21(31): 4801 – 4811. doi: 10.1038/ sj.onc.1205589.
51. Majumder M, Ghosh AK, Steele R et al. Hepatitis C virus NS5A physically associates with p53 and regulates p21/ waf1 gene expression in a p53-dependent manner. J Virol 2001; 75(3): 1401 – 1407. doi: 10.1128/ JVI.75.3.1401-1407.2001.
52. Goh PY, Tan YJ, Lim SP et al. Cellular RNA helicase p68 relocalization and interaction with the hepatitis C virus (HCV) NS5B protein and the potential role of p68 in HCV RNA replication. J Virol 2004; 78(10): 5288 – 5298. doi: 10.1128/ jvi.78.10.5288-5298.2004.
53. Zane L, Yasunaga J, Mitagami Y et al. Wip1 and p53 contribute to HTLV-1 Tax-induced tumorigenesis. Retrovirology 2012; 9 : 114. doi: 10.1186/ 1742-4690-9-114.
54. Pise-Masison CA, Mahieux R, Jiang H et al. Inactivation of p53 by human T-cell lymphotropic virus type 1 Tax requires activation of the NF-kappaB pathway and is dependent on p53 phosphorylation. Mol Cell Biol 2000; 20(10): 3377 – 3386. doi: 10.1128/ mcb.20.10.3377-3386.2000.
55. Tabakin-Fix Y, Azran I, Schavinky-Kharpunsky Y et al. Functional inactivation of p53 by human T-cell leukemia virus type 1 Tax protein: mechanisms and clinical implications. Carcinogenesis 2006; 27(4): 673 – 681. doi: 10.1093/ carcin/ bgi274.
56. Vogelstein B, Kinzler KW. Tumour-suppressor genes. X-rays strike p53 again. Nature 1994; 370(6486): 174 – 175. doi: 10.1038/ 370174a0.
57. Yang A, Kaghad M, Wang Y et al. p63, a p53 homolog at 3q27-29, encodes multiple products with transactivating, death-inducing, and dominant-negative activities. Mol Cell 1998; 2(3): 305 – 316.
58. Yang A, Walker N, Bronson R et al. p73-deficient mice have neurological, pheromonal and inflammatory defects but lack spontaneous tumours. Nature 2000; 404(6773): 99 – 103. doi: 10.1038/ 35003607.
59. Kaghad M, Bonnet H, Yang A et al. Monoallelically expressed gene related to p53 at 1p36, a region frequently deleted in neuroblastoma and other human cancers. Cell 1997; 90(4): 809 – 819. doi: 10.1016/ s0092-8674(00)80540-1.
60. Lane DP, Crawford LV. T antigen is bound to a host protein in SV40-transformed cells. Nature 1979; 278(5701): 261 – 263. doi: 10.1038/ 278261a0.
61. Accardi R, Fathallah I, Gruffat H et al. Epstein-Barr virus transforming protein LMP-1 alters B cells gene expression by promoting accumulation of the oncoprotein DeltaNp73alpha. PLoS Pathog 2013; 9(3): e1003186. doi: 10.1371/ journal.ppat.1003186.
62. Accardi R, Scalise M, Gheit T et al. IkappaB kinase beta promotes cell survival by antagonizing p53 functions through DeltaNp73alpha phosphorylation and stabilization. Mol Cell Biol 2011; 31(11): 2210 – 2226. doi: 10.1128/ MCB.00964-10.
63. Sahu SK, Mohanty S, Kumar A et al. Epstein-Barr virus nuclear antigen 3C interact with p73: Interplay between a viral oncoprotein and cellular tumor suppressor. Virology 2014; 448 : 333 – 343. doi: 10.1016/ j.virol.2013.10.023.
64. Alisi A, Giambartolomei S, Cupelli F et al. Physical and functional interaction between HCV core protein and the different p73 isoforms. Oncogene 2003; 22(17): 2573 – 2580. doi: 10.1038/ sj.onc.1206333.
65. Ben Khalifa Y, Teissier S, Tan MK et al. The human papillomavirus E6 oncogene represses a cell adhesion pathway and disrupts focal adhesion through degradation of TAp63beta upon transformation. PLoS Pathog 2011; 7(9): e1002256. doi: 10.1371/ journal.ppat.1002256.
66. Eldakhakhny S, Zhou Q, Crosbie EJ et al. Human papillomavirus E7 induces p63 expression to modulate DNA damage response. Cell Death Dis 2018; 9(2): 127. doi: 10.1038/ s41419-017-0149-6.
67. Gerlich WH, Robinson WS. Hepatitis B virus contains protein attached to the 5’ terminus of its complete DNA strand. Cell 1980; 21(3): 801 – 809. doi: 10.1016/ 0092-8674(80)90443-2.
68. DeCaprio JA. Merkel cell polyomavirus and Merkel cell carcinoma. Philos Trans R Soc Lond B Biol Sci 2017; 372(1732): 20160276. doi: 10.1098/ rstb.2016.0276.
69. Bouchard MJ, Schneider RJ. The enigmatic X gene of hepatitis B virus. J Virol 2004; 78(23): 12725 – 12734. doi: 10.1128/ JVI.78.23.12725-12734.2004.
70. Staskus KA, Sun R, Miller G et al. Cellular tropism and viral interleukin-6 expression distinguish human herpesvirus 8 involvement in Kaposi’s sarcoma, primary effusion lymphoma, and multicentric Castleman’s disease. J Virol 1999; 73(5): 4181 – 4187.
71. Raghu H, Sharma-Walia N, Veettil MW et al. Kaposi’s sarcoma-associated herpesvirus utilizes an actin polymerization-dependent macropinocytic pathway to enter human dermal microvascular endothelial and human umbilical vein endothelial cells. J Virol 2009; 83(10): 4895 – 4911. doi: 10.1128/ JVI.02498-08.
72. D’Souza G, Burk RD, Zhong Y et al. Cervicovaginal human papillomavirus (HPV)-infection before and after hysterectomy: evidence of different tissue tropism for oncogenic and nononcogenic HPV types in a cohort of HIV-positive and HIV-negative women. Int J Cancer 2012; 131(6): 1472 – 1478. doi: 10.1002/ ijc.27363.
73. Young LS, Murray PG. Epstein-Barr virus and oncogenesis: from latent genes to tumours. Oncogene 2003; 22(33): 5108 – 5121. doi: 10.1038/ sj.onc.1206556.
74. Vrzalikova K, Sunmonu T, Reynolds G et al. Contribution of Epstein-Barr Virus Latent Proteins to the Pathogenesis of Classical Hodgkin Lymphoma. Pathogens 2018; 7(3): E59. doi: 10.3390/ pathogens7030059.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2019 Issue Supplementum 3-
All articles in this issue
- Méně časté mutace EGFR v kontextu léčby nemalobuněčného karcinomu plic
- CRISPR-Cas9 jako nástroj v terapii nádorových onemocnění
- Syntetická letalita – její současné využití a potenciál v onkologické léčbě
- Pokroky ve využití organokovových sloučenin při vývoji protinádorových léčiv
- Souhrn aktuálních poznatků o úloze estrogenového receptoru α v nádorové buněčné signalizaci
- Glykoproteiny v sérech onkologických pacientů
- Glykozylácia ako dôležitý regulátor funkcie protilátok
- Výskum ubikvitinácie proteínov v onkológii
- Dlouhé nekódující RNA – současné možnosti detekce a využití v klinické praxi
- Interakce onkogenních virových proteinů s proteiny rodiny p53
- Editorial 2019
- Kooperace genomických, transkriptomických a proteomických metod v detekci mutovaných proteinů
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Výskum ubikvitinácie proteínov v onkológii
- CRISPR-Cas9 jako nástroj v terapii nádorových onemocnění
- Glykozylácia ako dôležitý regulátor funkcie protilátok
- Méně časté mutace EGFR v kontextu léčby nemalobuněčného karcinomu plic
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

