-
Medical journals
- Career
Výskum ubikvitinácie proteínov v onkológii
: J. Faktor; M. Pjechová; L. Hernychová; B. Vojtěšek
: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno
: Klin Onkol 2019; 32(Supplementum 3): 56-64
: Review
prolekare.web.journal.doi_sk: https://doi.org/10.14735/amko20193SVýchodiská: Ubikvitinácia je dôležitá posttranslačná modifikácia proteínov, ktorá reguluje mnoho signalizačných dráh u eukaryot. Aberantná ubikvitinácia je molekulárnou príčinou niektorých onkologických, neurodegeneratívnych, imunitných a kardiovaskulárnych ochorení. Rozvoj hmotnostne spektrometrických metód ponúka možnosť kvalitatívnej a kvantitatívnej analýzy ubikvitinácie proteínov v biologickom materiáli onkologických pacientov. Výskum ubikvitinácie môže objasniť molekulárnu príčinu zmeny hladiny niektorých proteínov, ktoré vystupujú ako onkogény alebo tumor supresory.
Cieľ: Cieľom článku je priblížiť čitateľovi zmysel a dôležitosť ubikvitinácie v niektorých molekulárnych procesoch prebiehajúcich v ľudskom tele. Predovšetkým je dôraz kladený na popis zapojenia ubikvitinácie do malígnych procesov. Na literárnu rešerš nadväzujeme priblížením procesu hmotnostne spektrometrickej identifikácie ubikvitinácií prostredníctvom diglycylových zbytkov v sekvencii proteínu CHIP. Predstavujeme identifikáciu ubikvitinácie proteínov metódami tandemovej hmotnostnej spektrometrie, proces validácie tandemových hmotnostných spektier a popis časovej závislosti ubikvitinácie proteínu CHIP.
Záver: Literárna rešerš oboznamuje čitateľa so známymi aberantnými mechanizmami ubikvitinácie u malígnych ochorení. Úspešne vytvorená hmotnostne spektrometrická metóda môže slúžiť na identifikáciu pozícií ubikvitinácie v sekvenciách proteínov obsiahnutých v lyzátoch nádorového tkaniva.
Klíčová slova:
ubikvitín – hmotnostná spektrometria – neoplazmy – proteín – proteomika
Úvod
Ubikvitín je krátky polypeptid (8,6 kDa) vyskytujúci sa takmer u všetkých eukaryotických organizmov [1]. Väzba ubikvitínu na proteín prebieha procesom ubikvitinácie, ktorá bola objavená už začiatkom 80. rokov minulého storočia [2]. Popis súvislostí medzi ubikvitináciou a proteostázou bol základom pre ďalšie objavy, ktoré v roku 2004 vyústili v udelenie Nobelovej ceny Aaronovi Ciechanoverovi, Avramovi Hershkovi a Irwinovi Rosemu za objasnenie mechanizmu degradácie proteínov ubikvitináciou [3].
Ubikvitinácia je charakteristická pripojením C-konca ubikvitínu kovalentnou izopeptidickou väzbou k ε-amino skupine cieľového lyzínového zbytku v sekvencii proteínu [4]. Jedná sa o sofistikovaný viacstupňový ATP-dependentný proces katalyzovaný troma skupinami enzýmov, E1, E2 a E3 ligázami. V prvom kroku dochádza k tvorbe thio-esterovej väzby medzi ubikvitínom a enzýmom E1 (ubiquitin activating enzyme). Tvorba komplexu E1-ubikvitín umožňuje tvorbu thio-esterovej väzby medzi enzýmom E2 (ubiquitin conjugating enzyme) a ubikvitínom. Následne je potrebná E3 ubikvitín ligáza, ktorá zaručí väzbu ubikvitínu na špecifické miesta v sekvencii modifikovaného proteínu [5]. Špecificita ubikvitinácie môže byť modulovaná posttranslačnými modifikáciami E2, E3 ubikvitín ligáz. Význam ubikvitinácie závisí tiež na posttranslačných modifikáciách samotného ubikvitínu a na umiestnení modifikácie v sekvencii proteínu [6,7], existuje teda akýsi „ubikvitínový kód“ [8].
Cieľom nášho výskumu je popis ubikvitínového kódu proteínu CHIP. Vzhľadom k prítomnosti množstva potencionálnych ubikvitínačných miest v sekvencii CHIP možno predpokladať autoubikvitináciu v rôznych doménach proteínu v závislosti na stave organizmu. Analýza ubikvitinácie proteínu CHIP za rôznych podmienok pomôže odhaliť preferenčné miesta ubikvitinácie v sekvencii, ktoré môžu súvisieť aj s aktivitou niektorých proteínov zapojených v onkogénnych signalizačných dráhach. K výskumu ubikvitinácie sme vytvorili metódu tandemovej hmotnostnej spektrometrie (liquid chromatography tandem-mass spectrometry – LC-MS/ MS) na identifikáciu diglycylových (GlyGly) zbytkov ubikvitínu na lyzínoch a jej funkčnosť sme overili sledovaním autoubikvitinácie E3 ubikvitín ligázy CHIP za rôznych podmienok. Skúmali sme aj efekt prídavku ATP do reakčnej zmesi a časový priebeh ubikvitinácie proteínu CHIP.
„Ubikvitínový kód“ – modulácia významu ubikvitinácie
K posttranslačnej modifikácii jedinou molekulou ubikvitínu dochádza zriedka, cieľové proteíny sú väčšinou polyubikvitínované. Ubikvitín môže byť ďalej ubikvitínovaný na siedmych zbytkoch lyzínu a topológia polyubikvitinácie môže vytvárať komplexné vzorce s rôznym významom. Aktivita polyubikvitinácie je závislá na ich štruktúre a na množstve ubikvitínov [9]. Najfrekventovanejšia je ubikvitinácia na Lys48 predurčujúca proteín k degradácii v proteazóme procesmi proteolýzy. Bolo dokázané, že najmenej štyri molekuly ubikvitínu musia byť pripojené k lyzínovému zbytku, aby bol rozpoznaný 26S proteazómom [10]. Polyubikvitinácia na Lys63 aktivuje neproteolytické procesy súvisiace s internalizáciou membránových proteínov, odozvou imunitného systému, moduláciou aktivity kináz a fosfatáz zapojených aj v procesoch karcinogenézy [11,12]. Významný je popis polyubikvitinácie Met1, ktorý spája (lineárnu) polyubikvitináciu s aktiváciou dráhy NF-κB (nuclear factor kappa light chain enhancer of activated B cells), ktorá rovnako zohráva dôležitú úlohu v karcinogenéze [13,14].
Ubikvitinácia v súvislosti s karcinogenézou
Ubikvitinácia je úzko spätá s onkologickými ochoreniami, kde zohráva duálnu rolu. Deregulácia E2 a E3 ubikvitín ligáz všeobecne významným spôsobom prispieva k aberantnej ubikvitinácii, čo v mnohých prípadoch vedie k iniciácii a k progresii malígnych ochorení [15]. Medzi E2 ubikvitín ligázy so známou úlohou v kontrole procesov progresie bunečného cyklu patrí UBE2N/ UBE2V1 enzým, ktorý katalyzuje polyubikvitináciu na Lys63. Aberantná polyubikvitinácia na Lys63 spúšťa aberantnú signalizáciu NF-κB, ktorá u mamárneho karcinómu môže viesť k hyperaktivácii zápalu v makroprostredí nádoru [16,17].
E2 ubikvitín ligáza E2-EPF UCP zodpovedá za degradáciu E3 ubikvitín ligázy VHL (von Hippel-Lindau), ktorá destabilizuje transkripčné faktory zapojené v angiogenéze a metastázovaní – vaskulárny endotelový rastový faktor (VEGF), matrixové metaloproteinázy (MMP) a hypoxia-inducible factor 1 (HIF1) [18,19]. Ďalším dobre popísaným príkladom je zapojenie E3 ubikvitín ligázy GP78 do metastázovania sarkómov. Knock-down GP78 vedie k zníženej ubikvitinácii a akumulácii nádorového supresoru KAI1, ktorý navodzuje apoptózu rakovinových buniek a regresiu sarkómov [20]. Inhibícia expresie, príp. inhibícia aktivity GP78 u metastatických sarkómov, by mohla viesť k inhibícii metastázovania sarkómov do pľúc.
E3 ubikvitín ligáza HUWE1 reguluje bunečnú adhéziu cez proteín TIAM1, ktorý sprostredkováva prepojenia medzi bunkami. Overexpresia HUWE1 spôsobuje aberantnú ubikvitináciu a degradáciu proteínu TIAM1, čo zvyšuje invazivitu a metastatický potenciál buniek [21].
Carboxyl terminus of Hsc70-interacting protein (CHIP)
Proteín CHIP spája chaperónový systém s ubikvitín-proteazómovým systémom. Skladá sa z troch tetratricopeptidových domén s chaperónovou funkciou, coiled-coil domény a z U-box domény s E3 ubikvitín ligázovou aktivitou (obr. 1). U-box interaguje s UbcH4/ UbcH5 proteínmi s E2 ligázovou aktivitou, tým je umožnená ubikvitinácia nesprávne zložených proteínov [22].
1. Funkčné domény proteínu CHIP. Proteín CHIP sa skladá z troch TPR domén, z coiled-coil domény a z U-box domény. TPR domény majú chaperónovú funkciu a U-box doména má E3 ubikvitín ligázovú aktivitu. Proteín CHIP spája E3 ubikvitín ligázovú aktivitu s chaperónou aktivitou. 
TPR – tetratricopeptidová doména CHIP je zapojený do procesov charakteristických abnormálnou produkciou proteínov, ktoré môžu byť súčasťou onkogénnych signalizačných dráh. CHIP má duálnu rolu a môže vystupovať ako tumor supresor alebo ako onkogén. Tumor supresorová funkcia proteínu CHIP bola objavená u mnohých nádorov, kde ubikvitinácia onkogénov ako napr. c-myc, SRC-3, HIF-1α, ERBB2, EGFR, a NF-κB vedie k ich down-regulácii [23 – 28]. In vivo a in vitro štúdiami karcinómu pankreasu bolo dokázané, že zvýšenie hladiny proteínu CHIP vedie k inhibícii metastázovania a rastu nádoru, zatiaľ čo knock-down proteínu CHIP spôsobuje metastatický zvrat. Ďalej bolo dokázané, že CHIP ubikvitináciou znižuje hladinu EGFR, čím inhibuje aberantné dráhy zapojené v malígnych procesoch [27]. U neskorých štádií kolorektálneho karcinómu je promótor proteínu CHIP hypermetylovaný, čo prispieva k jeho významnej downregulácii a zvýšenej aktivite NF-κB signalizácie [28]. Ďalej CHIP v úlohe tumor supresoru môže inhibovať IL8 indukovanú angiogenézu prostredníctvom ubikvitinácie NF-κB/ p65 [29].
Na druhej strane viaceré publikácie popisujú zapojenie proteínu CHIP v degradácii tumor supresorových proteínov. CHIP teda možno považovať aj za onkogén. Tumor supresorový proteín FOXO indukovaný proteínom TNFα podlieha degradácii ubikvitináciou sprostredkovanou proteínom CHIP [30]. CHIP je zapojený aj do regulácie tumor supresorov spojených s apoptózou, medzi ktoré patrí hlavne p53, AIF a IRF-1 [31 – 33]. Úloha proteínu CHIP v onkogénnej signalizácii je komplikovaná a závisí na mnohých aspektoch. CHIP má vplyv na rôzne substráty, ale ich preferencia nie je dostatočne popísaná. Analýza ubikvitinácie proteínu CHIP za rôznych podmienok môže objasniť mechanizmy jeho účinku vplývajúce na stabilitu proteínov zapojených v onkogénnych signalizačných dráhach.
Analýza ubikvitinácie hmotnostnou spektrometriou
V súčasnej dobe nachádzajú vo výskume ubikvitinácie veľmi dobré uplatnenie metódy hmotnostnej spektrometrie a imunochemické metódy, ktoré možno spolu kombinovať [34,35]. Hmotnostne spektrometrická analýza vychádza z predpokladu, že trypsín počas proteomickej prípravy vzorky neštiepi proteínovú sekvenciu v pozícii lyzínu modifikovaného ubikvitínom. V peptidovom digeste teda získavame tryptické peptidy s vynechaným štiepnym miestom za lyzínmi nesúcimi GlyGly pochádzajúcim z ubikvitínu. Metódou LC-MS/ MS možno určiť pozíciu ubikvitinácie v reťazci peptidu/ proteínu a z výsledku možno predpokladať aj jej biologickú úlohu [36].
Identifikácia ubikvitínovaných peptidov/ proteínov je však pomerne komplikovaná kvôli ich nízkemu zastúpeniu vo vzorke. Dôvodom je rýchla degradácia ubikvitínovaných proteínov a nízka stechiometria modifikácie. Robustná platforma na LC-MS/ MS identifikáciu endogénnej ubikvitinácie by mala obsahovať techniku špecificky obohacujúcu vzorku o peptidy nesúce GlyGly zbytok po proteolytickej digescii. Úspešnosť identifikácie ubikvitínačných miest sa všeobecne zlepšila po komercionalizácii protilátok špecifických proti GlyGly zbytkom na lyzínoch. Imunochemickým obohatením vzorky o GlyGly modifikované peptidy možno izolovať a identifikovať ubikvitínačné miesta v biologických vzorkách ako napr. bunečný lyzát alebo tkanivový lyzát [37,38]. Alternatívne možno použiť protilátky špecifické proti intaktnému ubikvitínu alebo ubikvitín značený afinitnými značkami ako napr. hemagglutinín (HA), polyhistidín (His6) alebo glutathion S transferáza (GST) [39 – 42]. Týmito spôsobmi možno obohatiť vzorku o ubikvitínované proteíny ešte pred zahájením proteolytického štiepenia [39].
K dokonalejšiemu popisu úlohy ubikvitinácie môže prispieť aj rozvoj hmotnostne spektrometrických metód umožňujúcich kvantifikáciu proteínov. Dá sa predpokladať, že kvantitatívne metódy hmotnostnej spektrometrie môžu určiť kvantitu ubikvitínovaného proteínu/ peptidu, čo by umožnilo dynamicky sledovať vývoj zmeny modifikácie v čase, príp. medzi dvoma stavmi (tumor vs. zdravá kontrola). Týmto spôsobom možno dynamicky popísať molekulárnu príčinu onkologického ochorenia, príp. stratifikovať onkologických pacientov na základe molekulárnej príčiny ochorenia.
Materiály a metódy
Ubikvitinačná reakcia
Ubikvitinačná reakcia in vitro bola uskutočnená v roztoku zloženom z 27 mM Hepes (pH 8,0), 6,5 mM MgCl2, 0,543 mM DTT, 3,26 mM ATP (kontrolné vzorky boli pripravené bez ATP), 100 nM Ube1 (E1), 1 µM UbcH5alpha (E2), 3,2 µg CHIP s His-tagom a 2 µg ubikvitínu. Reakcia bola uskutočnená v intervaloch 5, 20 a 40 min, potom bola zastavená.
Štiepenie proteínov v roztoku
Proteíny boli 5 min sonifikované, následne bolo pridaných 2,5 µl 200 mM dithiothreitolu a zmes bola kultivovaná pri 37 °C počas 1 hod. Alkylácia proteínov bola uskutočnená prídavkom 2,5 µl 200 mM jodoacetamidu počas 30 min pri izbovej teplote v temne. Prebytočný jodoacetamid bol odstránený prídavkom 2,5 µl 200 mM dithiothreitolu pri 37 °C, 15 min. Digescia proteínov na peptidy prebehla za účasti trypsínu (Promega, USA), ktorý bol pridaný v pomere 1 : 100 (enzým : proteín). Proteolytické štiepenie prebiehalo pri 37 °C cez noc. Vzorky boli odsolené podľa Bouchala et al [43].
Parametre LC-MS/ MS merania proteínových digestov
LC-MS/ MS analýza bola uskutočnená postupom inšpirovaným Faktor et al [44]. V skratke, peptidy boli separované na kvapalinovom chromatografe Eksigent ekspert™ nanoLC 400 (SCIEX, USA), ktorý bol priamo spojený s MS systémom TripleTOF 5600+ (SCIEX, Canada). Peptidy boli zachytené a odsolené na trap kolóne µ-precolumn, 30 µm i.d., 5 mm dĺžka, C18 PepMap 100, 5 µm veľkosť častíc, 100 Å veľkosť pórov (Thermo Scientific, USA). Následne boli peptidy eluované na analytickú emiterovú kolónu 75 μm × 250 mm PicoFrit® (New Objective, USA) s náplňou ProntoSIL 120-3-C18 AQ, 3 μm častice (Bischoff, Nemecko). Mobilná fáza A bola zložená z 0,1 % (v/ v) kyseliny mravčej v H2O a mobilná fáza B z 0,1 % (v/ v) kyseliny mravčej v acetonitrile. Elúcia z analytickej kolóny začínala na 5 % mobilnej fázy B počas 30 min. Percentuálny podiel mobilnej fázy lineárne stúpal na 40 % B počas 120 min a v nasledujúcich 5 min vzrástol až na 80 % B, na hodnote 80 % B zostal počas 5 min následne podiel mobilnej fázy B lineárne klesal na 5 % počas 2 min. Podiel mobilnej fázy B zostal na hodnote 2 % počas ďalších 28 min.
Hmotnostný spektrometer TripleTOF 5600+ pracoval v dáta dependentnom móde. TOF-MS spektrum (akumulačný čas 250 ms) bolo zmerané v každom cykle nasledované fragmentáciou 20 najintenzívnejších prekurzorových iónov a ukončené zmeraním spektier ich produktových iónov. Minimálna intenzita prekurzorového iónu bola nastavená na hodnotu 50 cps (counts per second) a čas exklúzie na hodnotu 12 s. Akumulačný čas prekurzorového iónu bol 100 ms.
Analýza LC-MS/ MS dát
Analýza LC-MS/ MS dát prebehla v programe ProteinPilot 4.5 (SCIEX, Canada). Parametre prehľadávania boli nastavené nasledovne: proteáza – trypsín, fixné modifikácie – karbamidomethyl na cysteíne, prehľadávanie s dôrazom na identifikáciu ubikvitinácie, prehľadávacia databáza – Uniprot+Swissprot (ver. 2) Homo sapiens. Analýza FDR (false discovery rate) prebehla proti obrátenej databáze.
Výsledky analýzy LC-MS/ MS dát
Analýzou LC-MS/ MS dát bolo identifikovaných 142 unikátnych peptidov pokrývajúcich sekvenciu proteínu CHIP z 99,7 %. Obr. 2A znázorňuje fragmentačné spektrum jedného z ubikvitínovaných peptidov SPSAQELK[UGG]EQGNR, ktorý sa podarilo identifikovať v reakčnej zmesi proteínu CHIP s ATP po inkubácii ubikvitínačnej reakcie počas 20 min. Peptid nesie charakteristické vynechané štiepne miesto za lyzínom modifikovaným ubikvitínom. Na lyzíne je identifikovaný charakteristický GlyGly zbytok na Lys8 vzniknutý tryptickým štiepením ubikvitínu. Tento peptid nie je však jediný z identifikovaných ubikvitínovaných peptidov. LC-MS/ MS analýza odhalila ubikvitináciu aj na ďalších lyzínoch a dá sa predpokladať, že niektoré identifikácie budú falošne pozitívne (obr. 2B). K filtrácii potenciálne pozitívne falošných priradení GlyGly modifikácie významne prispelo zaradenie validačného kroku do nášho postupu identifikácie ubikvitinácií. Validačný krok spočíva v kontrole fragmentačných spektier modifikovaných peptidov. Pri validácii spektier sa zameriavame na počet produktových iónov so zbytkom GlyGly (+114 Da) na lyzíne. Ukázalo sa, že niektoré peptidy identifikované s vysokou hodnotou peptidovej konfidencie (pep. konfidencia > 99 %) nesúce GlyGly zbytok obsahujú vo fragmentačnom spektre len niekoľko produktových iónov modifikovaných GlyGly zbytkom na lyzíne. Ich prekurzorový peptid môže byť izobarický s peptidmi s inou sekvenciou. Dobrým príkladom je izobarický peptid, ktorý miesto GlyGly modifikácie má aminokyseliny GlyGly zaradené priamo v peptidovej sekvencii za alebo pred lyzínom. Existencia ubikvitinácie na peptidoch s nízkym počtom produktových iónov potvrdzujúcich GlyGly modifikáciu nemusí byť reálna a ich identifikácia je teda pravdepodobne falošne pozitívna. Ako reprezentatívny príklad uvádzame dva peptidy s rovnakou peptidovou konfidenciou (pep. konfidencia = 99 %) nesúce GlyGly zbytky na lyzíne (obr. 3A, 3B). V obr. 3A a 3B sú u oboch peptidov (SPSAQELK[UGG]EQGNR a ALC[CAM]YLK[UGG]MQQHEQALADC[CAM]R) zelenou farbou vyznačené m/ z spoľahlivo identifikovaných produktových iónov z MS/ MS spektier a zároveň v obr. 2A a 2B sú tieto produktové ióny zvýraznené priamo v MS/ MS spektre. Z obr. 2B a 3B vyplýva, že GlyGly zbytok na Lys6 v peptide ALC[CAM]YLK[UGG]MQQHEQALADC[CAM]R potvrdzuje len jeden produktový ión (obr. 2B, 3B), zatiaľčo pri peptide SPSAQELK[UGG]EQGNR (obr. 2A, 3A) zaznamenávame až 21 produktových iónov s charakteristickým posunom o 114 Da (GlyGly zbytok) potvrdzujúcim ubikvitináciu na Lys8. Z obr. 2A, 2B a 3A, 3B je zrejmé, že pravdepodobnosť správnej identifikácie GlyGly zbytku lyzínu je odlišná u oboch peptidov a nesúvisí s peptidovou konfidenciou. Z tohto dôvodu doporučujeme pri identifikácii ubikvitinácie dôkladne prekontrolovať fragmentačné spektrá peptidov.
2. Fragmentačné spektrum peptidu SPSAQELK[UGG]EQGNR (A) a fragmentačné spektrum peptidu ALC[CAM]YLK[UGG]MQQHEQALADC[ CAM]R (B) so zvýraznenými produktovými iónami potvrdzujúcimi prítomnosť GlyGly modifi kácie na lyzíne. Fragmentačné spektrum A naznačuje, že Lys8 nesie GlyGly zbytok ubikvitínu. V produktových iónoch y6–y12 dochádza k charakteristickému posunu m/z o +114 Da (GlyGly zbytok). Rovnaký posun m/z možno sledovať aj v b sérii produktových iónov (b8–b12) indikujúci prítomnosť GlyGly zbytku ubikvitínu. Fragmentačné spektrum B obsahuje len jeden produktový ión potvrdzujúci modifi káciu GlyGly. Ubikvitinácia peptidu ALC[CAM]YLK[UGG]MQQHEQALADC[CAM]R nebola potvrdená, keďže jediný charakteristický produktový ión s posunom +114 Da môže pochádzať z iného kofragmentovaného prekurzorového iónu. ![Fragmentačné spektrum peptidu SPSAQELK[UGG]EQGNR (A) a fragmentačné spektrum peptidu ALC[CAM]YLK[UGG]MQQHEQALADC[
CAM]R (B) so zvýraznenými produktovými iónami potvrdzujúcimi prítomnosť GlyGly modifi kácie na lyzíne. Fragmentačné
spektrum A naznačuje, že Lys8 nesie GlyGly zbytok ubikvitínu. V produktových iónoch y6–y12 dochádza k charakteristickému posunu
m/z o +114 Da (GlyGly zbytok). Rovnaký posun m/z možno sledovať aj v b sérii produktových iónov (b8–b12) indikujúci prítomnosť
GlyGly zbytku ubikvitínu. Fragmentačné spektrum B obsahuje len jeden produktový ión potvrdzujúci modifi káciu GlyGly.
Ubikvitinácia peptidu ALC[CAM]YLK[UGG]MQQHEQALADC[CAM]R nebola potvrdená, keďže jediný charakteristický produktový ión
s posunom +114 Da môže pochádzať z iného kofragmentovaného prekurzorového iónu.](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image_pdf/246588c11007f5c6701a9c1f38844fad.jpeg)
MS/MS – tandemová hmotnostná spektrometria, CPS – counts per second 3. Zoznamy produktových iónov reprezentujúcich dva vybrané peptidy z proteínu CHIP nesúce GlyGly zbytok. Na základe identifi kácie prehľadávacím algoritmom ProteinPilot 4.5.0.0. bola obom peptidom pridelená vysoká peptidová konfi dencia (pep. konfi dencia > 99 %). Zelenou farbou sú vyznačené produktové ióny spoľahlivo identifi kované v fragmentačnom spektre tandemovej hmotnostnej spektrometrie. Červenou farbou sú podčiarknuté iba produktové ióny, ktoré potvrdzujú GlyGly zbytok v peptide a zahrňujú charakteristický posun m/z o 114 Da (zbytok GlyGly). 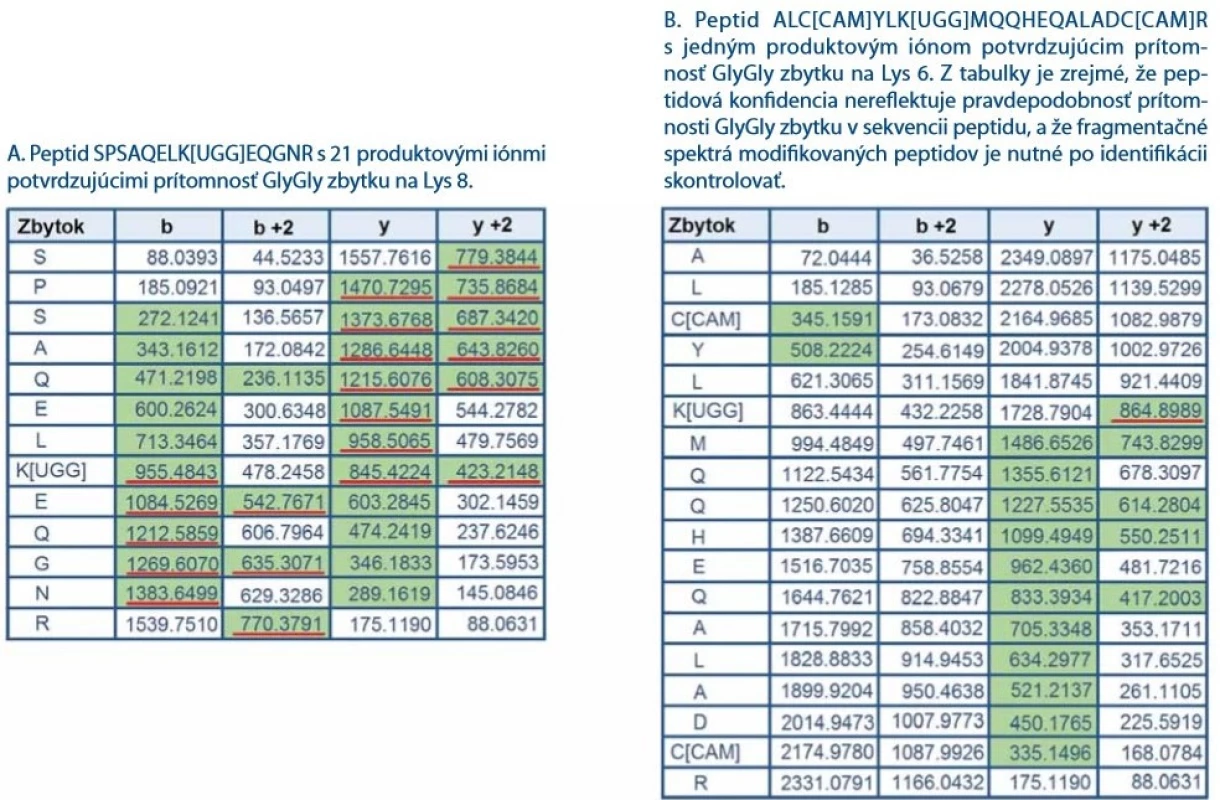
Overenie funkčnosti postupu identifikácie ubikvitinácie sme skúmali aj postupom vyplývajúcim z ATP-dependentnej podstaty reakcie. Množstvo ubikvitínovaných lyzínov sme sledovali v prítomnosti a v neprítomnosti ATP v reakčnej zmesi. Výsledky LC-MS/ MS analýz v oboch podmienkach sú zhrnuté v obr. 4A a 4B. Dáta jasne poukazujú na to, že dochádza k nárastu ubikvitinácie lyzínov po pridaní ATP do reakčnej zmesi. Pokračovali sme výskumom dynamiky priebehu ubikvitínačnej reakcie v prítomnosti ATP v intervaloch 5, 20 a 40 min od započatia ubikvitínačnej reakcie. Z obr. 5A – C je jasný postupný nárast ubikvitinácie lyzínov s časom.
4. Na obrázku je červenou farbou zvýraznená ubikvitinácia lyzínov v sekvencii CHIP-u s prídavkom (1B) a bez prídavku ATP (1A) do ubikvitínačnej reakčnej zmesi. Peptidy s vysokou konfi denciou (pep. konfi dencia > 95 %) sú znázornené zelenou farbou, peptidy so stredne vysokou konfi denciou (pep. konfi dencia ≤ 95 % a > 50 %) sú znázornené oranžovou farbou, nízko konfi dentné peptidy (pep. konfi dencia ≤ 50 %) sú znázornené červenou farbou a neidentifi kované peptidy sivou farbou písma. CHIP je jednoznačne viac ubikvitínovaný v prítomnosti ATP, zatiaľ čo v neprítomnosti ATP k ubikvitinácii nedochádza. 
ATP – adenozíntrifosfát 5. Časový priebeh ubikvitínačnej reakcie proteínu CHIP po 5 min (2A), 20 min (2B) a 45 min (2C) s prídavkom ATP do ubikvitínačnej reakčnej zmesi. Z obrázku 2A–C je zjavný nárast ubikvitinácie s postupom času. 
ATP – adenozíntrifosfát Diskusia
Literárna rešerš jasne naznačuje, že ubikvitinácia je sofistikovaným nástrojom regulácie proteostázy a má mnoho významov. Regulácia hladiny proteínu ubikvitináciou má duálny charakter a zohráva úlohu v mnohých nádorových ochoreniach. Zdá sa, že pozornosti neuniká ani možnosť cielenia liečebných postupov na ubikvitín ligázy a deubikvitínačné enzýmy. V súčasnosti už bolo objavených niekoľko molekúl zasahujúcich do procesu ubikvitinácie s cieľom zastaviť malígne procesy, no ich využitie ešte stále nie je zavedené do klinickej praxe [45]. Ukazuje sa, že E3 ubikvitín ligázy majú najväčší potenciál pre tvorbu cielených terapeutických postupov, nakoľko sa vyznačujú vysokou substrátovou špecificitou. Avšak z hľadiska zapojenia ubikvitinácie a ubikvitín ligáz v onkogenéze je stále mnoho neprebádaných malígnych procesov, ktoré unikajú pozornosti výskumníkov a lekárov. Práve zapojenie „-omics“ metód môže ponúknuť bližší náhľad na tieto procesy. Tvorba hmotnostne spektrometrickej metódy na popis ubikvitinácie nám umožnila skúmať ubikvitináciu na špecifických miestach v sekvencii proteínu. Lokalizácia pozície ubikvitinácie v sekvencii proteínu je významná pre porozumenie jej biologickej úlohe [46 – 48]. Na proteíne CHIP sa nám podarilo identifikovať ubikvitináciu (pozn. GlyGly zbytok) na viacerých lyzínoch. Autoubikvitinácia proteínu CHIP identifikovaná na Lys22 bola už v minulosti popísaná v publikácii od Graf et al [49] podobne ako aj ubikvitinácia na Lys30, Lys125, Lys221 a Lys255 v publikáciách od Lumpkin et al a Akimov et al [50,51]. Ubikvitinácia na Lys4, Lys7, Lys41, Lys223 a Lys234 nebola doposiaľ popísaná, treba však podotknúť, že ubikvitináciu v týchto pozíciách sa podarilo identifikovať vo vzorkách pripravených ubikvitínačnou reakciou in vitro. Z biologického hľadiska by bolo zaujímavé zistiť, či sa obdobná ubikvitinácia vyskytuje in vivo, a určiť jej zmysel.
Definícia pravidiel manuálnej kontroly peptidových LC-MS/ MS spektier na potvrdenie prítomnosti GlyGly zbytku bola kľúčová pre overenie správnosti identifikácie ubikvitínovaných peptidov. Peptidová konfidencia priamo súvisí s kvalitou fragmentačného spektra peptidu, avšak ukázalo sa, že prehľadávací algoritmus v tejto hodnote nezohľadňuje pravdepodobnosť správnej identifikácie GlyGly zbytku. V analýzach ubikvitínomu teda môže dochádzať k identifikácii falošne pozitívnych peptidov s vysokou konfidenciou (peptidová konfidencia > 95 %), kde sa LC-MS/ MS spektrum GlyGly modifikovaného a nemodifikovaného peptidu líši len v jednom fragmentovom ióne. Dobrým príkladom je identifikovaný peptid ALC[CAM]YLK[UGG]MQQHEQALADC[CAM]R, u ktorého nie je možné s dostatočnou určitosťou potvrdiť prítomnosť GlyGly modifikácie (+114 Da) na lyzíne. Prítomnosť modifikácie potvrdzuje len jeden fragmentový ión obr. 2B a 3B, ktorý môže pochádzať z iného ko-fragmentovaného prekurzorového iónu.
Aplikáciou pravidiel manuálnej kontroly sa nám podarilo rozlíšiť dva peptidy so zdanlivo identickou pravdepodobnosťou prítomnosti GlyGly zbytku v sekvencii. Zavedený postup odporúčame aplikovať na potvrdenie prítomnosti ľubovoľnej modifikácie peptidov alebo na odfiltrovanie falošne pozitívnych identifikácií modifikovaných peptidov. Nevýhodou je časová náročnosť postupu a teda aplikácia v komplexných peptidových vzorkách (digest tkaniva, modelových bunečných línií) je limitovaná.
Presná znalosť pozície ubikvitinácie nemusí byť dostačujúca pre výskum priebehu biologického procesu. Náhľad na dynamiku čiastočne poskytuje sledovanie vývoja ubikvitinácie v čase. Schopnosť sledovania zmeny počtu ubikvitinácií lyzínov v čase sme úspešne demonštrovali na proteíne CHIP. S predlžujúcim sa časom inkubácie ubikvitínačnej reakcie sa postupne zvyšoval počet ubikvitínovaných lyzínov. Sledovanie ubikvitinácie v čase prispelo k objavu lyzínov proteínu CHIP, kde k autoubikvitinácii dochádza prednostne.
Nevýhodou metódy je, že zo získaných dát nie je možné spoľahlivo určiť do akej miery boli ubikvitínované jednotlivé lyzíny za skúmaných podmienok. K dynamickému popisu ubikvitinácie jednotlivých lyzínov sa snažíme v súčasnosti vyvinúť semikvantitatívnu akvizičnú metódu. Vo vývoji semikvantitatívnej metódy sa snažíme aplikovať poznatky z label-free kvantitatívnej proteomiky. Pre presnejší popis dynamiky plánujeme využiť metódu SWATH (sequential windowed acquisition of all theoretical mass spectra), ktorá by teoreticky mala paralelne kvantifikovať všetky detekovateľné GlyGly modifikácie lyzínov počas jedinej LC-MS analýzy.
Dá sa predpokladať, že kvantitatívnemetódy hmotnostnej spektrometrie, medzi ktoré patrí aj metóda SWATH, môžu určiť kvantitu ubikvitínovaného proteínu/ peptidu, čo by umožnilo presnejšie sledovať zmeny modifikácie, aj medzi viacerými stavmi (napr. tumor vs. zdravá kontrola). Nemenej dôležitý je popis ubikvitinácie za rôznych podmienok, či už in vitro, alebo in vivo. Týmto spôsobom možno dynamicky popísať a objasniť príčiny zmien v hladinách proteínov, príp. určiť preferenciu substrátov ubikvitín ligáz v závislosti na biologickom stave. Detailnejšia charakterizácia proteostázy môže objasniť molekulárnu príčinu aberantnej signalizácie u niektorých nádorových ochorení.
Záver
Platforma na MS výskum ubikvitinácií bola vytvorená za zjednodušených podmienok na purifikovaných proteínoch, do budúcnosti sa však počíta s rozšírením na komplexné proteomické vzorky. Práve komplexné tkanivové vzorky obsahujú celkový náhľad na ubikvitinóm pacienta. Výskum ubikvitinácií v časových intervaloch umožňuje popísať lyzíny, u ktorých dochádza k ubikvitinácii prednostne, a lyzíny, u ktorých sa ubikvitinácia nevyskytuje, príp. je len veľmi málo pravdepodobná. Objav unikátnych ubikvitinácií môže napomôcť k objasneniu molekulárnej príčiny niektorých nádorových ochorení, čo by mohlo prispieť aj k zdokonaleniu niektorých súčasných terapeutických metód v onkológii. Popis ubikvitinácie je len veľmi malým krokom k tvorbe nových postupov špecificky cielených na molekulárnu príčinu nádorového ochorenia. Efektívne spracovanie získaných informácií bude vyžadovať zapojenie multioborového tímu a rozsiahlu validáciu.
Práca bola podporená projektom MŠMT – NPU I – LO1413 a MZ ČR – RVO (MOÚ, 00209805).
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
Mgr. Jakub Faktor, Ph.D.
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: jakub.faktor@mou.cz
Obdržané/Submitted: 14. 6. 2019
Prijaté/Accepted: 27. 8. 2019
Sources
1. Goldstein G, Scheid M, Hammerling U et al. Isolation of a polypeptide that has lymphocyte-differentiating properties and is probably represented universally in living cells. Proc Natl Acad Sci USA 1975; 72 : 11 – 15. doi: 10.1073/ pnas.72.1.11.
2. Ciechanover A, Elias S, Heller H et al. “Covalent affinity” purification of ubiquitin-activating enzyme. J Biol Chem 1982; 257(5): 2537 – 2542.
3. Ciechanover A. The 2008 Lindau Nobel laureate meeting: Aaron Ciechanover, Chemistry 2004. J Vis Exp 2009; (29): 1559. doi: 10.3791/ 1559.
4. Pickart CM. Mechanisms underlying ubiquitination. Annu Rev Biochem 2001; 70 : 503 – 533. doi: 10.1146/ annurev.biochem.70.1.503.
5. Hershko A, Ciechanover A. The ubiquitin system. Annu Rev Biochem 1998; 67 : 425 – 479. doi: 10.1146/ annurev.biochem.67.1.425.
6. Cheng Q, Cross B, Li B et al. Regulation of MDM2 E3 ligase activity by phosphorylation after DNA damage. Mol Cell Biol 2011; 31(24): 4951 – 4963. doi: 10.1128/ MCB.05553-11.
7. Herhaus L, Dikic I. Expanding the ubiquitin code through post-translational modification. EMBO Rep 2015; 16(9): 1071 – 1083. doi: 10.15252/ embr.201540891.
8. Komander D, Rape M. The ubiquitin code. Annu Rev Biochem 2012; 81 : 203 – 229. doi: 10.1146/ annurev-biochem-060310-170328.
9. Nguyen LK, Dobrzyński M, Fey D et al. Polyubiquitin chain assembly and organization determine the dynamics of protein activation and degradation. Front Physiol 2014; 5 : 4. doi: 10.3389/ fphys.2014.00004.
10. Thrower JS, Hoffman L, Rechsteiner M et al. Recognition of the polyubiquitin proteolytic signal. EMBO J 2000; 19(1): 94 – 102. doi: 10.1093/ emboj/ 19.1.94.
11. Sehat B, Andersson S, Girnita L et al. Identification of c-Cbl as a new ligase for insulin-like growth factor-Ireceptor with distinct roles from Mdm2 in receptor ubiquitination and endocytosis. Cancer Res 2008; 68(14): 5669 – 5677. doi: 10.1158/ 0008-5472.CAN-07-6364.
12. Yang WL, Zhang X, Lin HK. Emerging role of Lys-63 ubiquitination in protein kinase and phosphatase activation and cancer development. Oncogene 2010; 29(32): 4493 – 4503. doi: 10.1038/ onc.2010.190.
13. Rahighi S, Ikeda F, Kawasaki M et al. Specific recognition of linear ubiquitin chains by NEMO is important for NF-kappaB activation. Cell 2009; 136(6): 1098 – 1109. doi: 10.1016/ j.cell.2009.03.007.
14. Fiil BK, Gyrd-Hansen M. Met1-linked ubiquitination in immune signalling. FEBS J 2014; 281 : 4337 – 4350. doi: 10.1111/ febs.12944.
15. Shi D, Grossman SR. Ubiquitin becomes ubiquitous in cancer: emerging roles of ubiquitin ligases and deubiquitinases in tumorigenesis and as therapeutic targets. Cancer Biol Ther 2010; 10(8): 737 – 747. doi: 10.4161/ cbt.10.8.13417.
16. Gallo LH, Ko J, Donoghue DJ. The importance of regulatory ubiquitination in cancer and metastasis. Cell Cycle 2017; 16(7): 634 – 648. doi: 10.1080/ 153841 01.2017.1288326.
17. Wu Z, Shen S, Zhang Z et al. Ubiquitin-conjugating enzyme complex Uev1A-Ubc13 promotes breast cancer metastasis through nuclear factor-кB mediated matrix metalloproteinase-1 gene regulation. Breast Cancer Res 2014; 16(4): 75. doi: 10.1186/ bcr3692.
18. Jung CR, Hwang KS, Yoo J et al. E2-EPF UCP targets pVHL for degradation and associates with tumor growth and metastasis. Nat Med 2006; 12(7): 809 – 816. doi: 10.1038/ nm1440.
19. Tanimoto K, Makino Y, Pereira T et al. Mechanism of regulation of the hypoxia-inducible factor-1 alpha by the von Hippel-Lindau tumor suppressor protein. EMBO J 2000; 19(16): 4298 – 4309. doi: 10.1093/ emboj/ 19.16.4298.
20. Tsai YC, Mendoza A, Mariano JM et al. The ubiquitin ligase gp78 promotes sarcoma metastasis by targeting KAI1 for degradation. Nat Med 2007; 13(12): 1504 – 1509. doi: 10.1038/ nm1686.
21. Vaughan L, Tan CT, Chapman A et al. HUWE1 ubiquitylates and degrades the RAC activator TIAM1 promoting cell-cell adhesion disassembly, migration, and invasion. Cell Rep 2015; 10(1): 88 – 102. doi: 10.1016/ j.celrep.2014.12.012.
22. Rosser MF, Washburn E, Muchowski PJ et al. Chaperone functions of the E3 ubiquitin ligase CHIP. J Biol Chem 2007; 282(31): 22267 – 22277. doi: 10.1074/ jbc.M700513200.
23. Paul I, Ahmed SF, Bhowmik A et al. The ubiquitin ligase CHIP regulates c-Myc stability and transcriptional activity. Oncogene 2013; 32(10): 1284 – 1295. doi: 10.1038/ onc.2012.144.
24. Kajiro M, Hirota R, Nakajima Y et al. The ubiquitin ligase CHIP acts as an upstream regulator of oncogenic pathways. Nat Cell Biol 2009; 11(3): 312 – 319. doi: 10.1038/ ncb1839.
25. Ferreira JV, Fôfo H, Bejarano E et al. STUB1/ CHIP is required for HIF1A degradation by chaperone-mediated autophagy. Autophagy 2013; 9(9): 1349 – 1366. doi: 10.4161/ auto.25190.
26. Xu W, Marcu M, Yuan X et al. Chaperone-dependent E3 ubiquitin ligase CHIP mediates a degradative pathway for c-ErbB2/ Neu. Proc Natl Acad Sci USA 2002; 99(20): 12847 – 12852. doi: 10.1073/ pnas.202365899.
27. Wang T, Yang J, Xu J et al. CHIP is a novel tumor suppressor in pancreatic cancer through targeting EGFR. Oncotarget 2014; 5(7): 1969 – 1986. doi: 10.18632/ oncotarget.1890.
28. Wang Y, Ren F, Wang Y et al. CHIP/ Stub1 functions as a tumor suppressor and represses NF-κB-mediated signaling in colorectal cancer. Carcinogenesis 2014; 35(5): 983 – 991. doi: 10.1093/ carcin/ bgt393.
29. Wang S, Wu X, Zhang J et al. CHIP functions as a novel suppressor of tumour angiogenesis with prognostic significance in human gastric cancer. Gut 2013; 62(4): 496 – 508. doi: 10.1136/ gutjnl-2011-301522.
30. Li F, Xie P, Fan Y et al. C terminus of Hsc70-interacting protein promotes smooth muscle cell proliferation and survival through ubiquitin-mediated degradation of FoxO1. J Biol Chem 2009; 284(30): 20090 – 20098. doi: 10.1074/ jbc.M109.017046.
31. Esser C, Scheffner M, Höhfeld J. The chaperone-associated ubiquitin ligase CHIP is able to target p53 for proteasomal degradation. J Biol Chem 2005; 280(29): 27443 – 27448. doi: 10.1074/ jbc.M501574200.
32. Oh KH, Yang SW, Park JM et al. Control of AIF-mediated cell death by antagonistic functions of CHIP ubiquitin E3 ligase and USP2 deubiquitinating enzyme. Cell Death Differ 2011; 18(8): 1326 – 1336. doi: 10.1038/ cdd.2011.3.
33. Narayan V, Pion E, Landré V et al. Docking-dependent ubiquitination of the interferon regulatory factor-1 tumor suppressor protein by the ubiquitin ligase CHIP. J Biol Chem 2011; 286(1): 607 – 619. doi: 10.1074/ jbc.M110.153122.
34. Udeshi ND, Mertins P, Svinkina T et al. Large-scale identification of ubiquitination sites by mass spectrometry. Nat Protoc 2013; 8(10): 1950 – 1960. doi: 10.1038/ nprot.2013.120.
35. van der Wal L, Bezstarosti K, Sap KA et al. Improvement of ubiquitylation site detection by Orbitrap mass spectrometry. J Proteomics 2018; 172 : 49 – 56. doi: 10.1016/ j.jprot.2017.10.014.
36. Xu G, Paige JS, Jaffrey SR. Global analysis of lysine ubiquitination by ubiquitin remnant immunoaffinity profiling. Nat Biotechnol 2010; 28(8): 868 – 873. doi: 10.1038/ nbt.1654.
37. Udeshi ND, Mani DR, Eisenhaure T et al. Methods for quantification of in vivo changes in protein ubiquitination following proteasome and deubiquitinase inhibition. Mol Cell Proteomics 2012; 11(5): 148 – 159. doi: 10.1074/ mcp.M111.016857.
38. Udeshi ND, Svinkina T, Mertins P et al. Refined preparation and use of anti-diglycine remnant (K-ε-GG) antibody enables routine quantification of 10,000s of ubiquitination sites in single proteomics experiments. Mol Cell Proteomics 2013; 12(3): 825 – 831. doi: 10.1074/ mcp.O112.027094.
39. Vasilescu J, Smith JC, Ethier M et al. Proteomic analysis of ubiquitinated proteins from human MCF-7 breast cancer cells by immunoaffinity purification and mass spectrometry. J Proteome Res 2005; 4(6): 2192 – 2200. doi: 10.1021/ pr050265i.
40. Ogawa Y, Ono T, Wakata Y et al. Histone variant macroH2A1.2 is mono-ubiquitinated at its histone domain. Biochem Biophys Res Commun 2005; 336(1): 204 – 209. doi: 10.1016/ j.bbrc.2005.08.046.
41. Maine GN, Gluck N, Zaidi IW et al. Bimolecular affinity purification (BAP): tandem affinity purification using two protein baits. Cold Spring Harb Protoc 2009; 2009(11): 1 – 7. doi: 10.1101/ pdb.prot5318.
42. Kirkpatrick DS, Weldon SF, Tsaprailis G et al. Proteomic identification of ubiquitinated proteins from human cells expressing His-tagged ubiquitin. Proteomics 2005; 5(8): 2104 – 2111. doi: 10.1002/ pmic.200401089.
43. Bouchal P, Roumeliotis T, Hrstka R et al. Biomarker discovery in low-grade breast cancer using isobaric stable isotope tags and two-dimensional liquid chromatography-tandem mass spectrometry (iTRAQ-2DLC-MS/ MS) based quantitative proteomic analysis. J Proteome Res 2009; 8(1): 362 – 373. doi: 10.1021/ pr800622b.
44. Faktor J, Bouchal P. Building mass spectrometry spectral libraries of human cancer cell lines. Klin Onkol 2016; 29 (Suppl 4): 54 – 58. doi: 10.14735/ amko20164S54.
45. Liu J, Shaik S, Dai X et al. Targeting the ubiquitin pathway for cancer treatment. Biochim Biophys Acta 2015; 1855(1): 50 – 60. doi: 10.1016/ j.bbcan.2014.11.005.
46. Wilkinson KD, Tashayev VL, O’Connor LB et al. Metabolism of the polyubiquitin degradation signal: structure, mechanism, and role of isopeptidase T. Biochemistry 1995; 34(44): 14535 – 14546. doi: 10.1021/ bi00044a032.
47. Deng L, Wang C, Spencer E et al. Activation of the IkappaB kinase complex by TRAF6 requires a dimeric ubiquitin-conjugating enzyme complex and a unique polyubiquitin chain. Cell 2000; 103(2): 351 – 361. doi: 10.1016/ s0092-8674(00)00126-4.
48. Sun L, Chen ZJ. The novel functions of ubiquitination in signaling. Curr Opin Cell Biol 2004; 16(2): 119 – 126. doi: 10.1016/ j.ceb.2004.02.005.
49. Graf C, Stankiewicz M, Nikolay R et al. Insights into the conformational dynamics of the E3 ubiquitin ligase CHIP in complex with chaperones and E2 enzymes. Biochemistry 2010; 49(10): 2121 – 2129. doi: 10.1021/ bi901829f.
50. Lumpkin RJ, Gu H, Zhu Y et al. Site-specific identification and quantitation of endogenous SUMO modifications under native conditions. Nat Commun 2017; 8(1): 1171 – 1182. doi: 10.1038/ s41467-017-01271-3.
51. Akimov V, Barrio-Hernandez I, Hansen SVF et al. UbiSite approach for comprehensive mapping of lysine and N-terminal ubiquitination sites. Nat Struct Mol Biol 2018; 25(7): 631 – 640. doi: 10.1038/ s41594-018-0084-y.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2019 Issue Supplementum 3-
All articles in this issue
- Uncommon EGFR Mutations in Non-Small Cell Lung Cancer and Their Impact on the Treatment
- CRISPR-Cas9 as a Tool in Cancer Therapy
- Synthetic Lethality – Its Current Application and Potential in Oncological Treatment
- Progress in the Utilisation of Organometallic Compounds in the Development of Cancer Drugs
- Overview of Current Findings about the Role of Oestrogen Receptor α in Cancer Cell Signalling Pathways
- Glycoproteins in the Sera of Oncological Patients
- Glycosylation as an Important Regulator of Antibody Function
- Protein Ubiquitination Research in Oncology
- Long Non-Coding RNAs – Current Methods of Detection and Clinical Applications
- Oncogenic Viral Protein Interactions with p53 Family Proteins
- Editorial 2019
- Cooperation of Genomic, Transcriptomics and Proteomic Methods in the Detection of Mutated Proteins
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Protein Ubiquitination Research in Oncology
- CRISPR-Cas9 as a Tool in Cancer Therapy
- Glycosylation as an Important Regulator of Antibody Function
- Uncommon EGFR Mutations in Non-Small Cell Lung Cancer and Their Impact on the Treatment
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

