-
Články
- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Klonální charakterizace kmenů Streptococcus pneumoniae metodami MLST a MLVA – může metoda MLVA charakterizaci zkvalitnit?
Clonal characterisation of Streptococcus pneumoniae strains using MLST and MLVA – Can MLVA improve the characterisation?
Aim: To determine clonal characteristics of Streptococcus pneumoniae (S. pneumoniae) strains causing invasive pneumococcal disease (IPD) in the Czech Republic (CR) in 2017. Clonal assignment of strains was performed in the National Reference Laboratory for Streptococcal Infections (NRL) by the routinely used method, multilocus sequence typing (MLST), and a newly introduced method, multiple-locus variable number tandem repeat analysis (MLVA).
Material and method: The study strains were 87 isolates of S. pneumoniae selected from those referred to the NRL within the IPD surveillance programme from all over the CR in 2017. The study set covers S. pneumoniae isolates of both pneumococcal 13-valent conjugate vaccine serotypes (1, 4, and 9V) and non-vaccine serotypes (8, 9N, and 22F) widely spread in the CR. The study methods were MLST, the standard method used worldwide for the characterisation of pneumococcal isolates based on sequencing of a set of gene regions, and MLVA, which allows to characterise isolates based on the number of tandem repeats in intergenic regions.
Results: MLST revealed and confirmed a high level of clonal homogeneity of S. pneumoniae isolates of serotypes 1, 9N, 9V, and 22F and a considerable genetic variability of serotype 4 and 8 isolates. There was a general correlation between the MLST and MLVA clonal complex assignments. In comparison with MLST, MLVA has superior clonal discriminatory power. Isolates with the newly determined MLVA profiles should be assigned to new MLVA types (MT). Nevertheless, the new web support of the MLVA scheme for S. pneumoniae is less relevant as it does not provide services comparable to those available from the web support for MLST characterisation.
Conclusions: MLST continues to be the standard method for clonal characterisation of S. pneumoniae isolates from IPD for the purposes of both national and international surveillance. MLST characteristics of isolates are helpful in the study of clonal variability conducted by both national and transnational public health protection authorities. MLVA is not routinely used but can serve as a complementary method for rapid identification of clonal relatedness between isolates, e.g. those from local outbreaks. It is more suitable for the detection of emergence and spread of a virulent clonal variant.
Keywords:
surveillance – Streptococcus pneumoniae – MLST – clonal characterisation – MLVA – serotype
Autoři: J. Kozáková
; Z. Okonji
; M. Musílek
Působiště autorů: Oddělení bakteriálních vzdušných nákaz, Centrum epidemiologie a mikrobiologie, Státní zdravotní ústav, Praha
Vyšlo v časopise: Epidemiol. Mikrobiol. Imunol. 69, 2020, č. 1, s. 20-28
Kategorie: Původní práce
Souhrn
Cíl práce: Zjištění klonálních charakteristik souboru kmenů Streptococcus pneumoniae (S. pneumoniae) působících invazivní pneumokokové onemocnění (IPO) v České republice (ČR) v roce 2017. Porovnání klonálního členění kmenů probíhalo v Národní referenční laboratoři pro streptokokové nákazy (NRL) použitím rutinně využívané metody multilokusové sekvenační typizace (Multilocus Sequence Typing, MLST) a nově zavedené metody multilokusové analýzy tandemových repetic (Multiple-Locus Variable number tandem repeat Analysis, MLVA).
Materiál a metodika: Studován byl soubor 87 kmenů S. pneumoniae, vybraný z izolátů doručených v rámci programu surveillance IPO v roce 2017 do NRL z celé ČR. Výběr byl veden přes sérotypy S. pneumoniae tak, aby soubor zahrnoval izoláty sérotypů zastoupených v pneumokokové konjugované 13valentní vakcíně (sérotypy 1, 4, 9V) a izoláty sérotypů nevakcinačních, vyskytujících se však v ČR ve značném zastoupení (sérotypy 8, 9N, 22F). Pro studium byla použita metoda MLST, která je světově rozšířeným standardem klonální charakterizace pneumokokových izolátů a využívá sekvenování souboru genových oblastí a metoda MLVA, která charakterizuje izoláty podle počtu tandemově repetitivních úseků v intergenových oblastech.
Výsledky: MLST analýza ukázala a potvrdila vysokou míru klonální homogenity izolátů S. pneumoniae sérotypů 1, 9N, 9V, 22F a značnou genetickou proměnlivost izolátů sérotypů 4 a 8. Mezi klonálním členěním, poskytnutým oběma metodami, byla rámcová korelace. V porovnání s metodou MLST poskytovala metoda MLVA detailnější klonální odlišení. U izolátů s nově zjištěnými MLVA profily je žádoucí přiřazení nového MLVA typu (MT). Tato webová podpora MLVA schématu S. pneumonie však ustupuje do pozadí a neposkytuje aktuálně všechny služby oproti webové podpoře pro MLST charakterizaci.
Závěry: Metoda MLST je a nadále zůstává u izolátů S. pneumoniae standardem při klonální charakterizaci původců IPO při provádění surveillance na úrovni místní i mezinárodní. MLST charakteristiky izolátů jsou využity při sledování klonální proměnlivosti národními i nadnárodními orgány ochrany veřejného zdraví. Metoda MLVA rutinně používána není, lze ji však použít jako doplňkovou pro zrychlené zjištění klonální příbuznosti izolátů ze situací lokálního charakteru, například ohniskových výskytů infekce. Zde může snáze zachytit vznik a šíření virulentní klonální varianty.
Klíčová slova:
Streptococcus pneumoniae – klonální charakterizace – surveillance – MLST – MLVA – sérotyp
ÚVOD
Streptococcus pneumoniae (S. pneumoniae) je klasickým příkladem vysoce invazivního grampozitivního extracelulárního bakteriálního patogenu. Patří mezi hlavní bakteriální původce, způsobující vysokou nemocnost a úmrtnost po celém světě. Pneumokoková onemocnění sahají od mírných mukózních infekcí dýchacích cest, jako je zánět středního ucha a zánět vedlejších nosních dutin, až po těžší onemocnění, jako je pneumonie, septikémie a meningitida. Podle variant polysacharidového obalu lze v současnosti určit více než 90 sérotypů této bakterie. Ačkoliv S. pneumoniae patří mezi významné lidské patogeny, běžně se nachází i na sliznici horních cest dýchacích, kde je součástí normální mikroflóry u 5–10 % zdravých dospělých a u 20–40 % dětí. Pneumokoková onemocnění a závažné, tzv. invazivní pneumokokové onemocnění (IPO) patří mezi hlavní příčiny nemocnosti a úmrtnosti na infekční onemocnění v Evropě i ve světě, přičemž nejvyšší výskyt je zaznamenán u malých dětí a starších osob.
Od roku 2008 je v České republice (ČR) zaveden program surveillance IPO, probíhající v souladu s Metodickým návodem surveillance IPO z roku 2008 a vyhláškou č. 275/2010, příloha 21. Všechny případy IPO odpovídají platné evropské i české definici případu IPO [1]: závažné onemocnění s laboratorním průkazem pneumokoka z klinického materiálu, který je za normálních podmínek sterilní.
Nejúčinnější prevencí IPO je vakcinace. Od roku 2010 je v ČR zavedeno doporučené a hrazené očkování dětí pneumokokovými konjugovanými vakcínami (PCV). Od 1. 1. 2018 je očkování vakcínou PCV13 rozšířeno i pro věkovou skupinu seniorů 65 a starších bez poplatku.
Vzhledem k variabilitě pneumokoků je důležitá kvalitní laboratorní diagnostika s detailní charakterizací. V současné době jsou, kromě klasických kultivačních metod, využívány v široké míře také metody molekulární. Prvním nezbytným krokem je u S. pneumoniae určení sérotypu, které probíhá v Národní referenční laboratoři pro streptokokové nákazy (NRL) kombinací sérologické Quellung reakce a end-point multiplexové polymerázové řetězové rekce (mPCR), která identifikuje geny kapsulárních determinant [2]. Poté následuje proces klonální charakterizace. Standardně používanou metodou je multilokusová sekvenační typizace (Multilocus Sequence Typing, MLST), založená na amplifikaci a sekvenaci oblastí souboru genů základních metabolických struktur [3]. V rámci projektu byla též zavedena a vyzkoušena metoda multilokusové analýzy proměnlivého počtu tandemových repetitivních úseků (Multiple-Locus Variable number tandem repeat Analysis, MLVA) v intergenových BOX oblastech, založená na jejich amplifikaci a následném určení počtu repetic [4].
Vybrané kmeny byly podrobeny oběma metodám a následně byla získaná data klonálního členění analyzována a porovnána včetně zhodnocení možnosti využití jednotlivých metod.
MATERIÁL A METODY
Bakteriální kmeny S. pneumoniae
Z celé ČR byl vybrán soubor 87 izolátů S. pneumoniae z IPO za rok 2017. Výběr sérotypů byl proveden tak, aby soubor zahrnoval jak izoláty zastoupené v pneumokokové konjugované 13valentní vakcíně (sérotypy 1, 4, 9V ), tak izoláty nevakcinační, vyskytující se však v ČR ve značném zastoupení (sérotypy 8, 9N, 22F) – tabulka 1.
Identifikace a sérotypizace S. pneumoniae
Kmeny S. pneumoniae jsou v NRL identifikovány podle morfologie kmene, citlivosti k optochinu, rozpustnosti ve žluči a latexové aglutinace. Typizace se provádí Quellung reakcí v kombinaci s end-point multiplex polymerázovou řetězovou reakcí (mPCR) s vyhodnocením na agarózové elektroforéze. Používané geny jsou kapsulární determinanty typické pro určitou kapsulu séroskupiny či sérotypu S. pneumoniae (geny: cpsA, wzy, cpsH, cpsI, capB, wciY, cpsK, cpsG, galU, wciP, cpsO, wci, Nbeta, wcwL, werG, wzx, wciL). Do multiplex PCR reakcí je dále zařazeno testování cpsA genu pro molekulární potvrzení identifikace S. pneumoniae bez ohledu na séroskupinu či sérotyp [2].
Extrakce DNA
Extrakce DNA z bakteriálních kultur byla provedena pomocí kolonkové extrakční soupravy QIAmp DNA Mini Kit (Qiagen, SRN) podle protokolu pro extrakci DNA gramnegativních bakterií doporučeného výrobcem.
MLST
Jako standard klonální charakterizace S. pneumoniae byla použita multilokusová sekvenační typizace (Multilocus Sequence Typing, MLST) [3]. MLST poskytuje data o alelických variantách house-keeping genů S. pneumoniae aroE (shikimate dehydrogenase), gdh (glucose-6-phosphate dehydrogenase), gki (glucose kinase), recP (transketolase), spi (signal peptidase I), xpt (xanthine phosphoribosyltransferase), ddl (D-alanine-D-alanine ligase), na jejichž podkladu se určují sekvenační typy (ST), popř. i klonální komplexy (cc). Díky sekvenačním datům lze charakterizovat izoláty z pohledu totožnosti kmenů, příbuznosti či virulence a porovnávat je na národní i na mezinárodní úrovni.
PCR amplifikace pro MLST byly provedeny v termocykleru Veriti (Thermo Fisher Scientific, USA) v 60 µl reakčních směsích, obsahujících 1,7 U polymerázy (Top-Bio, ČR), 1,2 µl směsi dNTP (10 mM, Promega, USA), po 1,5 µl primerů (10 µM, Generi-Biotech, ČR), 3 µl extraktu DNA. Amplikony byly přečistěny kitem Wizard 96 SV (Promega, USA) dle protokolu výrobce. Sekvenační amplifikace byly provedeny v 10 µl reakčních směsích (mastermix BigDye Terminator v3.1 Cycle Sequencing Kit, Thermo Fisher Scientific, USA) podle protokolu výrobce. Produkty byly přečistěny srážením 70% etanolem s 8 mM obsahem octanu draselného a po usušení suspendovány ve 12 µl formamidu. Přečistěné reakční směsi byly analyzovány v genetickém analyzátoru 3130xl (Thermo Fisher Scientific, USA), nastaveném v módu sekvenační analýzy.
K hodnocení MLST dat NRL používá software DNASTAR Lasergene 12 Core Suite a PubMLST databázi [5].
MLVA
Metoda MLVA (Multiple-Locus Variable number tandem repeat Analysis) využívá BOX oblasti v intergenových částech chromozómu S. pneumoniae obsahující skupiny repetitivních sekvencí o proměnlivém počtu. BOX oblasti jsou tvořeny segmenty BoxA, BoxB a BoxC a mohou fungovat při regulaci exprese virulenčních znaků [6, 7].Analýza amplifikovaných úseků podoblastí BoxA či BoxB elektroforézou v agarózovém gelu neumožňovala potřebnou reproducibilitu ani mezilaboratorní standardizaci [8, 9]. Standardizaci metody umožnila analýza amplifikovaných úseků analyzátory na principu kapilární elektroforézy. Metoda analyzuje tandemové repetice v segmentu BoxB o délce 45 párů bazí [10].
PCR amplifikace úseků 8 chromozomálních oblastí obsahujících analyzované tandemové repetice (BOX-1, BOX-2, BOX-3, BOX-4, BOX-6, BOX-11, BOX-12, BOX-13) byla provedena podle publikovaného protokolu [10] v termocykleru Veriti (Thermo Fisher Scientific, USA). Oproti původnímu multiplexovému protokolu byly amplifikace BOX úseků provedeny v samostatných reakčních směsích. Tento postup usnadnil následnou detekci produktů amplifikace v analyzátoru, vzhledem ke značné proměnlivosti intenzity fluorescenčního signálu amplikonů BOX úseků u různých izolátů. 20 µl reakční směsi obsahovalo 10 µl HotStar Taq Master Mix (Qiagen, SRN), 6 µl vody pro PCR, po 1 µl primerů (10 µM, Generi-Biotech, ČR), 2 µl extraktu DNA. Amplifikační forward primery byly při syntéze značeny fluorochromem FAM.
Analýza produktů amplifikace: 2 µl produktů amplifikace byly naředěny 1 : 200 vodou pro PCR. 10 µl fluorescenčního standardu GeneScan 1200 LIZ Marker (Thermo Fisher Scientific, USA) bylo naředěno 1 : 50 vodou pro PCR. Vzorky pro analýzu byly připraveny kompletací 2 µl naředěných produktů amplifikace s 10 µl naředěného standardu. Zkompletované vzorky byly zahřáty 5 min při 95 °C, ochlazeny v ledové tříšti a bezprostředně analyzovány v genetickém analyzátoru 3130xl (Thermo Fisher Scientific, USA), nastaveném v módu fragmentační analýzy odečítající souběžně pozici produktů amplifikace a fragmentů standardu.
Určení počtu tandemových repetic v amplikonech BOX oblastí bylo provedeno z výstupních hrubých dat analyzátoru v software GeneMarker 2.4 (SoftGenetics, USA). Softwarové plug-in matrice pro odečet počtu repetic u jednotlivých BOX úseků byly vytvořeny na podkladě přímé sekvenace souborů amplikonů, reprezentujících odlišné tandemově repetitivní varianty BOX oblastí.
Přiřazení MLVA typů: MLVA typ (MT) je definován jedinečnou kombinací 8 přirozených čísel, vyjadřujících počet tandemových repetic u jednotlivých BOX oblastí izolátu. Jednotlivým číselným profilům jsou přiřazeny MT charakteristiky formou souhrnného číselného označení. Schéma MT a BOX-profilů, dosud popsaných jednotlivými laboratořemi, bylo dostupné formou interaktivního webového rozhraní [11] umožňujícího uživatelské určení MT u zkoumaných izolátů. V rámci studie jsme kromě již popsaných MT zjistili 24 dosud nepopsaných BOX-profilů, v článku je označujeme „ua“ (unassigned MT).
Vizualizace dat MLST a MLVA byly provedeny v software PHYLOViZ 2.0 pomocí algoritmu goeBURST [12, 13]. Výstupem je diagram, znázorňující příbuznost kmenů na základě zpracování číselně vyjádřených profilů.
VÝSLEDKY A DISKUSE
Vybraný soubor 87 izolátů S. pneumoniae byl podroben sérotypové analýze kombinací Quellung metody a mPCR. Touto kombinací došlo k identifikaci šesti sérotypů: sérotyp 1 (5 izolátů), 4 (11 izolátů), 9V (4 izoláty), 8 (27 izolátů), 9N (12 izolátů), 22F (28 izolátů) – viz tabulka 1. U všech případů se jednalo o kmeny izolované z invazivního pneumokokového onemocnění. Epidemiologické charakteristiky ukazují z klinického hlediska na převažující pneumonie, a to převážně v kategorii starších dospělých a seniorů. Věkové rozpětí bylo 10–90 let. Zastoupení žen a mužů v souboru bylo téměř totožné (47 mužů, 40 žen). Nejčastějším klinickým materiálem záchytu S. pneumonie byla krev (85 %). Ve 12 případech skončila IPO úmrtím.
Následovalo podrobení izolátů molekulárním analýzám metodami MLST a MLVA za účelem klonální charakterizace.
Metodou MLST se soubor rozdělil podle genetické příbuznosti do různě velkých skupin 21 sekvenačních typů (obr. 1). Jeden byl doposud neznámý „ua“. Číselný profil bude teprve na tomto podkladě přidělen kurátory MLST databáze. Identifikovány byly ST: 53, 66, 156, 162, 205, 306, 404, 410, 433, 800, 801, 944, 1480, 2190, 2299, 4738, 13623, 14731, 14732, 14735. Na diagramu můžeme vidět jednotlivé ST, velikost označení je přímo úměrná počtu izolátů dané charakteristiky. Toto grafické zobrazení nám, kromě ilustrace rozčlenění souboru izolátů na jednotlivé ST, ukazuje také jejich zajímavé diskrétní shlukování podle sérotypů S. pneumoniae. Sérotypy 1, 9N a 22F se ukázaly jako zcela homogenní a každý z nich obsahoval pouze jeden ST (sérotyp 1 ST306, 9N ST66, 22F ST433). Oproti tomu se jako variabilní z pohledu ST ukázal sérotyp 4, 8 a v menší míře i 9V. Sérotyp 4 je prezentován centrálním ST801, v jehož okolí stojí příbuzné ST2190, ST14731, ST800, ST205 a ST4738. Trochu stranou je ST410, podobný i ST66 sérotypu 9N. Sérotyp 9V obsahuje dva ST156 a ST162, taktéž s mírnou vazbou na ST801 sérotypu 4. Poslední sérotyp 8 je nejvariabilnější. Důležitou roli zde hraje ST944, ze kterého se v jedné linii nalézají ST1480, ST404, ST13623, ST14732 i jeden současně „ua“ izolát a ve druhé linii ST53, ST14735, ST2299. Diagram ukazuje i souvislosti se sérotypy 9N a 1.
Obr. 1. Diagram genetické příbuznosti podle MLST analýzy, algoritmus eBURST, u souboru kmenů S. pneumoniae, ČR, 1997
Figure 1. Diagram of genetic relatedness based on MLST, eBURST algorithm for selected strains of S. pneumoniae, CR, 2017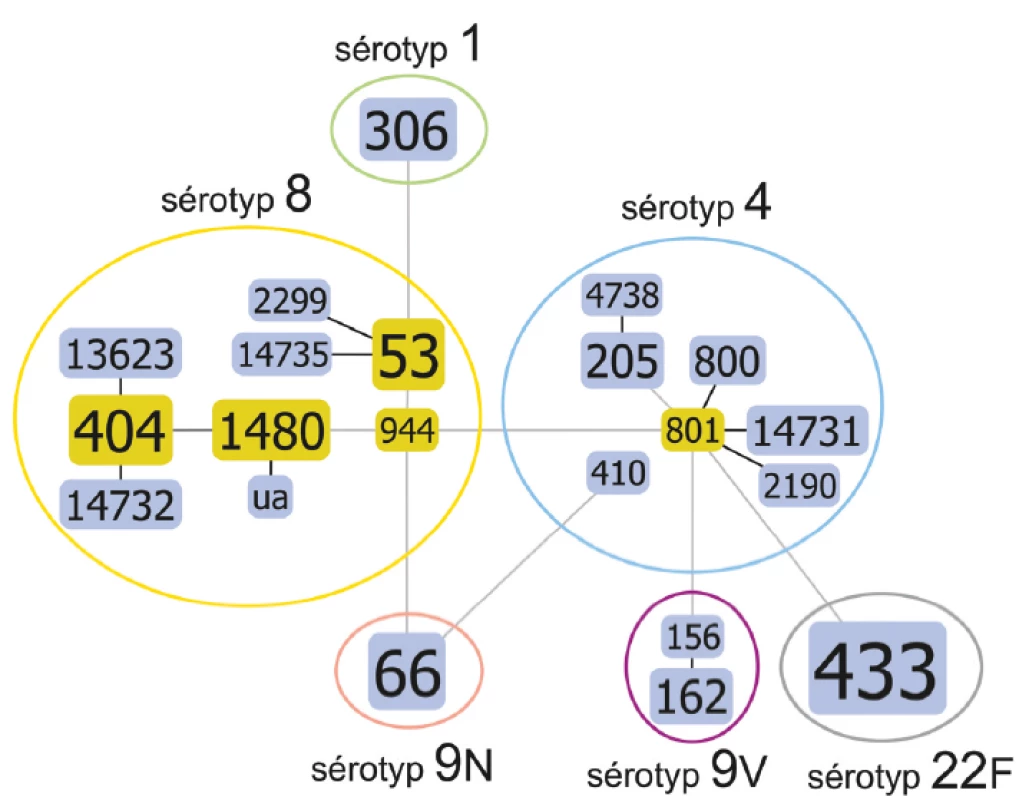
Při použití metody MLVA došlo obdobně k rozčlenění souboru na podkladě genetické příbuznosti do 42 skupin MLVA typů, které jsou rovněž zvýrazněny graficky v příbuzenském diagramu podle velikosti v přímé úměře četnosti (obr. 2). V tomto souboru však figuruje velké množství doposud neznámých MT „ua-1 - ua-24“, kterým je třeba přiřadit číselný profil kurátory MLVA databáze. Nalezeny byly MT: 3, 18, 27, 261, 263, 302, 325, 790, 792, 858, 888, 1028, 1107, 1209, 1577, 1709, 1766, 2936. Na grafickém znázornění můžeme vidět rozčlenění izolátů tentokrát metodou MLVA na jednotlivé MT, které se obdobně diskrétně shlukují podle sérotypů. V tomto případě je ovšem rozčlenění podrobnější a nevidíme u žádného ze sérotypů úplnou homogenitu MT. Při použití této metody je však kromě MT možné sledovat ještě charakteristiku označovanou jako MLVA Complex, jenž spojují příbuzné typy do komplexů. Tyto komplexy již mohou být pro sérotypy více homogenní (tab. 1). Sérotyp 1 není sice uniformní, ale zahrnuje pouze tři MLVA typy MT261, MT263 a MT1209. U nejčastějšího MT261 lze pozorovat příbuzenskou vazbu na sérotyp 22F, přes jeho dosud nepopsaný ua-11. Stejně velká je i skupina sérotypu 9V s MT790, MT792 a ua-09. Ještě zde můžeme pozorovat propojení se sérotypem 4 i 22F. Velice často byl identifikován MT302, který spolu s ua-11, ua-12, MT325, ua-10 vytváří síť sérotypu 22F. Lokalizaci tohoto sérotypu můžeme pozorovat na diagramu ve střední linii s četnými spoji na ostatní sérotypy 8, 1, 9V i 9N. Sérotyp 9N má centrální MT1766 s okolními ua-13, ua-14, MT858, MT1709 a MT1107. Sérotyp 4 je i při použití této metodiky variabilnější. Lze zde pozorovat MT1028 se skupinou doposud necharakterizovaných izolátů ua-01, ua-02, ua-03, ua-04, ua-05, ua-06, ua-07, ua-08. Z nichž má ua-08 příbuzenskou vazbu na sérotyp 9V přes izolát s MT790. Obdobně je tomu u ua-06 sérotypu 4 a ua-09 sérotypu 9V. Sérotyp 8 představoval nejpestřejší skupinu MLVA typů. Nejfrekventovanější typ v souboru izolátů kupodivu doposud nebyl popsán, jedná se o ua-18. Zásadní roli u tohoto sérotypu však hraje MT18, jenž k sobě sdružuje příbuzné MT1577, MT27, MT3 i se skupinou ua-16, ua-24, ua-19, ua-15, ua-17, ua-22, ua-21, ua-18, ua-23, ua-20, MT2936 a MT888. Opět zde lze pozorovat příbuzenské spojení s odlišným sérotypem. Tentokrát se jedná o sérotyp 8 a MT302, nejčastější MLVA typ souboru sérotypu 22F.
Obr. 2. Diagram genetické příbuznosti podle MLVA analýzy, algoritmus eBURST, u souboru kmenů S. pneumoniae, ČR, 2017
Figure 2. Diagram of genetic relatedness based on MLVA, eBURST algorithm for selected strains of S. pneumoniae, CR, 2017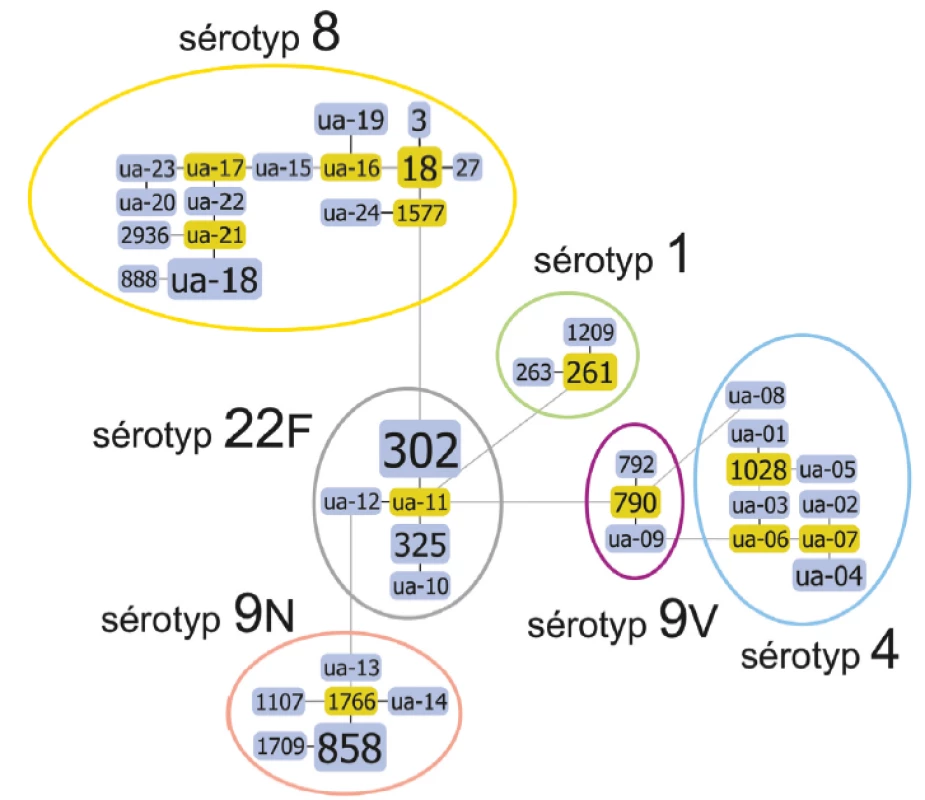
Tab. 1. Přehled epidemiologických charakteristik a výsledků MLST analýzy a MLVA analýzy u souboru kmenů S. pneumoniae, ČR, 2017
Table 1. Review of epidemiological characteristics and results of MLST analysis and MLVA analysis in S. pneumoniae strains, Czech Republic 2017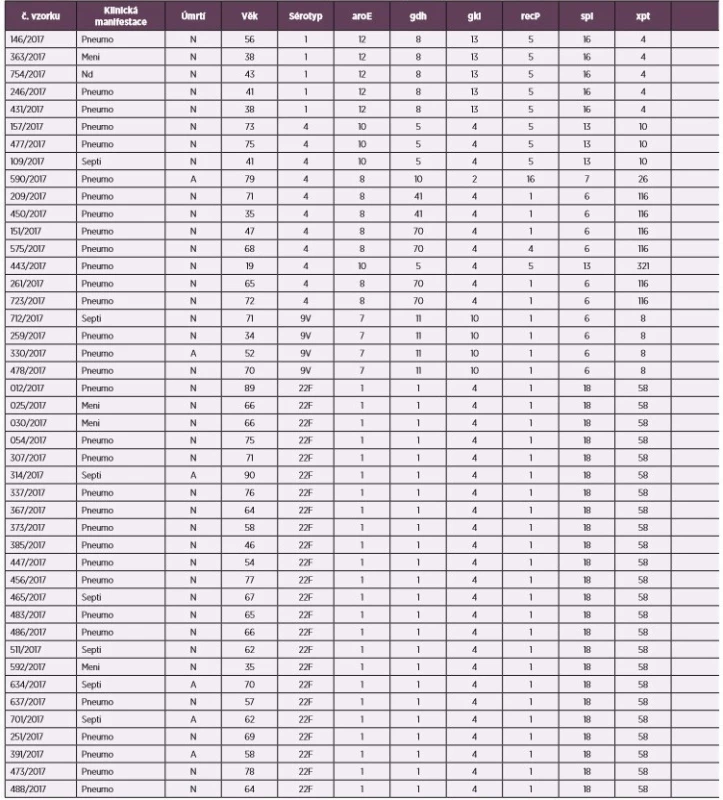
Tabulka 1. Pokračování
Table 1. Continued
Nd – nezjištěno, Meni – meningitida, Pneumo – pneumonie, Septi – sepse, N – no, A – ano, MT – MLVA typ
Nd – not identified, Meni – meningitis, Pneumo – pneumonia, Septi – sepsis, N – No, A – yes, MT – MLVA typeSrovnání výsledků MLST a MLVA ukazuje, že jen některé ST jsou představovány pouze jedním MT (obr. 3). Jedná se o ST156, ST410, ST801, ST944, ST2190, ST2299, ST4738, ST14731, ST14735, ua. Naopak u jiných ST je patrná pestrost v MT distribuci. Například u ST66 nalezneme šest rozdílných MT. Toto prezentuje zmiňovanou detailnější charakterizaci izolátů MLVA metodou. Toto srovnání ukazuje, že vztah mezi MT a ST není diskrétní a určité MT se vyskytují u více ST. Příkladem je MT18, který nalezneme u ST53, ST1480 i ST2299.
Obr. 3. Procento výskytu MLVA typů (MT) u sekvenačních typů (ST) analyzovaného souboru S. pneumoniae, ČR, 2017.
Figure 3. Distribution of MLVA types (MT) in sequence types (ST) for selected strains of S. pneumoniae, CR, 2017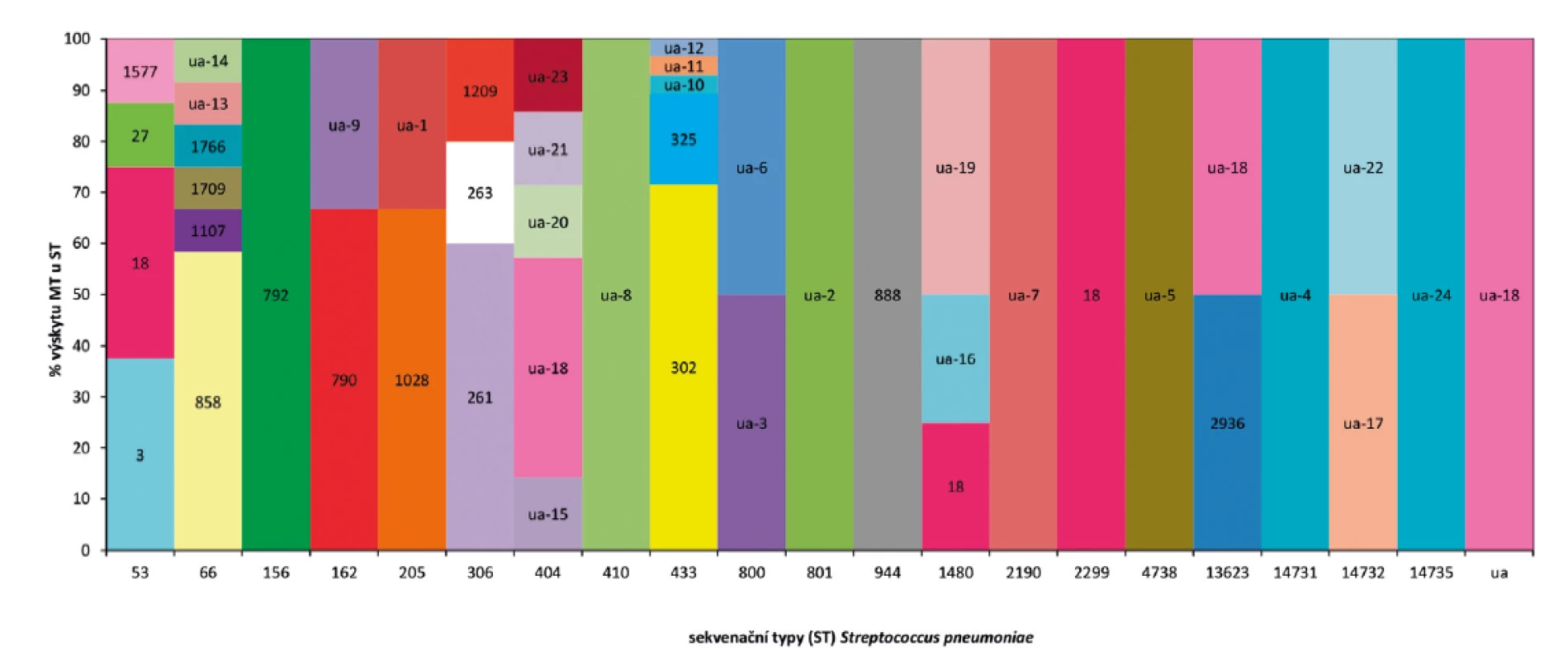
Aktuální pokrok ve využívání metody sekvenace celého genomu (WGS) působí u pneumokoků perspektivní odklon od dosavadních metod klonálního členění. Tuto metodu v současnosti NRL testuje, s možností její implementace do celorepublikového programu surveillance IPO.
ZÁVĚR
Metody MLST a MLVA popisují klonálně typovou charakteristiku izolátů S. pneumoniae sekvenčním typem (ST), resp. MLVA typem (MT), jenž jsou vztaženy k profilu alelických variant. Rozlišení klonů je při použití obou metod obdobné. Oproti sekvenační metodě MLST je provedení metody MLVA podstatně metodicky jednodušší, uživatelsky snazší a méně pracné. Získání výsledků je rychlejší i méně nákladné. Pro obě metody jsou dostupná webová rozhraní pro uživatelské určení klonální typové charakteristiky. Nevýhodou MLVA metody je však neúplná funkčnost a aktuálnost této kurátorsky spravované webové databáze. I přes dílčí výhody metody MLVA se tento přístup klonální analýzy pneumokokových izolátů nestal v referenčních laboratořích standardem, kterým je a nadále zůstává metoda MLST. Nicméně metoda MLVA může být doplňkem pro klonální porovnání příbuznosti izolátů například z lokálních epidemiologických situací. Perspektivní odklon od MLST metody může v budoucnu zřejmě způsobit až rutinní využívání metod sekvenace celého genomu.
Poděkování
Podpořeno z programového projektu Ministerstva zdravotnictví ČR s reg. č. 17-29256A. Veškerá práva podle předpisů na ochranu duševního vlastnictví jsou vyhrazena.
Do redakce došlo dne 18. 11. 2019.
MUDr. Jana Kozáková
SZÚ Praha
Šrobárova 48
100 42 Praha 10
e-mail: jana.kozakova@szu.cz
Zdroje
1. Vyhláška č. 275/2010, příloha 21 Systém epidemiologické bdělosti invazivních pneumokokových onemocnění.
2. Vacková Z, Klímová M, Kozáková J. Nová metoda a schéma typizace Streptococcus pneumoniae. Epidemiol Mikrobiol Imunol, 2013;62(2):10–18.
3. Enright MC, Spratt BG. A multilocus sequence typing scheme for Streptococcus pneumoniae: identification of clones associated with serious invasive disease. Microbiology, 1998;144(11):3049–3060.
4. Kozáková J, Musílek M, Okonji Z. Charakterizace kmenů Streptococcus pneumoniae metodou MLVA (Multiple-Locus Variable number tandem repeat Analysis) v NRL pro streptokokové nákazy SZÚ. Zprávy CEM (SZÚ, Praha), 2018;27(11–12):284–286.
5. https//pubmlst.org/spneumoniae/
6. Martin B, Humbert O, Camara M, et al. A highly conserved repeated DNA element located in the chromosome of Streptococcus pneumoniae. Nucleic Acids Res, 1992;20(13):3479–3483.
7. Knutsen E, Johnsborg O, Quentin Y, et al. BOX elements modulate gene expression in Streptococcus pneumoniae: impact on the fine-tuning of competence development. J Bacteriol, 2006;188 : 8307–8312.
8. van Belkum A, Sluijter M, de Groot R, et al. Novel BOX repeat assay for high-resolution typing of Streptococcus pneumoniae strains. J Clin Microbiol, 1996;34(5):1176–1179.
9. Koeck J-L, Njanpop-Lafourcade B-M, Cade S, et al. Evaluation and selection of tandem repeat loci for Streptococcus pneumoniae MLVA strain typing. BMC Microbiol, 2005;5 : 66.
10. Elberse KEM, Nunes S, Sá-Leáo R, et al. Multiple-Locus Variable Number Tandem Repeat Analysis for Streptococcus pneumoniae: Comparison with PFGE and MLST. PLoS ONE, 2011;6(5):e19668.
11. https://mlva.net/
12. Francicso AP, Bugalho M, Ramirez M, Carrico JA. Global optimal eBURST analysis of multilocus typing data using a graphic matroid approach. BMC Bioinformatics, 2009;10 : 152.
13. Francisco AP, Vaz C, Monteiro PT, et al. PHYLOViZ: phylogenetic inference and data visualization for sequence based typing methods. BMC Bioinformatics, 2012;13 : 87.
Štítky
Hygiena a epidemiologie Infekční lékařství Mikrobiologie
Článek vyšel v časopiseEpidemiologie, mikrobiologie, imunologie
Nejčtenější tento týden
2020 Číslo 1- Stillova choroba: vzácné a závažné systémové onemocnění
- Perorální antivirotika jako vysoce efektivní nástroj prevence hospitalizací kvůli COVID-19 − otázky a odpovědi pro praxi
- Diagnostika virových hepatitid v kostce – zorientujte se (nejen) v sérologii
- Jak souvisí postcovidový syndrom s poškozením mozku?
-
Všechny články tohoto čísla
- Možnosti terapeutického ovlivnění stafylokokových infekcí prostřednictvím bakteriofágů a vybrané metody testování citlivosti stafylokoků in vitro
- Klonální charakterizace kmenů Streptococcus pneumoniae metodami MLST a MLVA – může metoda MLVA charakterizaci zkvalitnit?
- Využití nízkoteplotního plazmatu v mikrobiologii a medicíně
- Tvorba biofilmu močovými patogénmi izolovanými z chronických a rekurentných infekcií močových ciest a vplyv biofilmu na in vitro účinok gentamicínu a kolistínu
- Preexpoziční profylaxe jako novinka v prevenci HIV infekce: zkušenosti HIV centra Ústřední vojenské nemocnice Praha
- Biologická agens bioterorismu – připravenost je nezbytná
- První potvrzený záchyt kmene Staphylococcus argenteus v České republice
- Vzpomínka na MUDr. Martinu Havlíčkovou, CSc.
- Zemřel doc. MUDr. Zdeněk Ježek, DrSc., Rytíř českého lékařského stavu
- Epidemiologie, mikrobiologie, imunologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Možnosti terapeutického ovlivnění stafylokokových infekcí prostřednictvím bakteriofágů a vybrané metody testování citlivosti stafylokoků in vitro
- Biologická agens bioterorismu – připravenost je nezbytná
- První potvrzený záchyt kmene Staphylococcus argenteus v České republice
- Preexpoziční profylaxe jako novinka v prevenci HIV infekce: zkušenosti HIV centra Ústřední vojenské nemocnice Praha
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Autoři: MDDr. Eleonóra Ivančová, PhD., MHA
Autoři: prof. MUDr. Eva Kubala Havrdová, DrSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání





