-
Medical journals
- Career
Kooperace genomických, transkriptomických a proteomických metod v detekci mutovaných proteinů
Authors: F. Zavadil Kokáš; J. Faktor; B. Vojtěšek
Authors‘ workplace: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno
Published in: Klin Onkol 2019; 32(Supplementum 3): 78-84
Category: Review
doi: https://doi.org/10.14735/amko20193SOverview
Východiska: Současná protinádorová terapie se vyznačuje vysokou nespecifitou, a to z důvodu různorodé povahy nádorů, která významně snižuje účinnost léčby. Masivní rozvoj genomických, transkriptomických a proteomických metod v posledních desetiletích umožnil detailní charakterizaci nádorů na genomové, transkriptomové a proteomové úrovni a jejich vzájemná kombinace tak představuje potenciál, jak zvýšit efektivitu procesu detekce neopeptidů a následného navržení specifické terapie. Mezi v současné době široce používané genomické a transkriptomické metody patří zejména celogenomové, celotranskriptomové, příp. exomové sekvenování, která umožňují detekovat jednonukleotidové polymorfizmy. V případě proteomických metod, pokud je k dispozici peptidová knihovna, je možné detekovat mutované proteiny v biologickém vzorku. Nedílnou součástí kooperace těchto metod jsou softwary, které umožní interpretovat získané výsledky, jejich vizualizaci, příp. zprostředkují konverzi mezi datovými formáty často specifickými pro použitou metodu/ přístroj.
Cíl: Článek primárně popisuje bioinformatickou analýzu vzorků v rámci genomických a transkriptomických metod a jejich možné limitace a související problémy, které musí být zváženy v průběhu analýzy, zejména týkající se kvality vstupních dat. V textu je rovněž věnována pozornost problémům vycházejícím ze zarovnání sekvencí na referenční genom. Součástí publikace je popis softwaru TransPEM, který byl vytvořen za účelem konverze výsledků analýzy jednonukleotidových polymorfizmů do podoby peptidové knihovny sekvencí využitelné k detekci neopeptidů pomocí proteomických metod. Nechybí ani stručný popis proteomických metod využívajících tuto knihovnu a představení jejich omezení.
Klíčová slova:
genomika – transkriptomika – proteomika – bioinformatika – vývoj softwaru
Úvod
Současná protinádorová terapie vychází zejména z histologického vyšetření nádoru, související analýzy symptomů pacienta a z jeho předchozí léčby. Terapie nádorů se vyznačují poměrně velkou nespecifitou a s tím související menší úspěšností a krátkou dobou přežití pacienta. Kvalitní charakteristika nádorů tedy představuje esenciální komponent pro zvolení, příp. vývoj terapie, která bude danému pacientovi tzv. ušita na míru.
Jednou z možností zkoumání nádorů představují tzv. omics metody [1], jejichž rozvoj v posledních desetiletích přinesl nové možnosti pro vývoj dokonalejší terapie. Z hlediska povahy zkoumané entity rozeznáváme metody genomické, epigenomické, transkriptomické, proteomické a metabolomické (schéma 1). Jejich vzájemná kooperace může být využita pro řadu výzkumných záměrů, mezi něž bezesporu patří identifikace nových proteinových izoforem [2] nebo detekce mutovaných proteinů. Prezentovaný článek je primárně věnován genomickým, transkriptomickým a proteomickým metodám a řeší jejich možnosti a limitace v souvislosti s bioinformatickou analýzou získaných dat.
Schéma 1. Diagram zobrazující rozdělení „omics“ metod (zapsáno oranžovou barvou) a výsledků, které lze obdržet jejich prostřednictvím. 
mRNA – mediátorová RNA Genomické a transkriptomické přístupy se vyznačují určitou podobností ve vlastním provedení, jelikož oba mohou být založeny na sekvenování nukleových kyselin. Široce používanými metodami jsou dnes techniky zprostředkovávající genomové sekvenování, celotranskriptomové sekvenování a příp. exomové sekvenování [3]. Z pohledu přesnosti však existuje několik důležitých faktorů, které významně ovlivňují jejich výsledek a následnou interpretaci získaných dat.
Prvním důležitým aspektem všech metod je laboratorní příprava vzorků a následné experimentální provedení sekvenace. V současném výzkumu a diagnostice je nejrozšířenější sekvenační platformou Illumina [4], která se vyznačuje vysokou přesností, přijatelnou cenou a nenáročností na laboratorní vybavení. Její nevýhodou je ovšem relativně malá délka produkovaných readů ve srovnání s dříve využívanou Sangerovou metodou [5]. Tato skutečnost může výrazně komplikovat následnou bioinformatickou analýzu, zejména pokud se jedná o celotranskriptomové sekvenování [6]. Bioinformatická analýza získaných sekvenačních dat může být rozdělena do několika kroků, jejichž specifikace je závislá na povaze experimentu. Také volba vhodného softwaru v závislosti na vstupních datech a cílech experimentu představuje zásadní faktor pro korektní bioinformatickou analýzu. Ta je rozdělena na kontrolu kvality vstupních dat, mapování readů na referenci, vlastní analýzu v závislosti na požadavcích experimentu a konečně interpretaci výsledků [7].
V případě proteomiky je v současnosti dostupná široká škála hmotnostně spektrometrických metod, které je možné rozdělit z hlediska přípravy vzorku na „top-down“ a „bottom-up“ přístupy. Metody „top-down“ zkoumají intaktní proteiny, zatímco metody „bottom-up“ analyzují peptidy získané proteomickým štěpením proteinů. „Bottom-up“ metody představují nejvýhodnější přístup pro kvalitativní i kvantitativní detekci proteinů ve vysoce komplexních biologických vzorcích, jako je nádorová tkáň, plazma nebo buněčná kultura, a proto se jim budeme dále věnovat. Tandemovou hmotnostní spektrometrii (liquid chromatography tandem-mass spectrometry – LC-MS/ MS) je možné využít pro kvalitativní analýzu, tj. identifikaci proteinů, a lze ji úspěšně kombinovat s genomickými i transkriptomickými metodami. Analýzu LC-MS/ MS dat však komplikuje dostupnost tzv. prohledávacích knihoven, které slouží jako reference pro správnou identifikaci zkoumaného proteinu. A právě genomická a transkriptomická analýza je schopna nabídnout nejkvalitnější prohledávací knihovny reprezentující zkoumaný vzorek.
Metody kvantitativní „bottom-up“ analýzy, jako např. monitorování vybraných reakcí [8], je možné kombinovat s proteogenomikou zejména za účelem validace již identifikovaných mutovaných proteinů/ peptidů. Platforma kombinující všechny popsané metody má potenciál detailně charakterizovat nádorovou tkáň a popsat proteiny/ peptidy, které doposud unikaly pozornosti (schéma 2).
Schéma 2. Diagram zobrazující kombinaci softwaru TransPEM navazujícím na bioinformatickou analýzu jednonukleotidových polymorfi zmů s proteomickou analýzou za účelem detekce mutovaných peptidů/proteinů. 
LC-MS/MS – tandemová hmotnostní spektrometrie Kvalita vstupních dat je základ
Prvním a zásadním krokem bioinformatické analýzy u sekvenace nukleových kyselin je kontrola kvality vstupních dat. Data jsou nejčastěji obdržena ve formě krátkých sekvencí, tzv. readů, přičemž kvalita každé báze v sekvenci může být odlišná [9]. U sekvenační platformy Illumina se například často setkáváme se sníženou kvalitou osekvenovaných bází na koncích readů. Za účelem posouzení kvality readů byl vyvinutý značný počet programů. Příkladem je FastQC [10], který je specifický pro platformu Illumina, nebo NGSQC (next-generation sequencing quality control) [11]. Mezi kritéria hodnocení kvality náleží zejména detekce obsahu sekvenačních adaptérů, zastoupení nukleotidů GC ve zkoumaných readech nebo sledování skóre kvality získaného ze vstupního souboru. Pokud se kvalita readů na základě těchto parametrů ukáže jako nevyhovující, je nezbytné provést opatření pro zlepšení jejich spolehlivosti, zejména ořezání readů, příp. odstranění vybraných readů. Operace ořezávání readů je široce používaná za účelem eliminace nekvalitních konců readů. K odstranění readů se přistupuje při výskytu kontaminace sekvenačními adaptéry. Obě operace mohou být samozřejmě kombinovány a v důsledku zlepšují kvalitu readů pro následné zarovnání na referenční sekvenci [2,12].
Volba softwaru pro mapování
Zarovnání readů na referenci (nejčastěji referenční genom) tvoří druhý krok v rámci bioinformatické analýzy sekvenačních dat. Volba programu určeného pro provedený typ sekvenování (exomové nebo celotranskriptomové) je dalším důležitým aspektem, který musí být zvážen zejména z důvodu odlišnosti mezi povahou dat poskytnutých sekvenováním exomů v porovnání se sekvenováním transkriptomu. Exomová sekvenování vycházejí z DNA jako vstupního materiálu, a tedy v průběhu mapování není třeba brát do úvahy alternativní sestřih primárního transkriptu RNA (pre-mRNA). Ve srovnání s celotranskriptomovým sekvenováním, v jehož případě ready obsahují sekvence transkriptu, které mohou být modifikované alternativním sestřihem, je z hlediska výpočetní náročnosti vyhodnocení exomového sekvenování snadnější [13,14].
Mapování readů na referenci je rovněž ovlivněno několika negativními faktory, které musí být při vyhodnocování brány do úvahy. Nejčastěji uváděným negativním faktorem je tzv. problém multimapování, při němž je jeden read přiřazen na několik míst v použité referenci a který nastává zejména v oblastech genomu s vysokým obsahem repetitivních sekvencí. Tento problém může být do jisté míry odstraněn použitím technologie „paired-end“ sekvenování [15,16], při níž ready, které jsou v páru, poskytují přesnější zarovnání na referenci díky v součtu delší sekvenci. Velmi často se též objevuje problém krátkých readů, které jsou obecně náchylné k falešně pozitivnímu zarovnání ve srovnání s delšími ready. Řešení tohoto problému spočívá v použití jiné technologie sekvenování produkující delší ready, což ovšem výrazně zvyšuje cenu sekvenování [14,17].
Na základě povahy dat a zohlednění alternativního sestřihu se programy pro mapování readů na referenci dělí na dvě skupiny: „unspliced read aligners“ a „spliced read aligners“. První skupina sdružuje programy, které v rámci mapování readů nezahrnují alternativní sestřih, např. BWA [18] nebo Bowtie2 [19] a jsou primárně určeny pro zpracování dat z exomového nebo genomového sekvenování. Druhá skupina programů bere do úvahy alternativní sestřih transkriptů a je vhodná pro sekvenování transkriptomu (RNA-seq). Typickými a široce používanými softwary jsou Tophat2 [20], GSNAP [21] nebo QPALMA [22].
Detekce jednonukleotidových polymorfizmů
Analýza jednonukleotidových polymorfizmů (single nucleotide polymorphism – SNP) je nezbytná pro získání peptidové prohledávací knihovny, která je nutná pro detekci potenciálních mutovaných peptidů proteomickými metodami. Hlavním problémem při detekci pravých SNP je jejich separace od artefaktů vzniklých v průběhu sekvenování, příp. v průběhu mapování readů na referenci [23]. Existuje několik volně dostupných softwarových balíčků široce používaných pro analýzu SNP, jako např. GATK [24], VarScan2 [25] nebo SAMtools [26], vzájemně se však ve schopnosti detekovat SNP liší [27]. Jejich výstupem je seznam predikovaných SNP. Na základě skutečnosti, jestli má SNP vliv na výsledný protein, rozlišujeme mutace nesynonymní (s vlivem na výslednou proteinovou sekvenci) a synonymní (bez vlivu na sekvenci aminokyselin, a to v důsledku degenerace genetického kódu). Pro případnou proteomickou analýzu jsou tedy důležité pouze mutace nesynonymní. Proteomické analýzy ovšem vyžadují SNP ve formátu peptidových sekvencí obsahujících tyto mutace, které tvoří referenční databázi nutnou pro následné prohledávání MS dat, tzv. prohledávací knihovnu. Vyhledání specifické sekvence a změna příslušné aminokyseliny mohou být u dané sekvence provedeny manuálně na základě informací získaných z analýzy SNP, nicméně s narůstajícím počtem peptidových sekvencí a za předpokladu, že jedna mutace může vést ke vzniku několika výsledných proteinů (alternativní sestřih pre-mRNA), podstatným způsobem narůstá časová náročnost těchto operací a rovněž pravděpodobnost chyby způsobené lidským faktorem.
Za účelem automatizace popsaného procesu zejména z důvodu úspory času a minimalizace pravděpodobnosti vytvoření chybné sekvence byl vytvořen software TransPEM (software for Translation sequence into PEptide based on Mutation information) [28]. Představuje nový bioinformatický nástroj pro rychlou a robustní extrakci kódujících sekvencí z genomové sekvence a jejich translace do proteinů zohledňující výsledky analýzy SNP.
Vytvořené soubory obsahující všechny takto vytvořené proteinové sekvence mohou následně posloužit proteomické analýze jako prohledávací knihovny. Pro svou činnost program vyžaduje celkem tři vstupní soubory. První ve formátu FASTA obsahující genomovou sekvenci, která musí být shodná s referencí, na kterou bylo provedeno mapování readů. Druhým je GTF soubor, který obsahuje informace o poloze genů a kódujících sekvencích v rámci genomu. Poslední soubor obsahuje výsledky z analýzy SNP poskytnuté např. programem VarScan2 [25] v tabulkovém formátu obsahující pro každé SNP chromozom, pozici v rámci chromozomu, referenční a mutantní alelu. Software TransPEM [28] poskytuje mutované a původní sekvence všech možných transkriptů daného genu. Ve srovnání s manuálním přístupem přináší významnou časovou úsporu a rovněž minimalizuje možné chyby prostřednictvím kontroly vstupních souborů a systému hodnocení a hlášení chyb vzniklých v průběhu získávání mutovaných sekvencí proteinů. Jelikož jsou generovány všechny možné peptidové sekvence pro daný gen, je rovněž navýšena pravděpodobnost detekce neopeptidů, jelikož program tímto postihuje všechny možné podoby výsledného genového produktu.
Proteomická analýza mutovaných proteinů
Tandemová hmotnostní spektrometrie nabízí informace o genové expresi na úrovni proteinů. Získané výsledky poskytují vyšší výpovědní hodnotu v porovnání s genomickými metodami, jelikož přímo popisují efektorové molekuly. Mezi největší úskalí LC-MS/ MS patří různorodá ionizovatelnost peptidů, vysoký dynamický koncentrační rozsah proteinů ve vzorku a nutnost mít k dispozici prohledávací knihovnu.
Výsledky LC-MS/ MS analýzy jsou obvykle zatíženy nedostatečným pokrytím sekvence a nízkou efektivitou přiřazení změřených spekter k teoretickým spektrům obsaženým v prohledávací knihovně. Ukázalo se, že až 10 % vysoce kvalitních a 65 % MS/ MS spekter s nižší kvalitou není vůbec přiřazeno k sekvenci proteinu/ peptidu, ze kterého pocházejí [29]. Genomické/ transkriptomické metody umožňují získat prohledávací knihovnu šitou na míru pro danou LC--MS/ MS analýzu. Přípravou těchto prohledávacích knihoven (jak bylo popsáno výše) je možné zvýšit efektivitu identifikace proteinů pomocí LC-MS/ MS, a dokonce je možné identifikovat i peptidy/ proteiny s mutovanou sekvencí.
Významně z toho benefitují LC--MS/ MS přístupy zaměřené na identifikaci tzv. neopeptidů, které pokrývají mutovanou část sekvence proteinu. Software na prohledávání LC-MS/ MS dat (např. ProteinPilot, Proteome Discoverer) v principu použije prohledávací mutantní knihovnu založenou na genomických/ transkriptomických datech, kterou in silico naštěpí proteázou a vzniklé peptidy posttranslačně modifikuje na základě pravidel definovaných uživatelem. Získaná data LC-MS/ MS analýzy jsou následně importována a zpracována. Po dobu jejich zpracování dochází k přiřazování změřených spekter ke spektrům teoretickým, a pokud je mezi spektry nalezena dostatečná shoda, je peptid považován za identifikovaný. Identifikované peptidy jsou následně přiřazeny k příslušným sekvencím proteinů. Výsledkem analýzy dat LC-MS/ MS je seznam identifikovaných proteinů a peptidů. V seznamu identifikací je potřeba vyhledat neopeptidy. V následujícím kroku je dále nutné zjistit, jestli jsou neopeptidy unikátní, a tedy jestli jejich sekvence není sdílená s jinými proteiny. K tomuto účelu je využívána funkce peptid/ protein BLAST. Za klíčovou součást nově vzniklé proteogenomické platformy považujeme zavedení verifikačního kroku, kterým ověřujeme, zda prohledávací algoritmus skutečně identifikoval neopeptid s aminokyselinovou záměnou. Problémem však zůstává, že současné prohledávací algoritmy neposkytují funkci zohledňující množství a kvalitu produktových iontů, které přímo obsahují mutované/ ou aminokyseliny/ u v jejich sekvenci. Pro lepší pochopení problému uvádíme obr. 1, která reprezentuje dva neopeptidy identifikované se stejnou peptidovou konfidencí s pomocí prohledávacího algoritmu ProteinPilot 4.5.0.0 (peptidová konfidence = 99 %). V obr. 1 jsou zeleně vyznačeny m/ z produktových iontů, které byly spolehlivě identifikovány v MS/ MS spektrech obou neopeptidů (obr. 2 a 3). Z hlediska potvrzení přítomnosti mutované aminokyseliny v sekvenci ovšem neopeptidy nejsou rovnocenné a jejich neopeptidová konfidence není identická. Je zjevné, že mutace (A na P) v neopeptidu IIPTVLMTEDIK potvrzuje jen produktové ionty z y a y 2+ série (y2, y3, y3 2+, podtrženy červenou barvou; obr. 1A), které jsou navíc velmi nespecifické vzhledem k nízkému m/ z. K opačné situaci dochází u neopeptidu VSGSPEQAVEENLSSYFLDR (S na F; obr. 1B), u něhož vidíme až osm produktových iontů ze série y a y 2+ (y-1, y 2+, podtrženy červenou barvou) potvrzující mutaci. Z obr. 1A a B je zřejmé, že pravděpodobnost správné identifikace mutací je odlišná u obou neopeptidů a nedá se ztotožňovat s peptidovou konfidencí. Z tohoto důvodu doporučujeme při identifikaci neopeptidů důkladně kontrolovat fragmentační spektra a zároveň spatřujeme potenciál v tvorbě softwaru, který by určil neopeptidovou konfidenci a zautomatizoval proces jejího určení.
Image 1. Seznam produktových iontů reprezentujících dva vybrané neopeptidy identifi kované prohledávacím algoritmem Protein- Pilot 4.5.0.0 s vysokou peptidovou konfi dencí (peptidová konfi dence > 99 %). Zelenou barvou jsou vyznačeny produktové ionty spolehlivě identifi kované v LC-MS/MS fragmentačním spektru. Červeně jsou podtrženy produktové ionty, které potvrzují aminokyselinovou záměnu a zahrnují ve své aminokyselinové sekvenci mutovanou aminokyselinu. Z obrázku je patrné, že peptidová konfi dence nerefl ektuje pravděpodobnost přítomnosti mutace v sekvenci neopeptidů a že fragmentační spektra neopeptidů je nutné po identifi kaci zkontrolovat. 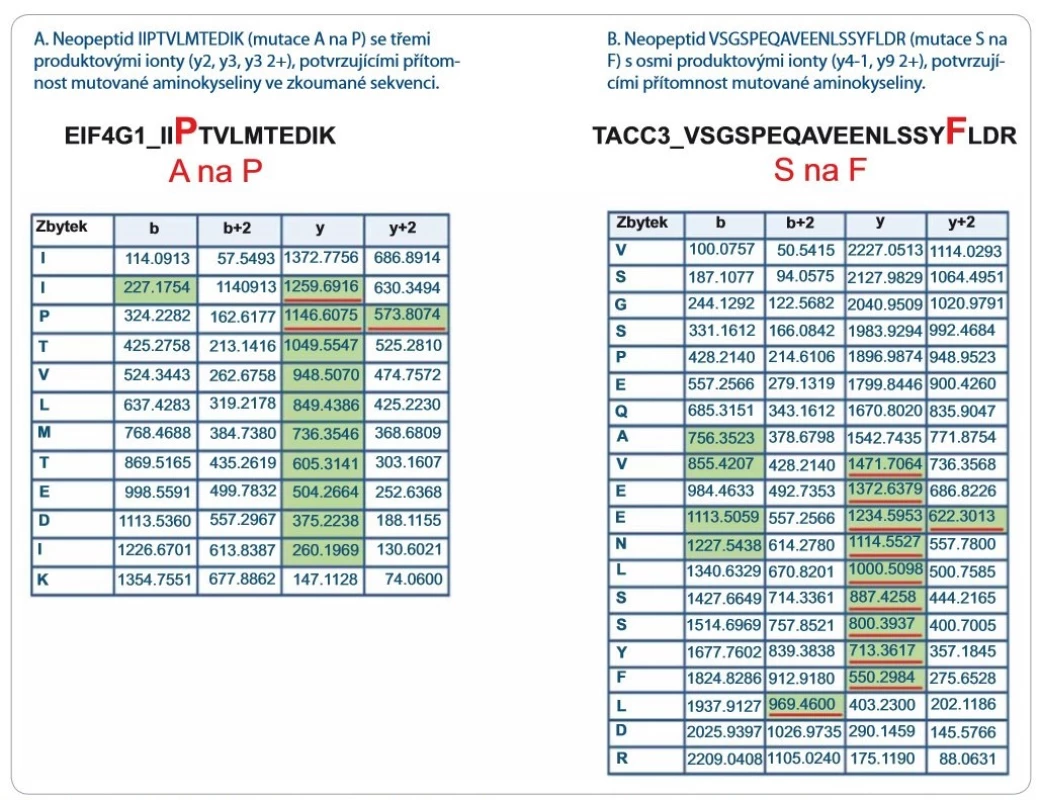
Image 2. Fragmentační spektrum produktových iontů reprezentující peptid IIPTVLMTEDIK z proteinu EIF4G1 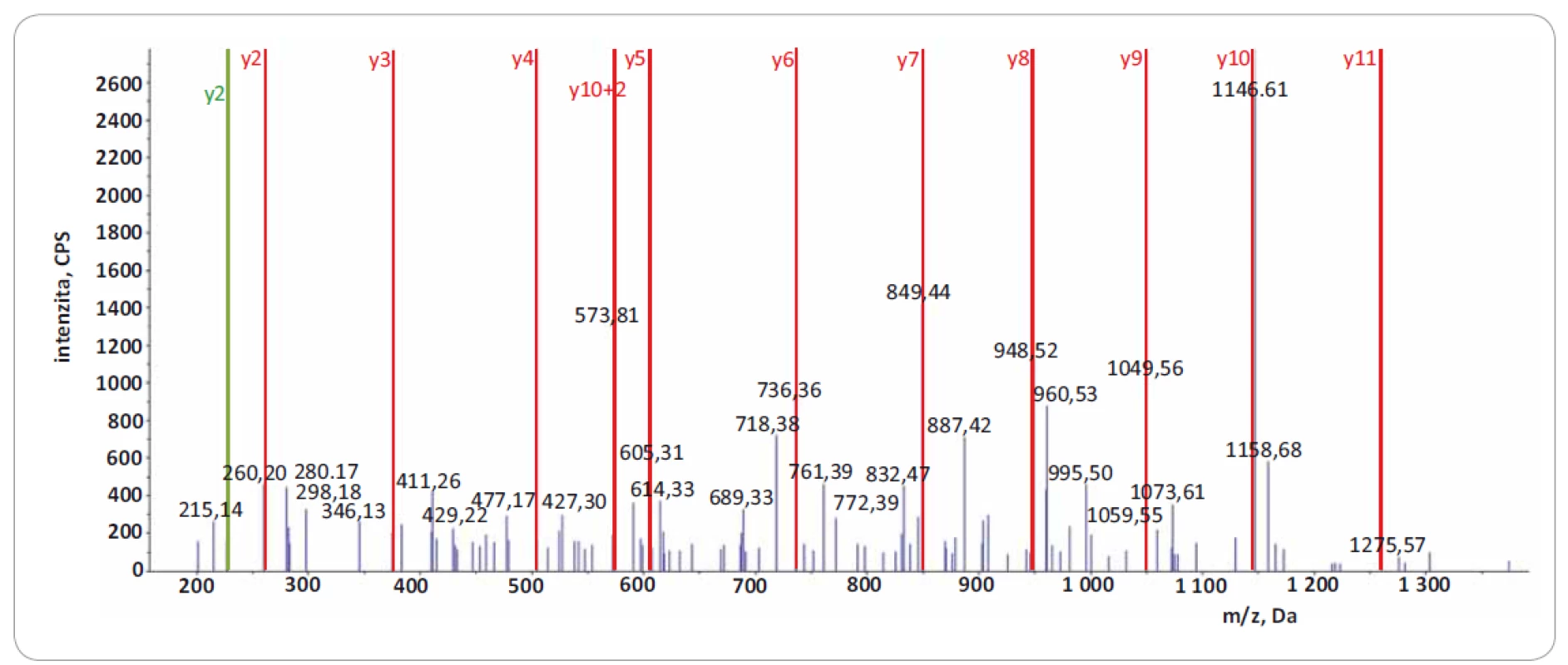
Image 3. Fragmentační spektrum produktových iontů reprezentující peptid VSGSPEQAVEENLSSYFLDR z proteinu TACC3. 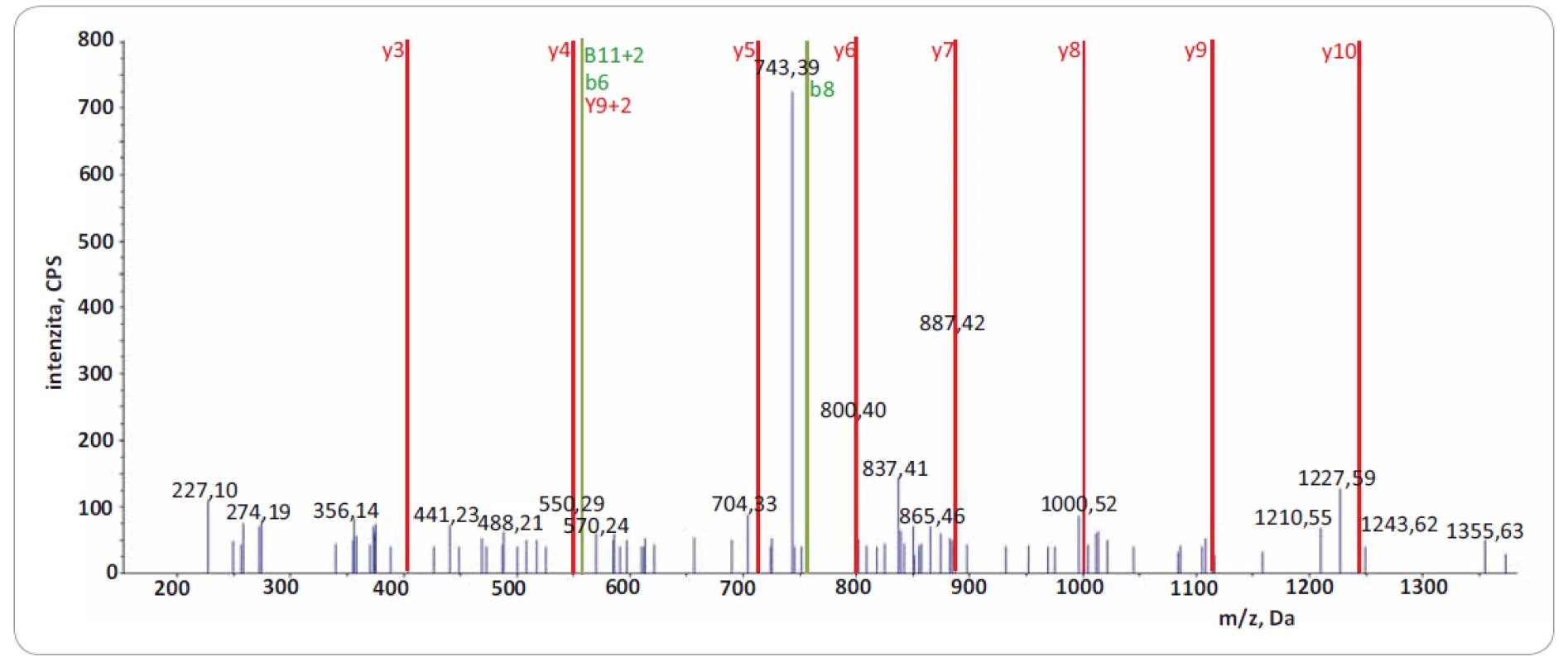
Závěr
Detailní popis nádorů zprostředkovaný vzájemnou kooperací genomických, transkriptomických a proteomických metod vede k účinnější detekci neopeptidů a potenciálně k výběru účinnější protinádorové terapie. Software TransPEM [28] zprostředkovává konverzi výsledků získaných prostřednictvím SNP analýzy do tvaru prohledávací peptidové knihovny, kterou lze následně využít proteomickými metodami k detekci neopeptidů. Software je ovládán prostřednictvím příkazové řádky a v současnosti neobsahuje grafické uživatelské rozhraní. Podrobnější popis softwaru je uveden v manuálu, který je ke stažení společně se zdrojovým kódem softwaru na webu Recamo.cz [28]. Popisovanou kooperací genomických, transkriptomických a proteomických metod lze identifikovat imunogenní neoantigeny dále využitelné v rámci přípravy protinádorových vakcín. Je ovšem nutné poukázat na skutečnost, že biologická funkce identifikovaných neoantigenů musí být dále objasněna jinými molekulárně biologickými metodami. Identifikovaný neoantigen s potenciálem pro tvorbu neoantigenových protinádorových vakcín by měl být rozpoznáván CD8 T lymfocyty. Rovněž navození efektivní imunitní odpovědi cílené na tumor specifické neoantigeny je komplikované a vyžaduje zapojení multioborového týmu vědců. Dalším důležitým aspektem je, že k navození tumor supresivního účinku neoantigenové vakcinace často dochází až po kombinaci s jinou formou imunoterapie, jako např. anti-PD1 anti-CTLA4 biologická léčba [30,31]. Úspěšná proteogenomická identifikace mutovaného peptidu, resp. neoantigenu, tedy představuje pouze velmi malý krok k vývoji specifické terapie.
Práce byla podpořena projektem MŠMT – NPU I – LO1413.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
Mgr. Bc. Filip Zavadil Kokáš, Ph.D.
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: filip.zavadil@mou.cz
Obdrženo/Submitted: 3. 6. 2019
Přijato/Accepted: 26. 8. 2019
Sources
1. Vucic EA, Thu KL, Robinson K et al. Translating cancer “omics” to improved outcomes. Genome Res 2012; 22(2): 188–195. doi: 10.1101/ gr.124354.111.
2. Conesa A, Madrigal P, Tarazona S et al. A survey of best practices for RNA-seq data analysis. Genome Biol 2016; 17 : 13. doi: 10.1186/ s13059-016-0881-8.
3. Morey M, Fernández-Marmiesse A, Castiñeiras D et al. A glimpse into past, present, and future DNA sequencing. Mol Genet Metab 2013; 110(1–2): 3–24. doi: 10.1016/ j.ymgme.2013.04.024.
4. Liu L, Li Y, Li S et al. Comparison of next-generation sequencing systems. J Biomed Biotechnol 2012; 2012 : 251364. doi: 10.1155/ 2012/ 251364.
5. Sanger F, Air GM, Barrell BG et al. Nucleotide sequence of bacteriophage φX174 DNA. Nature 1977; 265(5596): 687–695. doi: 10.1038/ 265687a0.
6. Heather JM, Chain B. The sequence of sequencers: the history of sequencing DNA. Genomics 2016; 107(1): 1–8. doi: 10.1016/ j.ygeno.2015.11.003.
7. Wolf JBW. Principles of transcriptome analysis and gene expression quantification: an RNA-seq tutorial. Mol Ecol Resour 2013; 13(4): 559–572. doi: 10.1111/ 1755-0998.12109.
8. Faktor J, Michalova E, Bouchal P. p-SRM, SWATH a HRM – cílené proteomické přístupy na hmotnostním spektrometru TripleTOF 5600+ a jejich aplikace v onkologickém výzkumu. Klin Onkol 2014; 27 (Suppl 1): 110–115. doi: 10.14735/ amko20141S110.
9. Ewing B, Green P. Base-calling of automated sequencer traces using phred. II. Error probabilities. Genome Res 1998; 8(3): 186–194.
10. Babraham Bioinformatics. FastQC: a quality control tool for high throughput sequence data. [online]. Available from: http:/ / www.bioinformatics.babraham.ac.uk/ projects/ fastqc.
11. Dai M, Thompson RC, Maher C et al. NGSQC: cross-platform quality analysis pipeline for deep sequencing data. BMC Genomics 2010; 11 (Suppl 4): S7. doi: 10.1186/ 1471-2164-11-S4-S7.
12. Yang IS, Kim S. Analysis of whole transcriptome sequencing data: workflow and software. Genomics Inform 2015; 13(4): 119–125. doi: 10.5808/ GI.2015.13.4.119.
13. Garber M, Grabherr MG, Guttman M et al. Computational methods for transcriptome annotation and quantification using RNA-seq. Nature Methods 2011; 8(6): 469–477. doi: 10.1038/ nmeth.1613.
14. Martin JA, Wang Z. Next-generation transcriptome assembly. Nat Rev Genet 2011; 12(10): 671–682. doi: 10.1038/ nrg3068.
15. Campbell PJ, Stephens PJ, Pleasance ED et al. Identification of somatically acquired rearrangements in cancer using genome-wide massively parallel paired-end sequencing. Nature Genet 2008; 40(6): 722–729. doi: 10.1038/ ng.128.
16. Wang Z, Gerstein M, Snyder M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet 2009; 10(1): 57–63. doi: 10.1038/ nrg2484.
17. Góngora-Castillo E, Buell CR. Bioinformatics challenges in de novo transcriptome assembly using short read sequences in the absence of a reference genome sequence. Nat Prod Rep 2013; 30(4): 490–500. doi: 10.1039/ c3np20099j.
18. Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics 2009; 25(14): 1754–1760. doi: 10.1093/ bioinformatics/ btp324.
19. Langmead B, Trapnell C, Pop M et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol 2009; 10(3): R25. doi: 10.1186/ gb-2009-10-3-r25.
20. Kim D, Pertea G, Trapnell C et al. TopHat2: accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biol 2013; 14(4): R36. doi: 10.1186/ gb-2013-14-4-r36.
21. Wu TD, Nacu S. Fast and SNP-tolerant detection of complex variants and splicing in short reads. Bioinformatics 2010; 26(7): 873–881. doi: 10.1093/ bioinformatics/ btq057.
22. De Bona F, Ossowski S, Schneeberger K et al. Optimal spliced alignments of short sequence reads. Bioinformatics 2008; 24(16): 174–180. doi: 10.1093/ bioinformatics/ btn300.
23. De Wit P, Pespeni MH, Ladner JT et al. The simple fool’s guide to population genomics via RNA-Seq: an introduction to high-throughput sequencing data analysis. Mol Ecol Resour 2012; 12(6): 1058–1067. doi: 10.1111/ 1755-0998.12003.
24. McKenna A, Hanna M, Banks E et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res 2010; 20(9): 1297–1303. doi: 10.1101/ gr.107524.110.
25. Koboldt D, Zhang Q, Larson D et al. VarScan 2: Somatic mutation and copy number alteration discovery in cancer by exome sequencing. Genome Res 2012; 22(3): 568–576. doi: 10.1101/ gr.129684.111.
26. Li H, Handsaker B, Wysoker A et al. The sequence alignment/ map format and SAMtools. Bioinformatics 2009; 25(16): 2078–2079. doi: 10.1142/ S0219720015500250.
27. Yu X, Sun S. Comparing a few SNP calling algorithms using low-coverage sequencing data. BMC bioinformatics 2013; 14 : 274. doi: 10.1186/ 1471-2105-14-274.
28. Recamo.cz. TransPEM (Software for Translation sequence into peptide based on mutation information). [online]. Dostupné z: https:/ / www.recamo.cz/ cz/ software/ transpem/ .
29. Ning K, Fermin D, Nesvizhskii A. Computational analysis of unassigned high-quality MS/ MS spektra in proteomic data sets. Proteomics 2010; 10(14): 2712–2718. doi: 10.1002/ pmic.200900473.
30. Sahin U, Derhovanessian E, Miller M et al. Personalized RNA mutanome vaccines mobilize poly-specific therapeutic immunity against cancer. Nature 2017; 547(7662): 222–226. doi: 10.1038/ nature23003.
31. Ott PA, Hu Z, Keskin DB et al. An immunogenic personal neoantigen vaccine for patients with melanoma. Nature 2017; 547(7662): 217–221. doi: 10.1038/ nature22991.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2019 Issue Supplementum 3-
All articles in this issue
- Méně časté mutace EGFR v kontextu léčby nemalobuněčného karcinomu plic
- CRISPR-Cas9 jako nástroj v terapii nádorových onemocnění
- Syntetická letalita – její současné využití a potenciál v onkologické léčbě
- Pokroky ve využití organokovových sloučenin při vývoji protinádorových léčiv
- Souhrn aktuálních poznatků o úloze estrogenového receptoru α v nádorové buněčné signalizaci
- Glykoproteiny v sérech onkologických pacientů
- Glykozylácia ako dôležitý regulátor funkcie protilátok
- Výskum ubikvitinácie proteínov v onkológii
- Dlouhé nekódující RNA – současné možnosti detekce a využití v klinické praxi
- Interakce onkogenních virových proteinů s proteiny rodiny p53
- Editorial 2019
- Kooperace genomických, transkriptomických a proteomických metod v detekci mutovaných proteinů
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Výskum ubikvitinácie proteínov v onkológii
- CRISPR-Cas9 jako nástroj v terapii nádorových onemocnění
- Glykozylácia ako dôležitý regulátor funkcie protilátok
- Méně časté mutace EGFR v kontextu léčby nemalobuněčného karcinomu plic
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

