-
Medical journals
- Career
Detekce superantigenů u izolátů Streptococcus pyogenes na základě dat celogenomové sekvenace
Authors: R. Veselá 1; S. Vohrnová 1,2; J. Kozáková 1
Authors‘ workplace: Státní zdravotní ústav, Centrum epidemiologie a mikrobiologie, Národní referenční laboratoř pro streptokokové nákazy 1; 3. lékařská fakulta Univerzity Karlovy, Praha 2
Published in: Epidemiol. Mikrobiol. Imunol. 72, 2023, č. 3, s. 191-194
Category: Short Communication
Overview
Streptococcus pyogenes způsobuje rozličná lidská onemocnění od nekomplikovaných infekcí dýchacích cest a kůže až po vážná invazivní onemocnění, která mohou být doprovázena syndromem toxického šoku. Významnými faktory virulence vedle M proteinu kódovaného genem emm jsou pyrogenní exotoxiny, které se považují za superantigeny. V Národní referenční laboratoři pro streptokokové nákazy byly nově zavedeny bioinformatické nástroje pro zpracování dat z celogenomové sekvenace S. pyogenes. Použitím programu SRST2 a platformy BV-BRC byla analyzována WGS data 10 kmenů S. pyogenes izolovaných od pacientů s invazivním onemocněním a byly stanoveny emm typy, sekvenční typy a profily genů kódujících superantigeny. K sestavení sekvencí genomů z krátkých čtení byla zvolena assembly pipeline Unicycler s de novo assemblerem SPAdes.
Klíčová slova:
celogenomová sekvenace – Streptococcus pyogenes – GAS – superantigen
ÚVOD
Streptococcus pyogenes (streptokok skupiny A, Group A Streptococcus, GAS) způsobuje rozličná lidská onemocnění od nekomplikovaných infekcí dýchacích cest a kůže až po vážná invazivní onemocnění spojená s vysokou nemocností a smrtností. Od 80. let 20. století se do popředí zájmu v USA i v Evropě dostaly případy streptokokového syndromu toxického šoku (Streptococcal Toxic Shock Syndrome, STSS), invazivní GAS infekce kůže a měkkých tkání, bakteriémie a sepse [1].
Genom GAS obsahuje geny pro produkci faktorů virulence. Typování GAS je založeno na N-koncové hypervariabilní sekvenci genu emm kódujícího povrchový M protein (anti-phagocytic M protein). Nejčastěji zastoupené emm typy invazivních GAS jsou emm1, emm28 a emm89 [2]. Vedle genu emm některé GAS vykazují v genomu až dva emm-like geny, také známé jako mrp (M-related protein), enn a sic (streptococcal inhibitor of complement). Transkripci těchto genů řídí regulon mga (M-protein trans-acting positive regulator).
Kmeny GAS spojené s invazivním onemocněním produkují také exotoxiny a specifické superantigeny (SAg), které vyvolávají systémovou zánětlivou odezvu. Superantigeny se váží na molekuly MHC II (major histocompatibility complex class II) na povrchu antigen prezentujících buněk a současně na variabilní oblast β řetězce receptoru T lymfocytů. Tato interakce vede k aktivaci velkého množství T lymfocytů produkujících prozánětlivé cytokiny a k potlačení produkce imunoglobulinů [3]. V současné době je známo 13 SAg. Klasickými biochemickými metodami byly identifikovány superantigeny SpeA (streptococcal pyrogenic exotoxin), SpeC, SSA (streptococcal superantigen) a smeZ (streptococcal mitogenic exotoxin Z). Pomocí počítačové analýzy genomů byly dále identifikovány další SAg – SpeG, SpeH, SpeI, SpeJ, SpeK, SpeL, SpeM, SpeQ a SpeR. Geny speG, speJ a smeZ leží na chromozomu. Ostatní geny jsou neseny na profázích nebo na jiných mobilních elementech, jejichž integrace do genomu bakterie silně přispívá ke genomové diverzifikaci GAS a k výskytu vysoce virulentních kmenů.
Metoda celogenomové sekvenace (whole genome sequencing, WGS) poskytuje data, ze kterých lze získat informace o genomu bakterií. V současné době se v některých databázích podstatně navýšil počet popsaných genů GAS, což umožnilo skládání genomů invazivních izolátů. Jedním z programů využívaných v naší studii je SRST2 (Short Read Sequence Typing for Bacterial Pathogens) [4], který pracuje s databází VFDB (Virulence Factor DataBase) [5]. SRST2 umožňuje srovnání genomu vyšetřovaných izolátů s referenční databází genů, a tak stanoví geny SAg přítomné v genomu GAS. Další platformou použitou v naší studii je PubMLST databáze [6] nabízející rozsáhlou škálu nástrojů k analýze WGS dat a platforma Bacterial and Viral Bioinformatic Center (BV-BRC) [7]. Platforma BV-BRC pracuje s databázemi VFDB a Victor [8].
Table 1. Charakterizace invazivních izolátů S. pyogenesTable 1. Characterization of invasive S. pyogenes isolates 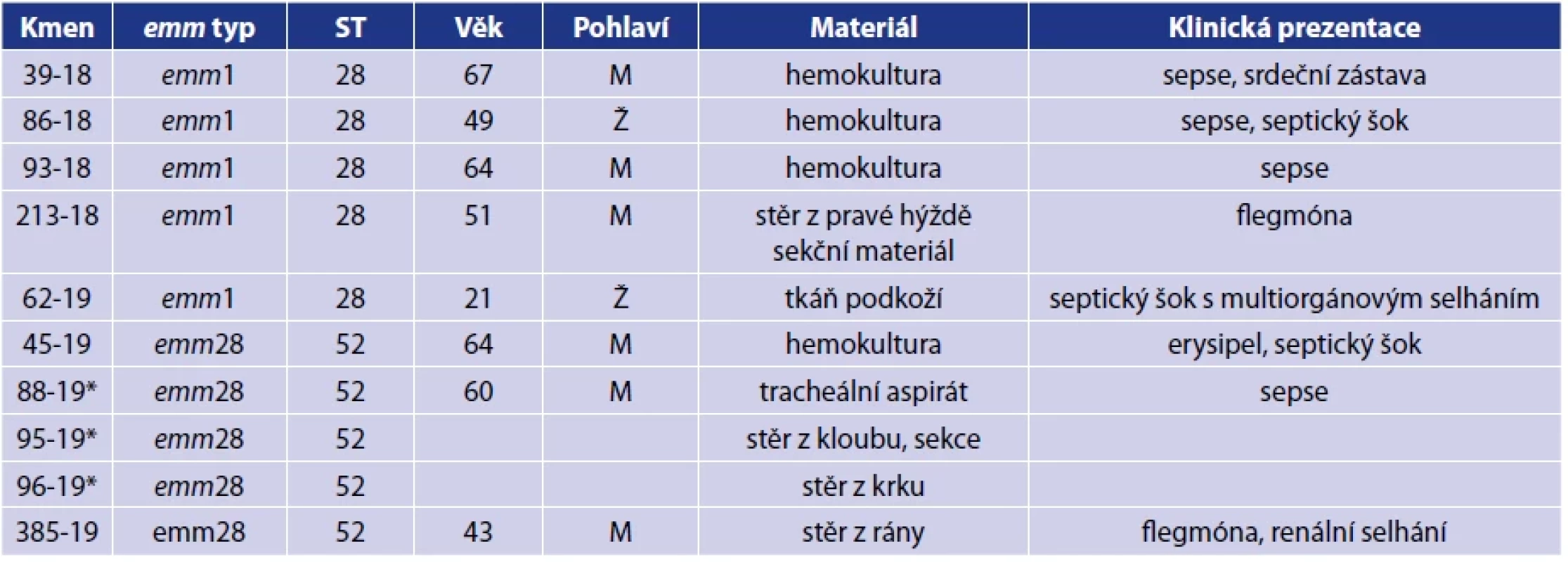
*kmeny izolované od jednoho pacienta; ST – sekvenční typ; M – muž; Ž – žena*isolates from one patient; ST – sequence type, M – male, F – female MATERIÁLY A METODY
Kmeny S. pyogenes
V rámci naší studie bylo celogenomové sekvenaci podrobeno deset izolátů invazivních GAS. Kmeny invazivních GAS byly získány z klinického materiálu pacientů s invazivním onemocněním v letech 2018–2019. Uvedené kmeny byly spojeny s diagnózami sepse, septický šok, flegmóna. Sedm z osmi pacientů zemřetypů, a to 5 kmenů typu emm1 a 5 kmenů typů emm28. Kmeny typu emm28 označené 96-19, 95-19 a 88-19 byly izolovány od jednoho pacienta v časovém rozmezí tří dnů a z materiálu rozličných biologických lokalit. Souhrn kmenů, klinické i epidemiologické informace o pacientech jsou uvedeny v tabulce 1.
Extrakce DNA
Z kmenů invazivních GAS byla izolována deoxyribonukleová kyselina (DNA) použitím kitu MagAttract® HMW DNA Mini kit (QIAGEN). Izolovaná DNA byla následně podrobena kvalitativní kontrole s použitím gelové elektroforézy a kvantitativní kontrole spektrofotometrickým měřením koncentrace DNA.
Celogenomová sekvenace a úprava dat
Příprava knihoven a celogenomová sekvenace byla provedena na pracovišti EMBL (European Molecular Biology Labolatory, Heidelberg, Německo) s využitím přístroje Illumina MiSeq. Našemu pracovišti byla následně poskytnuta raw WGS data ve formátu fastq souborů. K sestavení genomů z primárních raw WGS dat se použila platforma BV-BRC, která využívá assembly pipeline Unicycler verze v0.4.8 [9] s de novo assemblerem SPAdes [10]. Hodnota parametru K-mer byla zvolena 127. U dvou izolátů byla využita hodnota parametru K-mer 115. Průměrný počet kontigů v sestavených genomech byl 55 (47–62).
Zpracování dat z celogenomové sekvenace
K emm typování invazivních GAS se použila databáze PubMLST podporovaná platformou Bacterial Isolate Genome Sequence Database (BIGSdb) [11]. Ke zjištění přítomnosti genů virulentních faktorů speA, speC, speG, speH, speI, speJ, speK, speL, speM, smeZ a ssa a emm z dat WGS byl použit nástroj komplexní analýza genomu (Comprehensive Genome Analysis) platformy BV-BRC a program SRST2.
VÝSLEDKY A DISKUSE
Použitím databáze PubMLST byl pro všechny izoláty stanoven emm typ (viz tab. 1). Emm typy byly dále ověřeny použitím programu SRST2 a platformy BV-BRC, které zpracovávají primární raw WGS data. Program SRST2 potvrdil totožné emm typy všech kmenů. Platforma BV-BRC potvrdila emm geny u všech izolátů a umožnila vizualizovat uspořádání emm a emm-like genů. Mga locus u pěti kmenů typu emm28 označené 45-19, 88-19, 95-19, 96-19 a 385-19 obsahoval geny mga, mrp, emm, enn a scpA (C5a peptidáza). Další variantu uspořádání emm a emm-like genů vykazují izoláty typu emm1 označené 39-18, 86-18, 93-18, 213-18 a 62-19, kde se ve Mga lokusu vyskytují geny mga, emm, sic, scpA a je zde začleněn gen mlp (mobile element protein).
U všech deseti kmenů invazivních GAS se WGS analýzou s použitím platformy BV-BRC určil MLST (multilocus sequence typing) sekvenční typ (ST). Kmeny emm1 patří shodně k ST-28 a kmeny emm28 k ST-52.
Distribuce streptokokových pyrogenních toxinů speA, speC, speG, speH, speI, speJ, speK, speL, speM a streptokokového mitogenního toxinu smeZ a streptokového superantigenu ssa byla u kmenů stejného emm typu stejná. Potvrdila se jak použitím programu SRST2 pracujícího s referenční databází VFDB, tak komplexní analýzou genomu pomocí platformy BV-BRC pracující s referenčními databázemi VFDB a Victors.
Na základě alignmentu genomů všech izolátů invazivních GAS typu emm28 provedených použitím platformy BV-BRC se domníváme, že kmeny 95-19 a 96-19 izolované od jednoho pacienta jsou shodné, přičemž izolát 88-19 od téhož pacienta je geneticky odlišný.
Table 2. Distribuce genů sledovaných superantigenů hodnocená programem SRST2 a platformou BV-BRCTable 2. Superantigen gene distribution analysed by the SRST2 software and BV-BRC platform 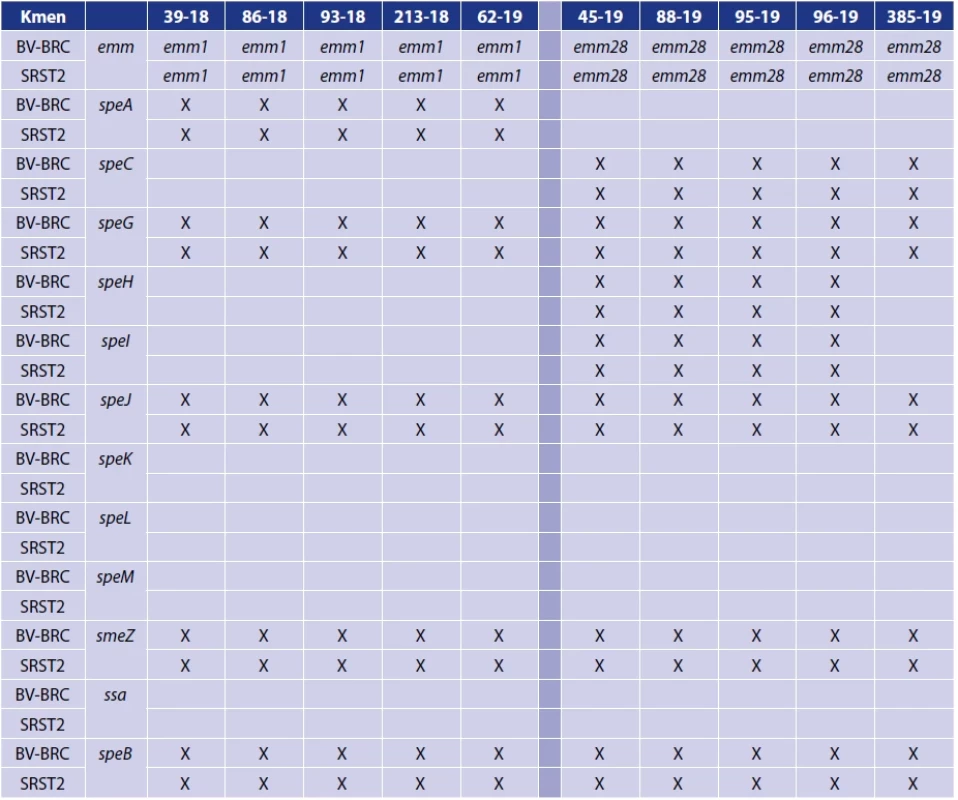
X – přítomnost daného genu v genomu kmeneX – presence of the gene in the strain genome ZÁVĚR
Tato studie vedla k zavedení možnosti využití programu SRST2 (tab. 2) a nástrojů platforem PuMLST a BV-BRC ke zpracování WGS dat invazivních GAS do NRL pro streptokokové nákazy. Analýza WGS dat invazivních GAS různými nástroji přispěje k upřesnění konfliktních výsledků týkajících se profilů streptokokových superantigenů publikovaných v literatuře. Streptokokové superantigeny se považují za významné faktory virulence, i když jejich spojitost s konkrétními klinickými projevy se nepodařilo definitivně prokázat. Geny streptokokových superantigenů mohou být považovány za markery přítomnosti a přenosu profágů. Přítomnost genů superantigenů v genomech invazivních GAS je podmínka nutná, nikoliv však dostačující, k jejich expresi na povrchu mikroorganismu nebo jako extracelulární produkt.
Podpořeno MZ ČR – RVO („Státní zdravotní ústav –SZU, 75010330“)
Do redakce došlo dne 8. 12. 2022.
Adresa pro korespondenci:
Ing. Renata Veselá
Státní zdravotní ústav
Šrobárova 49/48
110 00 Praha 10
e-mail: renata.vesela@szu.cz
Sources
- Holm SE. Invasive group A streptococal infections. The New England Journal of Medicine, 1996;335 : 590–591.
- Imohl M, Fitzner Ch, Perniciaro S, et al. Epidemiology and distribution of 10 superantigens among invasive Streptococcus pyogenes disease in Germany from 2009 to 2014. Plos One, 2017;12(7):e0180757. doi: 10.1371/journal.pone.0180757.
- Lintges M, Arlt S, Uciechowski P, et al. A new closed-tube multiplex real-time PCR to detect eleven superantigens od Streptococcus pyogenes dentifies a strain without superantigen aktivity. International Journal of Medical Microbiology, 2007;297 : 471–475.
- Inouye M, Dashnow H, Raven LA, et al. SRST2. Rapid genomic surveillance for public health and hospital mikrobiology labs. Genome Medicine, 2014;6(90). doi:10.1186/s13073-014-0090-6.
- Liu B, Zheng D, Zhou S, et al. VFDB 2022: a general classification scheme for bacterial virulence factors. Nucleic Acid Research, 2022;50(D1):D912–D917.
- Jolley KA, Bray JE, Maiden MCJ. Open-access bacterial population genomics: BIGSdb software, the PubMLST.org website and their applications. Wellcome Open Res, 2018;3 : 124. doi: 10.12688/wellcomeopenres.14826.1.
- Olson RD, Assaf R, Brettin T, et al. Introducing the Bacterial and Viral Bioinformatics Resource Center (BV-BRC: a resource combining PATRIC, IRD and ViPR. Nucleic Acids Research, 2023;51(D1):D678–D689.
- Sayers S, Li L, Ong E, et al. Victors: a web-based knowledge base of virulence factors in human and animal pathogens. Nucleic Acids Researchs, 2019;47(D1):D693–D700.
- Wick RR, Judd LM, Gorrie CL, et al. Unicycler: resolving bacteria genome assemblies from short and long sequencing reads. PloS Computational Biology, 2017;13(6):e1005595. doi: 10.1371/journal.pcbi.1005595.
- Bankevich A, Nurk S, Antipov D, et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. Journal of Computational Biology, 2012;19(5):455–477.
- Jolley KA, Maiden MCJ. BIGSdb: Scalable analysis of bacterial genome variation at the population level. BMC Bioinformatics, 2010;11(1). doi: 10.1186/1471-2105-11-595.
Labels
Hygiene and epidemiology Medical virology Clinical microbiology
Article was published inEpidemiology, Microbiology, Immunology

2023 Issue 3-
All articles in this issue
- Monitoring hladiny protilátok v súvislosti s očkovaním proti SARS-CoV-2 – 11-mesačné sledovanie
- Úloha endogénnych retrovírusov v ľudskom organizme
- Mykobakteriózy – nejčastější původci
- Extracelulární vezikuly v infekčním lékařství – význam a perspektivy
- Nárůst prevalence celiakie – kde hledat odpovědi?
- Lidská babesióza
- Detekce superantigenů u izolátů Streptococcus pyogenes na základě dat celogenomové sekvenace
- Prevalencia meticilín-rezistentného Staphylococcus aureus medzi obyvateľmi domovov dôchodcov na Slovensku
- Vzpomínka na RNDr. Václava Rupeše, CSc.
- Epidemiology, Microbiology, Immunology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Mykobakteriózy – nejčastější původci
- Lidská babesióza
- Nárůst prevalence celiakie – kde hledat odpovědi?
- Extracelulární vezikuly v infekčním lékařství – význam a perspektivy
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

