-
Medical journals
- Career
Chronická lymfocytární leukemie – současné využití moderních prognostických a prediktivních faktorů v diagnostice
Authors: Š. Pospíšilová 1,2; M. Jarošová 1,2; M. Doubek 1,2
Authors‘ workplace: Interní hematologická a onkologická klinika LF MU a FN Brno 1; Centrum molekulární medicíny, CEITEC, Masarykova univerzita, Brno 2
Published in: Transfuze Hematol. dnes,25, 2019, No. 1, p. 66-71.
Category: Review/Educational Papers
Overview
Poznatků o biologii a patogenezi chronické lymfocytární leukemie (CLL) za posledních 25 let dramaticky přibylo. Výrazně se rovněž změnila terapie této nemoci. Jde o jeden z nejvýraznějších pokroků v rámci celé onkologické hematologie. CLL má různorodý klinický průběh, který závisí na mnoha faktorech. Zjištění prognostických markerů CLL tak má dnes důležitou roli v přístupu k onemocnění u konkrétního pacienta i ve volbě vhodné terapie. Rozhodujícím prognostickým a prediktivním faktorem jsou aberace v genu TP53. Jejich význam roste v současné době nových léčebných možností u CLL. Zavedení zevrubných genomických přístupů do analýzy CLL vedlo k identifikaci dalších genetických abnormalit, které mohou přispět ke zlepšení stratifikace pacientů s CLL a ke sledování tak zvané klonální evoluce u této nemoci.
Klíčová slova:
chronická lymfocytární leukemie – prognostické faktory – prediktivní faktory – cytogenetika – TP53 – IGHV
ÚVOD
Když jsme v polovině 90. let 20. století léčili pacienta s chronickou lymfocytární leukemií (CLL), měli jsme jen omezené léčebné možnosti. Byl k dispozici chlorambucil, někteří nemocní dostávali kortikoidy, případně chemoterapeutické režimy podobně jako pacienti s lymfomy. Prognóza nemoci byla určována v podstatě jen podle systémů Raie či Bineta [1, 2]. Ve druhé polovině 90. let se začal používat fludarabin v monoterapii. Tento lék jsme považovali za malý zázrak. O to větší bylo naše zklamání, když u mnoha nemocných neměl fludarabin žádný léčebný účinek. Dodnes si tyto pacienty, mnohdy mladé, vybavujeme. Proč tomu tak bylo? První odpověď jsme dostali v roce 1997, kdy byl objeven prognostický význam delece krátkého raménka chromozomu 17, del(17p), tedy prognostická role genu TP53 [3]. V roce 2000 byl publikován první genomický model prognózy CLL [4]. A poté se již vršil jeden objev za druhým, což nakonec vedlo k tomu, že pacienti s CLL dnes žijí daleko déle než před 25 lety. Prevalence nemoci se za posledních 25 let více než zdvojnásobila (data České hematologické společnosti).
Dnes ještě lépe, než kdy dříve víme, že CLL má velmi variabilní klinický průběh závislý na celé řadě biologických, biochemických a genetických faktorů. Klasické prognostické faktory jako věk, pohlaví, klinické stadium, hladina β2-mikroglobulinu, výskyt somatických hypermutací v genu pro variabilní oblast těžkého řetězce imunoglobulinu (mutační stav IGHV) a chromozomální aberace jsou využívány v diagnostice již od začátku 21. století a dodnes jsou součástí prognostických modelů či skórovacích systémů [5–9]. Prudký rozvoj genomických technologií v posledních letech, převážně masivního paralelního sekvenování DNA neboli sekvenování nové generace (NGS), však umožnil další detailní analýzy genomu leukemických buněk a nalezení nových genetických markerů, které korelují s prognózou onemocnění a mohou predikovat odpověď na léčbu [10–12]. Celogenomové, resp. exomové sekvenování (WGS/WES) pomohlo identifikovat genetické mutace asociované s rozvojem a progresí onemocnění (tzv. „driver“ mutace), jejichž detekce může přispět k lepší stratifikaci pacientů a optimalizaci volby terapie. Nicméně i přes řadu probíhajících studií byl zatím přímý prognostický a/nebo prediktivní význam prokázán jen u několika genetických abnormalit.
PROGNOSTICKÉ A PREDIKTIVNÍ MARKERY DNES
Zatímco mnoho pacientů s CLL nemá po řadu let klinické příznaky onemocnění, jiní potřebují časný terapeutický zásah a taktéž vykazují rozdílnou odpověď na léčbu. Je zřejmé, že se tyto skupiny pacientů liší přítomností pozitivních nebo negativních prognostických a prediktivních faktorů. U CLL bylo popsáno mnoho prognostických faktorů poskytujících informaci o pravděpodobném vývoji onemocnění, přičemž některé z nich mohou sloužit i jako prediktivní faktory předpovídající efekt vybrané léčby. Avšak ne všechny z prognostických a prediktivních faktorů už byly validovány multivariačními analýzami nebo prospektivními klinickými studiemi. Za účelem definování nejvýznamnějších faktorů bylo pro CLL pacienty vytvořeno několik prognostických skórovacích systémů. Mezinárodní tým vedený prof. Hallekem publikoval metaanalýzu zahrnující data z 8 randomizovaných klinických studií, která identifikovala 5 nezávislých prognostických faktorů: delece a/nebo mutace TP53 genu, přítomnost somatických hypermutací IGHV, hladina sérového β2-mikroglobulinu, klinické stadium a věk [7, 9, 13]. Tento prognostický index (IPI, International Prognostic Index) tak zahrnuje klinické, biochemické i genetické faktory (tab. 1).
Table 1. Mezinárodní prognostický index 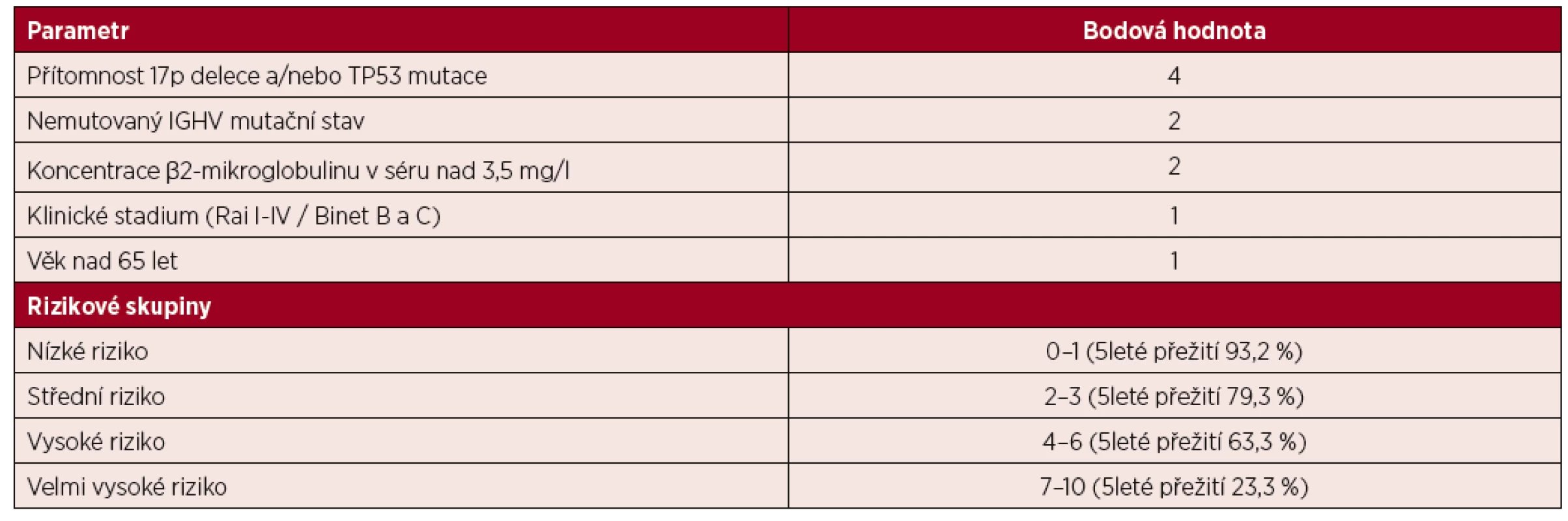
Index (IPI, International Prognostic Index) chronické lymfocytární leukemie [9]; IGHV – gen pro těžký řetězec imunoglobulinu Klinické stadium onemocnění (Rai 0–IV, Binet A-B-C) má přímý vliv na indikaci terapie, avšak neidentifikuje pacienty s vysokým rizikem progrese, a ani nepredikuje odpověď na léčbu. Jako významný prognostický faktor se ukazuje také věk (hranice 65 let), který má významný vliv na celkové přežití pacienta [9]. Z biochemických parametrů se jeví jako nejvýznamnější prognostický faktor sérová koncentrace β2-mikroglobulinu [14]. Z genetických faktorů je nejdéle využíván tzv. mutační stav IGHV neboli přítomnost somatických hypermutací v genu pro variabilní oblast těžkého řetězce imunoglobulinu. Mnoho studií jasně prokázalo, že jde o robustní prognostický marker založený na asociaci výskytu nemutovaných/minimálně mutovaných sekvencí IGHV genu s nepříznivou prognózou onemocnění [15, 16]. Mutační stav IGHV ve většině případů není ovlivněn progresí onemocnění, a obvykle je analyzován v počátečních fázích onemocnění – viz aktuální doporučení Evropské CLL organizace (ERIC) [17–20]. Jen vzácně může v průběhu onemocnění dojít ke změně mutačního stavu, obvykle v případě paralelního výskytu více leukemických klonů a v souvislosti s terapií [21, 22]. Zvyšující se prognostický význam má i podrobná imunogenetická charakterizace B-buněčného receptoru (BcR), zejména v souvislosti s terapeutickým využitím inhibitorů BcR dráhy. Analýzou rozsáhlého mezinárodního souboru CLL pacientů bylo zjištěno, že třetina CLL pacientů má tzv. stereotypní B-buněčné receptory [23] seskupené do jednotlivých skupin – tzv. subsetů, které vykazují podobné biologické i klinické vlastnosti – od velmi indolentních subsetů (např. subset 4) až po subsety s velmi agresivním chováním (např. subsety 1 a 2) [24]. Různé subsety stereotypních B-buněčných receptorů také korelují s výskytem různých typů rekurentních genetických mutací [25].
Klíčovou roli při stanovení prognózy CLL má nepochybně také detekce chromozomálních abnormalit pomocí fluorescenční in situ hybridizace (FISH). Na základě typu genomických aberací bylo definováno pět kategorií: del(17p), del(11q), trizomie 12q, normální karyotyp a samostatná del(13q), přičemž pacienti s del(17p) mají nejkratší medián doby bez terapie [4]. Lokus 17p13 kóduje antionkogen TP53 a jeho delece je často asociována s mutacemi druhé alely genu. TP53 mutace se vyskytují také samostatně, nezávisle na del(17p). Frekvence výskytu dysfunkce TP53 genu se významně liší v závislosti na stadiu onemocnění. Při diagnóze a v počátečních stadiích onemocnění se TP53 defekty vyskytují jen u několika procent nemocných, před první terapií je už TP53 gen inaktivován v 15–20 % případů a u pacientů refrakterních na chemoimunoterapii jsou TP53 abnormality detekovány až u poloviny CLL pacientů [26]. Defekty v genu TP53 jsou v současnosti nejvýznamnějším prognostickým i prediktivním markerem u CLL pacientů. Jsou spojeny s velmi špatnou prognózou a rezistencí na chemoimunoterapii. Mutace v TP53 genu a del(17p) proto musí být testovány vždy před začátkem každé terapie [27, 28], opakované analýzy jsou nezbytné kvůli možné selekci nových mutací v průběhu vývoje onemocnění [27]. Evropská CLL organizace (ERIC) provádí pravidelné certifikace laboratoří provádějících mutační analýzy TP53 genu u CLL, certifikaci už získalo více než 130 laboratoří z 28 zemí včetně ČR. Poslední výzkumy navíc ukazují, že dokonce i minoritní TP53 mutace (přítomné jen v malé frakci – klonu leukemických buněk) detekované pomocí vysoce senzitivní NGS technologie mohou představovat velmi špatnou prognózu [29–31]. O problematice významu minoritních mutací v TP53 genu se v současné době velmi intenzivně diskutuje také v rámci organizace ERIC, která koordinuje prospektivní studii cílenou na objasnění vlivu TP53 mutací přítomných v méně než 10 % leukemických buněk.
Delece 11q22-23 zahrnující gen ATM (ataxia-telangiectasia mutated) má taktéž negativní dopad na prognózu onemocnění, zvláště u starších pacientů. CLL pacienti s bialelickou delecí ATM mají zkrácené období do progrese (PFS) i celkové přežití (OS) [32].
V poslední době se ukazuje, že velmi významným prognostickým a prediktivním faktorem je rovněž přítomnost komplexního karyotypu. Komplexní karyotyp je možné určit pomocí klasického cytogenetického vyšetření, které se provádí kultivací buněk kostní dřeně, ale mnohem častěji kultivací buněk periferní krve. Z důvodů nízké proliferační aktivity nádorových B lymfocytů in vitro se B lymfocyty kultivují po dobu 72 hodin v médiu se stimulačními látkami, jako jsou CpG oligonukleotidy DSP30 a interleukin 2 (IL-2) [33, 34]. Komplexní karyotyp je obecně definován nálezem ≥ 3 nebo ≥ 5 chromozomových změn ve dvou a více metafázích [35–36]. Nepříznivý prognostický význam komplexního karyotypu nálezem 3 a více změn byl u nemocných s CLL opakovaně potvrzen [34–36]. U nemocných léčených ibrutinibem zkracuje například PFS i OS [37]. V poslední době se diskutuje o větším prognostickém významu komplexního karyotypu s nálezem 5 a více změn, a to zejména v kombinaci s defekty TP53 genu. Jaglowski et al. potvrdili, že tento nález nepříznivě ovlivňuje průběh onemocnění u CLL nemocných po alogenní transplantaci [38], Baliakas et al. pozorovali, že nález 5 a více změn nepříznivě ovlivňuje čas do první léčby (TTFT) [39]. Detekce komplexního karyotypu se proto stává součástí rutinní diagnostiky, zejména v souvislosti s možnou aplikací BcR inhibitorů.
Kromě uvedených nejčastěji používaných prognostických a prediktivních faktorů se začínají uplatňovat i další geny, jejichž prognostický význam byl identifikován celoexomovými a celogenomovými NGS analýzami [10, 11, 40]. Bylo prokázáno, že mutace v genech NOTCH1 a SF3B1 korelují s kratším celkovým přežitím CLL pacientů v klinických studiích [41, 42]. Mutace v NOTCH1 genu navíc asociují s kratším intervalem do první terapie (TFS) a vyšším rizikem Richterovy transformace [43]. Defekty v NOTCH1 and SF3B1 společně s mutacemi v dalších genech (BIRC3 nebo MYD88) byly zahrnuty do nových genetických prognostických modelů CLL [44, 45].
Prognostický nebo prediktivní význam dalších faktorů, jako jsou například exprese znaků CD38 a ZAP70, periferní lymfocytóza, infiltrace kostní dřeně nebo solubilní antigen CD23, už v současné době ustupuje do pozadí [8]. Lze naopak předpokládat, že u CLL poroste význam analýzy minimální zbytkové nemoci v průběhu terapie [46, 47]. V posledních letech je také přikládán stále větší význam roli metylací v rozvoji a průběhu onemocnění. Změny v metylačních profilech CLL pacientů byly jasně prokázány už v raných fázích onemocnění a metylační stav některých míst v genomu je také spojen s agresivnějším průběhem nemoci [48].
ZÁVĚR
Heterogenní klinický průběh CLL lze vysvětlit četnými rozdíly v imuno-, cyto - a molekulárněgenetických faktorech. Analýza těchto molekulárních faktorů při stanovení diagnózy a/nebo při progresi onemocnění (před první terapií) a/nebo při relapsu (před následnou terapií) je dnes jednoznačně doporučována (blíže tab. 2).
Table 2. Význam prognostických a prediktivních genetických faktorů u pacientů s chronickou lymfocytární leukemií 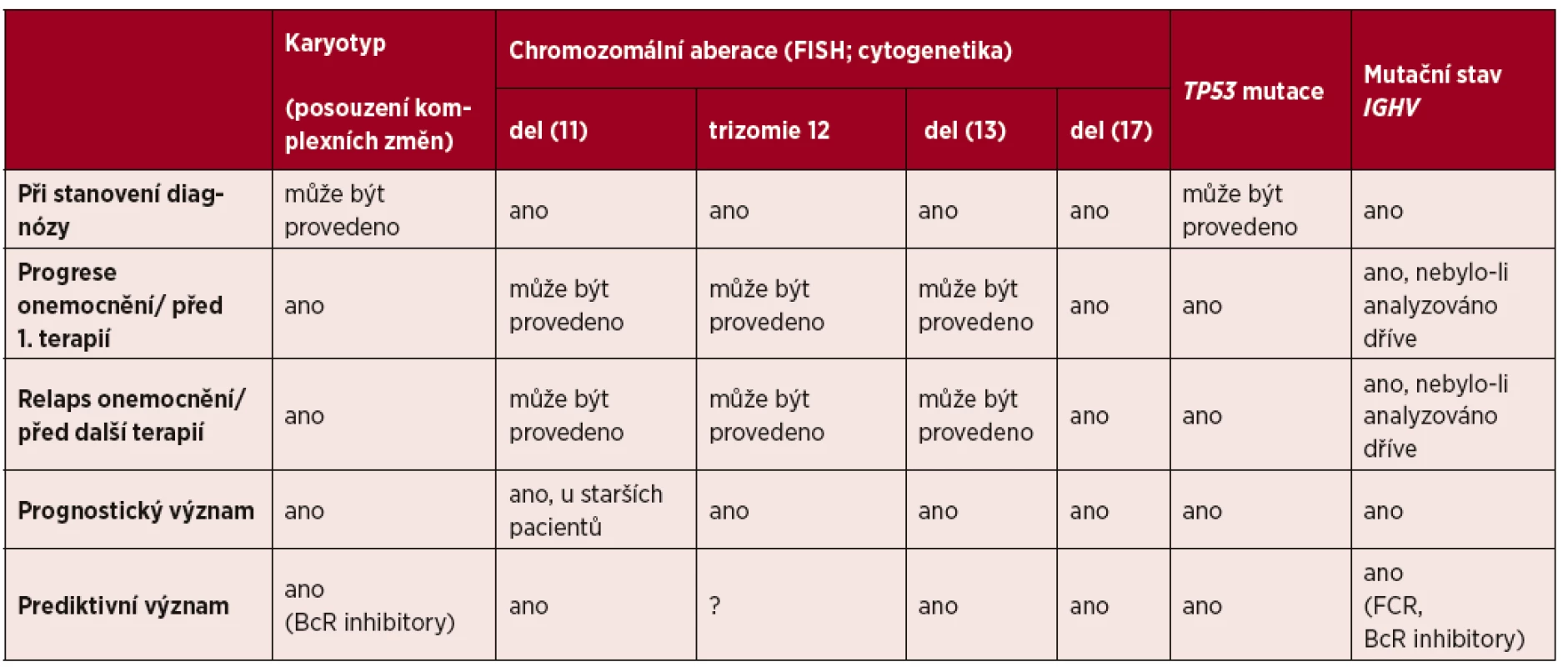
BcR-B –buněčný receptor; FCR – fludarabine, cyklofosfamid, rituximab; IGHV – gen pro těžký řetězec imunoglobulinu Využití moderních genomických technologií, především cíleného amplikonového NGS s vysokou citlivostí umožňuje rozkrýt podstatu nádorové heterogenity a klonální evoluce CLL [49] a stále více se stává součástí rutinní diagnostiky onemocnění. Zvyšující se dostupnost celogenomového sekvenování usnadňuje personalizovaný přístup k pacientům a optimalizaci volby terapie [50, 51].
Podíl autorů na přípravě rukopisu
ŠP, MJ, MD – příprava rukopisu
ŠP, MD – závěrečná kontrola rukopisu
Čestné prohlášení
Autoři práce prohlašují, že v souvislosti s tématem, vznikem a publikací tohoto článku nejsou ve střetu zájmů a vznik ani publikace článku nebyly podpořeny žádnou farmaceutickou firmou.
Poděkování
Práce byla podpořena grantovými projekty AZV MZ ČR 15-31834A-4/2015, AZV MZ ČR 15-30015A-4/2015 a EATRIS-CZ, reg.č. CZ.02.1.01/0.0/0.0/16_013/0001818 financovaného z EFRR.
Doručeno do redakce dne 8. 10. 2018.
Přijato po recenzi dne 13. 11. 2018.
prof. MUDr. Michael Doubek, Ph.D.
Interní hematologická a onkologická klinika LF MU a FN Brno
Jihlavská 20
625 00 Brno
e-mail: doubek.michael@fnbrno.cz
Sources
1. Rai KR, Sawitsky A, Cronkite EP, et al. Clinical staging of chronic lymphocytic leukemia. Blood 1975;46 : 219–234.
2. Binet JL, Auquier A, Dighiero G, et al. A new prognostic classification of chronic lymphocytic leukemia derived from a multivariate survival analysis. Cancer 1981;48 : 198–206.
3. Lens D, Dyer MJ, Garcia-Marco JM, et al. p53 abnormalities in CLL are associated with excess of prolymphocytes and poor prognosis. Br J Haematol 1997;99(4):848–857.
4. Dohner H, Stilgenbauer S, Benner A, et al. Genomic aberrations and survival in chronic lymphocytic leukemia. N Engl J Med 2000;343 : 1910–1916.
5. Hallek M, Cheson BD, Catovsky D, et al. iwCLL guidelines for diagnosis, indications for treatment, response assessment, and supportive management of CLL. Blood 2018;21;131(25):2745–2760.
6. Delgado J, Doubek M, Baumann T, et al. Chronic lymphocytic leukemia: A prognostic model comprising only two biomarkers (IGHV mutational status and FISH cytogenetics) separates patients with different outcome and simplifies the CLL-IPI. Am J Hematol 2017;92(4):375–380.
7. Doubek M, Špaček M, Pospíšilová Š, et al. Doporučení pro diagnostiku a léčbu chronické lymfocytární leukemie (CLL) – 2018. Transfuze Hematol dnes 2018;24(3):208–221.
8. Pflug N, Bahlo J, Shanafelt TD, et al. Development of a comprehensive prognostic index for patients with chronic lymphocytic leukemia. Blood 2014;124 : 49–62.
9. International CLL-IPI working group. An international prognostic index for patients with chronic lymphocytes leukemia (CLL-IPI): a meta-analysis of individual patient data. Lancet Oncol 2016;17 : 779–790.
10. Landau DA, Tausch E, Taylor-Weiner AN, et al. Mutations driving CLL and their evolution in progression and relapse. Nature 2015;526(7574):525–530.
11. Puente XS, Beà S, Valdés-Mas R, et al. Non-coding recurrent mutations in chronic lymphocytic leukaemia. Nature 2015;526(7574):519–524.
12. Rodríguez-Vicente AE, Bikos V, Hernández-Sánchez M, et al. Next-generation sequencing in chronic lymphocytic leukemia: recent findings and new horizons. Oncotarget 2017;8(41):71234–71248.
13. Navrkalová V, Kantorová B, Jarošová M, Pospíšilová Š. Molekulární prognostické markery CLL a jejich klinický význam. Klin Onkol 2015;28(S3):3S6–S15.
14. Pratt G, Thomas P, Marden N, et al. Evaluation of serum markers in the LRF CLL4 trial: β2-microglobulin but not serum free light chains, is an independent marker of overall survival. Leuk Lymphoma 2016;57(10):2342–2350.
15. Hamblin TJ, Davis Z, Gardiner A, Oscier DG, Stevenson FK. Unmutated Ig V(H) genes are associated with a more aggressive form of chronic lymphocytic leukemia. Blood 1999;94(6):1848–1854.
16. Damle RN, Wasil T, Fais F, et al. Ig V gene mutation status and CD38 expression as novel prognostic indicators in chronic lymphocytic leukemia. Blood 1999;94(6):1840–1847.
17. Ghia P, Stamatopoulos K, Belessi C, et al. ERIC recommendations on IGHV gene mutational status analysis in chronic lymphocytic leukemia. Leukemia 2007;21 : 1–3.
18. Langerak AW, Davi F, Ghia P, Hadzidimitriou A, Murray F, Potter KN, et al. Immunoglobulin sequence analysis and prognostication in CLL: guidelines from the ERIC review board for reliable interpretation of problematic cases. Leukemia 2011;25(6):979–84.
19. Rosenquist R, Ghia P, Hadzidimitriou A, et al. Immunoglobulin gene sequence analysis in chronic lymphocytic leukemia: updated ERIC recommendations. Leukemia 2017;31(7):1477–1481.
20. Sutton LA, Hadzidimitriou A, Baliakas P, et al.; European Research Initiative on CLL (ERIC). Immunoglobulin genes in chronic lymphocytic leukemia: key to understanding the disease and improving risk stratification. Haematologica 2017;102(6):968–971.
21. Plevova K, Francova HS, Burckova K, et al. Multiple productive immunoglobulin heavy chain gene rearrangements in chronic lymphocytic leukemia are mostly derived from independent clones. Haematologica 2014;99(2):329–338.
22. Brazdilova K, Plevova K, Skuhrova Francova H, et al. Multiple productive IGH rearrangements denote oligoclonality even in immunophenotypically monoclonal CLL. Leukemia 2018;32(1):234–236.
23. Agathangelidis A, Darzentas N, Hadzidimitriou A, et al. Stereotyped B-cell receptors in one-third of chronic lymphocytic leukemia: a molecular classification with implications for targeted therapies. Blood 2012;119(19):4467–4475.
24. Baliakas P, Hadzidimitriou A, Sutton LA, et al. Clinical effect of stereotyped B-cell receptor immunoglobulins in chronic lymphocytic leukaemia: a retrospective multicentre study. Lancet Haematol 2014;1(2):e74–e84.
25. Sutton LA, Young E, Baliakas P, et al. Different spectra of recurrent gene mutations in subsets of chronic lymphocytic leukemia harboring stereotyped B-cell receptors. Haematologica 2016;101(8):959–967.
26. Zenz T, Häbe S, Denzel T, et al. Detailed analysis of p53 pathway defects in fludarabine-refractory chronic lymphocytic leukemia (CLL): dissecting the contribution of 17p deletion, TP53 mutation, p53-p21 dysfunction, and miR34a in a prospective clinical trial. Blood 2009; 114(13):2589–2597.
27. Pospisilova S, Gonzalez D, Malcikova J, et al. ERIC recommendations on TP53 mutation analysis in chronic lymphocytic leukemia. Leukemia 2012;26(7):1458–1461.
28. Malcikova J, Tausch E, Rossi D, et al. ERIC Recommendations for TP53 mutation analysis in chronic lymphocytic leukemia – update on methodological approaches and results interpretation. Leukemia 2018;32(5):1070–1080.
29. Rossi D, Khiabanian H, Spina V, et al. Clinical impact of small TP53 mutated subclones in chronic lymphocytic leukemia. Blood 2014;123 : 2139–2147.
30. Malcikova J, Stano-Kozubik K, Tichy B, et al. Detailed analysis of therapy-driven clonal evolution of TP53 mutations in chronic lymphocytic leukemia. Leukemia 2015;29(4):877–885.
31. Nadeu F, Delgado J, Royo C, et al. Clinical impact of clonal and subclonal TP53, SF3B1, BIRC3, NOTCH1, and ATM mutations in chronic lymphocytic leukemia. Blood 2016;127(17):2122–2130.
32. Skowronska A, Parker A, Ahmed G, et al. Biallelic ATM inactivation significantly reduces survival in patients treated on the United Kingdom Leukemia Research Fund Chronic Lymphocytic Leukemia 4 trial. J Clin Oncol 2012;30(36):4524–4532.
33. Dicker F, Schnittger S, Haferlach T, et al. Immunostimulatory oligonucleotide-induced metaphase cytogenetics detect chromosomal aberrations in 80% of CLL patients: a study of 132 CLL cases with correlation to FISH, IgVH status, and CD38 expression. Blood 2006;108(9):3152–3160.
34. Haferlach C, Dicker F, Schnittger S, et al. Comprehensivegenetic characterization of CLL: a study on 506 cases analysed with chromosome banding analysis, interphase FISH, IgVH status and immunophenotyping. Leukemia 2007;21(12):2442–2451.
35. Baliakas P, Jeromin S, Iskas M, et al. Cytogenetic complexity in chronic lymphocytic leukemia: definitions, associations with other biomarkers and clinical impact; a retrospective study on behalf of Eric. Haematologica 2017;102(s2):170.
36. Puiggros A, Collado R, Calasanz MJ, et al. Patients with chronic lymphocytic leukemia and complex karyotype show an adverse outcome even in absence of TP53/ATM FISH deletion. Oncotarget 2017;8 : 54297–54303.
37. Thompson PA, O’Brien SM, Wierda WG, et al. Complex karyotype is a stronger predictor than del(17p) for an inferior outcome in relapsed or refractory chronic lymphocytic leukemia patients treated with ibrutinib-based regimens. Cancer 2015;121 : 3612–3621.
38. Jaglowski SM, Ruppert AS, Heerema NA, et al. Complex karyotype predicts for inferior outcomes following reduced-intensity conditioning allogeneic transplant for chronic lymphocytic leukaemia. Br J Haematol 2012;159 : 82–87.
39. Baliakas P, Iskas M, Gardiner A, et al. Chromosomal translocations and karyotype complexity in chronic lymphocytic leukemia: A systematic reappraisal of classic cytogenetic data. Am J Hematol 2014;89 : 249–255.
40. Hernández-Sánchez M, Kotaskova J, Rodríguez AE, et al. CLL cells cumulate genetic aberrations prior to the first therapy even in outwardly inactive disease phase. Leukemia; publikováno electronicky 12. září 2018; DOI: 10.1038/s41375-018-0255-1.
41. Quesada V, Conde L, Villamor N, et al. Exome sequencing identifies recurrent mutations of the splicing factor SF3B1 gene in chronic lymphocytic leukemia. Nat Genet 2011;44(1):47–52.
42. Oscier D, Rose-Zerilli MJ, Winkelmann N, et al. The clinical significance of NOTCH1 and SF3B1 mutations in the UK LRF CLL4 trial. Blood 2013;121 : 468–475.
43. Rossi D, Rasi S, Fabbri G, et al. Mutations of NOTCH1 are an independent predictor of survival in chronic lymphocytic leukemia. Blood 2012;119(2):521–529.
44. Rossi D, Rasi S, Spina V, et al. Integrated mutational and cytogenetic analysis identifies new prognostic subgroups in chronic lymphocytic leukemia. Blood 2013;121(8):1403–1412.
45. Baliakas P, Hadzidimitriou A, Sutton LA, et al. Recurrent mutations refine prognosis in chronic lymphocytic leukemia. Leukemia 2015;29(2):329–336.
46. Rawstron AC, Kreuzer KA, Soosapilla A, et al. Reproducible diagnosis of chronic lymphocytic leukemia by flow cytometry: an European Research Initiative on CLL (ERIC) & European Society for Clinical Cell Analysis (ESCCA) harmonisation project. Cytometry B Clin Cytom 2018;94(1):121–128.
47. Rawstron AC, Fazi C, Agathangelidis A, et al. A complementary role of multiparameter flow-cytometry and high-throughput sequencing for minimal residual disease (MRD) detection in chronic lymphocytic leukemia (CLL): An European research initiative on CLL (ERIC) study. Leukemia 2015;30(4):929–936.
48. Poppová L, Plevová K, Pospíšilová Š. Epigenetické změny jako nový nástroj pro prognostickou stratifikaci pacientů s chronickou lymfocytární leukemií. Transfuze Hematol dnes 2017;23(4):210–214.
49. Landau DA, Carter SL, Stojanov P, et al. Evolution and impact of subclonal mutations in chronic lymphocytic leukemia. Cell 2013;152(4):714–726.
50. Pospisilova S, Sutton LA, Malcikova J, et al. Innovation in the prognostication of chronic lymphocytic leukemia: how far beyond TP53 gene analysis can we go? Haematologica 2016;101(3):263–265.
51. Tom N, Pardy F, Kotašková J, Plevová K, Pospíšilová Š: Základní bioinformatické pojmy a postupy využívané pro analýzu DNA pomocí sekvenování nové generace. Transfuze Hematol dnes 2018;24(3):174–181.
Labels
Haematology Internal medicine Clinical oncology
Article was published inTransfusion and Haematology Today

2019 Issue 1-
All articles in this issue
- Úvodník
- Transfuze a hematologie dnes – průřez historií
- Vývoj transfuzní služby v České republice po roce 1990
- 25 let nepříbuzenského darování krvetvorných buněk
- Vývoj transplantací hematopoetických buněk v posledních 25 letech
- 25 rokov podpornej antiinfekčnej liečby v hematoonkológii
- 25 let vývoje metod molekulární biologie a jejich uplatnění v hemato(onko)logii
- 25 let vývoje dětské hematologie
- 25 rokov v komplexnej starostlivosti a liečbe hemofílie
- Ohlédnutí za vývojem antitrombotické profylaxe s akcentem na posledních 25 let
- Chronická lymfocytární leukemie – současné využití moderních prognostických a prediktivních faktorů v diagnostice
- Ohlédnutí za čtvrtstoletím léčby chronické lymfocytární leukemie
- Non-Hodgkinův lymfom v České republice
- Hodgkinův lymfom – nekončící příběh
- Vývoj diagnostiky a léčby mnohočetného myelomu v průběhu posledních 25 let
- Posledních 25 let v diagnostice a léčbě myelodysplastického syndromu
- Vývoj léčby chronické myeloidní leukemie v posledních 25 letech
- Transfusion and Haematology Today
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Hodgkinův lymfom – nekončící příběh
- Non-Hodgkinův lymfom v České republice
- Vývoj transfuzní služby v České republice po roce 1990
- Chronická lymfocytární leukemie – současné využití moderních prognostických a prediktivních faktorů v diagnostice
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

