-
Medical journals
- Career
Epigenetické změny jako nový nástroj pro prognostickou stratifikaci pacientů s chronickou lymfocytární leukemií
Authors: L. Poppová; K. Plevová; Š. Pospíšilová
Authors‘ workplace: CEITEC – Středoevropský technologický institut, Masarykova univerzita, Brno
Published in: Transfuze Hematol. dnes,23, 2017, No. 4, p. 210-214.
Category:
Overview
Chronická lymfocytární leukemie (CLL) je klonální B lymfocytární malignita s velmi heterogenním průběhem. Někteří pacienti zůstávají asymptomatičtí řadu let a nevyžadují léčbu, oproti nim pacienti s agresivní formou nemoci podstupují léčbu brzy po diagnóze. V posledních letech je přikládán stále větší význam roli metylací nejen pro přirozený vývoj, ale také při vzniku a vývoji různých onemocnění včetně nádorových. Z výsledků celogenomových metylačních studií vyplývá, že u CLL, podobně jako u jiných malignit, vznikají aberantní změny v metylačním profilu v raných fázích nemoci. Metylační profily CLL pacientů se mezi sebou liší a metylační status některých míst je spojen s agresivnějším průběhem nemoci. V tomto přehledu se zaměřujeme na metylační změny a jejich roli v prognóze CLL.
Klíčová slova:
chronická lymfocytární leukemie – CLL – epigenetika – metylace – prognóza – diferenciaceÚVOD
Chronická lymfocytární leukemie (CLL) je nejčastější leukemií dospělých v Evropě a Severní Americe. Jedná se o klonální nádorové onemocnění, při kterém dochází ke kumulaci malých zrale vypadajících B lymfocytů s fenotypem CD5+ CD19+ [1, 2]. Pacienti jsou často diagnostikovaní náhodně v asymptomatické fázi nemoci. CLL má však velmi heterogenní průběh od indolentní formy, která dlouhodobě nevyžaduje léčbu, po agresivní formu s nutností časné léčebné intervence. Z toho důvodu je žádoucí stanovení rizika progrese nemoci, aby bylo možné rozpoznat, u kterých pacientů je riziko nepříznivého průběhu nejvyšší. K tomuto účelu u CLL slouží řada prognostických faktorů. Mezi nejvýznamnější a široce přijímané patří mutační status genu kódujícího variabilní oblast těžkého řetězce imunoglobulinu (IGHV) [3, 4] a chromozomové aberace [5], z nichž největší význam má delece 17p zahrnující nádorový supresorový gen TP53 [6]. Recentní práce však poukazují také na velký potenciál zhodnocení epigenetického stavu maligních buněk pro stanovení prognózy CLL.
EPIGENETIKA A METYLACE DNA
Epigenetika se zabývá studiem změn v genové expresi, při nichž nedochází ke změnám v sekvenci DNA. Nejčastějšími epigenetickými modifikacemi DNA jsou metylace, které se vyskytují především na cytosinu v dinukleotidech s guaninem (cytosin-fosfát-guanin; CpG) [7]. Většina CpG dinukleotidů je soustředěna v CpG ostrůvcích, které jsou často hypometylované a najdeme je hlavně v promotorech genů [8, 9]. Naopak CpG dinukleotidy nacházející se mimo CpG ostrůvky jsou velmi často metylovány a vyskytují se v promotorech tkáňově specifických genů a v repetitivních oblastech, kde je jejich funkcí umlčet repetitivní oblasti a zachovat integritu genomu.
Metylace umožňují tkáňově specifickou expresi, a tím i diferenciaci a morfogenezi buněk. Také při diferenciaci multipotentní hematopoetické kmenové buňky do B lymfocytu je na základě signálů z okolního prostředí měněn metylační profil buňky a s ním i genová exprese, což umožňuje vývoj B lymfocytu (obr. 1). Metylační profil je dědičný a díky DNA metyltransferázám je zachováván při dělení buňky i mezigeneračně [10]. Během diferenciace a stárnutí narůstá metylace některých CpG ostrůvků, zatímco celková hladina metylace klesá [11]. Podobný, ale výraznější efekt, byl pozorován u nádorových buněk, kde dochází k masivní globální hypometylaci spojené s nestabilitou genomu a zároveň k metylaci promotorů tumor-supresorových genů.
Image 1. Stadia buněčné diferenciace B-lymfocytu 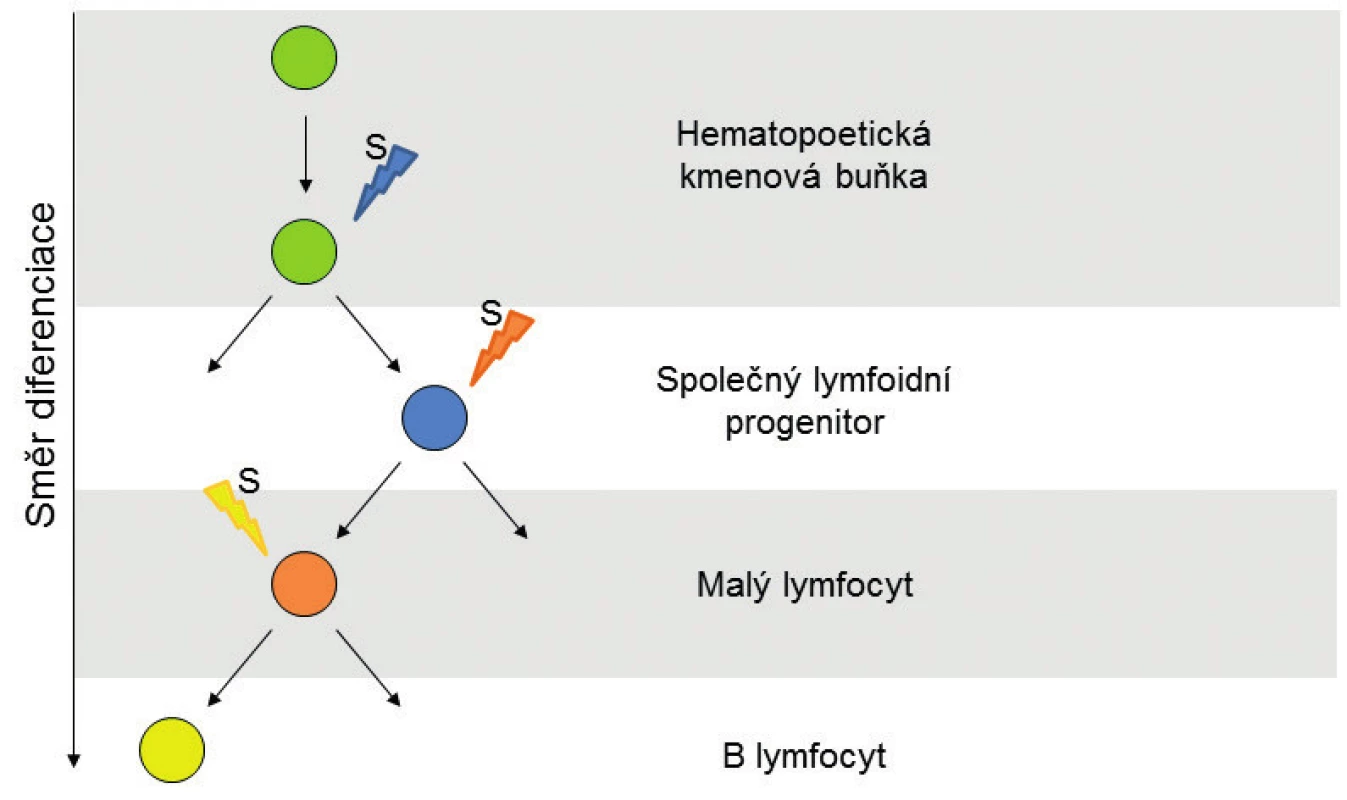
METYLACE DNA U CLL
U nádorů je metylační profil spojovaný nejen s deregulací genové exprese, ale také s buněčným původem a stadiem vývoje, což je u CLL velmi diskutované téma. Metylace výrazně ovlivňují normální vývoj B lymfocytů i jejich funkci [12]. Během zrání B lymfocytu dochází k výrazné celkové demetylaci a naopak k lokálním hypermetylacím. Některé z těchto procesů jsou přímo ovlivněny podněty z okolního prostředí, např. při aktivaci proliferace dochází k demetylaci některých promotorových oblastí a zesilovačů transkripce.
Skupina Martin-Subera identifikovala pomocí metod celogenomového bisulfitového sekvenování a vysokohustotních čipů 1649 CpG míst, jejichž míra metylace souvisí s buněčným původem CLL [13]. CLL buňky se nejvíce podobají dvěma stadiím vývoje lymfocytu: naivním buňkám, které se nepotkaly s antigenem a nedošlo u nich k hypermutacím imunoglobulinů, a paměťovým buňkám, které již prošly germinálním centrem a procesem hypermutace imunoglobulinů a přesmyku tříd. Z těchto 1 649 míst bylo vybráno pět CpG míst, která se nachází v genech B3GNTL1, CTBP2, TNF, v promotoru genu SCARF1 a v intergenové oblasti chromozomu 14. Pomocí těchto pěti CpG míst je možné rozdělit CLL pacienty do tří prognostických skupin [14].
První skupina pacientů „n-CLL“ (naive B-cell-like CLL) má leukemické buňky podobné naivním B lymfocytům, převažuje u ní vysoká míra metylace a prognóza pacientů z této skupiny je nejhorší vzhledem ke krátkému času do první terapie (TTFT) i kratšímu celkovému přežití (OS). Druhá skupina „m-CLL“ (memory B-cell-like CLL) vykazuje podobnosti k paměťovým B lymfocytům s nízkou mírou metylace a nejlepší prognózou. Kromě těchto dvou skupin byla identifikována ještě třetí skupina „i-CLL“ (intermediate CLL) vykazující střední hodnoty metylace i střední prognózu. Tyto tři prognostické skupiny se částečně překrývají s rozdělením podle procenta IGHV identity k nejbližší zárodečné sekvenci, avšak korelují lépe s TTFT než při rozdělení pacientů podle IGHV [14].
Bylo zjištěno, že skupiny pacientů definované na základě metylačních profilů mají také odlišné biologické charakteristiky. V souladu s nerovnoměrným zastoupením IGHV subgenů mezi pacienty s mutovaným a nemutovaným IGHV se liší jejich využití v jednotlivých skupinách podle metylačního profilu: u n-CLL je nejvýraznější IGHV1-69 (27 %) [4, 14]; u m-CLL IGHV4-34 (29 %); a u i-CLL skupiny IGHV3-23 (17 %) a IGHV1-18 (13 %) [1, 15]. O výskytu stereotypních subsetů ve skupinách definovaných na základě metylačních profilů dosud referuje pouze sdělení prezentované na konferenci Evropské hematologické asociace, které mimo jiné ukazuje, že stereotypní subset #2 s přestavbou IGHV3-21 tvoří jasně definovanou jednotku v rámci i-CLL [16]. Z hlediska chromozomových aberací a genových mutací u n-CLL převažují delece 11q, trizomie chromozomu 12 a mutace v genu NOTCH1, i-CLL má významné zastoupení delece 13q a zejména mutací v genu SF3B1 a u m-CLL je nejvýznamnější zastoupení delece 13q. Přes nesporný význam delecí 17p a mutací v genu TP53 dosud publikovaná data neukazují jejich asociaci s metylačními skupinami, což je dáno nízkou čestností defektů TP53 v testovaných kohortách [13, 14], a proto bude nutné tuto souvislost objasnit v budoucích studiích. Na obrázku 2 je schematicky znázorněn vztah jednotlivých skupin k přirozeným vývojovým stadiím B lymfocytu, úrovni celkové metylace, procentu IGHV identity a dalším faktorům.
Image 2. Schematické znázornění prognostických kategorií CLL definovaných na základě metylačního profilu a jejich vztahu k přirozeným vývojovým stadiím B lymfocytu, úrovni celkové metylace a klinickým faktorům 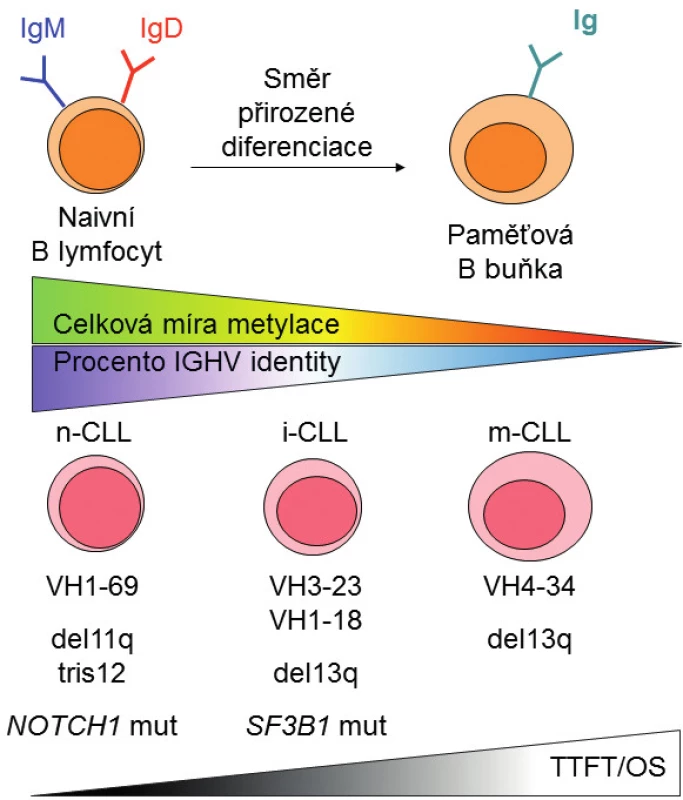
Existence tří metylačně odlišných prognostických skupin u CLL asociovaných k vývojovým stadiím zrání B lymfocytu byla nezávisle pozorována také dalšími autory [12], kteří analyzovali metylace spojené s procesem zrání B lymfocytu v místech vazby transkripčních faktorů. V průběhu zrání B lymfocytu dochází zejména k hypometylaci vazebných motivů a pouze lokální hypermetylaci CpG v transkripčně elongačních místech. Metylace vazebných míst pro transkripční faktory AP-1, EBF1 a RUNX3 spolu s profily hypermetylovaných CpG v transkripčně elongačních místech umožnily stratifikaci pacientů do tří prognostických skupin odpovídajících n-CLL, i-CLL a m-CLL [12, 14].
Mezi geny, které se významně liší v metylaci a zároveň v genové expresi u pacientů s mutovaným a nemutovaným IGHV, patří k nejvýznamnějším ZAP-70, PAX9 a CRY1 [13, 17, 18]. U těchto genů byla také potvrzena negativní korelace mezi hladinou metylace a genovou expresí.
Přibližně polovina míst aberantně metylovaných u CLL leží v nekódujících oblastech [19]. Subhash et al. detekovali dvě dlouhé nekódující RNA (lncRNA) s odlišně metylovanými promotory ve srovnání s kontrolními vzorky. Exprese těchto dvou lncRNA korelovala s expresí genů GDF7 a IRX5 zapojených do procesů podporujících nádorové bujení. Podobně jako u jiných malignit autoři zjistili výraznou hypometylaci v oblasti repetitivních elementů, která vede k nestabilitě genomu a umožňuje kumulaci genových mutací [11, 19].
Z hlediska stanovení prognózy CLL pacientů je důležité, že se metylační stav CLL při raných fázích nemoci a během jejího dalšího vývoje téměř nemění [12, 14, 20, 21]. Je také celkově stabilní i při srovnání hladin metylací u CLL pacientů před terapií a po ní [14]. Díky své stabilitě v čase a signifikanci, s jakou odlišuje pacienty s indolentní a agresivní formou nemoci, se stává dalším nástrojem umožňujícím stratifikaci CLL pacientů.
ZÁVĚR
Metylace DNA jsou epigenetickou modifikací, která významně ovlivňuje přirozený proces zrání B lymfocytu [12]. Při vývoji z naivního B lymfocytu k paměťové buňce dochází k celkové demetylaci. Při měření metylačního profilu CLL buněk se zjistilo, že metylační status CLL se konzervuje při raných fázích nemoci a během dalšího vývoje CLL se již téměř nemění. Byly publikovány různé studie, které se zaměřují na prognostický význam metylačních změn u CLL pacientů. Na základě postupu založeného na zhodnocení metylačního profilu výše uvedených pěti CpG míst lze CLL pacienty rozdělit do tří skupin s různou prognózou [14]. Dvě z těchto tří skupin vykazují významnou metylační podobnost s přirozenými vývojovými stadii B lymfocytu. Toto rozdělení není ovlivněno terapií a zároveň lépe koreluje s časem do první terapie než mutační status IGHV. Z hlediska klinické aplikace se tento postup do budoucna jeví jako využitelný při určování prognózy pacientů s CLL.
Seznam zkratek
- CLL – chronická lymfocytární leukemie
- CpG – cytosin-fosfát-guanin
- i-CLL – intermediateCLL
- IGHV – variabilní oblast těžkého řetězce imunoglobulinu
- lncRNA – dlouhé nekódující RNA
- m-CLL – memory B-cell-likeCLL
- n-CLL – naive B-cell-likeCLL
- OS – celkové přežití
- TTFT – čas do první terapie
Podíl autorů na přípravě rukopisu
LP – hlavní autor práce
KP – spoluautor práce
ŠP – korespondující autor, spoluautor práce
Čestné prohlášení autorů
Autoři práce prohlašují, že v souvislosti s tématem, vznikem a publikací tohoto článku nejsou ve střetu zájmů, a vznik ani publikace článku nebyly podpořeny žádnou farmaceutickou firmou.
Poděkování
Tato práce vznikla v rámci projektu CEITEC 2020 (LQ1601) za finančního přispění Ministerstva školství, mládeže a tělovýchovy české republiky v rámci účelové podpory z prostředků Národního programu udržitelnosti II.
Doručeno do redakce dne 8. 9. 2017.
Přijato po recenzi dne 25. 9. 2017.
prof. RNDr. Šárka Pospíšilová, Ph.D.
CEITEC – Středoevropský technologický institut
Kamenice 753/5
625 00 Brno
e-mail: pospisilova.sarka@fnbrno.cz
Sources
1. Dameshek W. Chronic lymphocytic leukemia – an accumulative disease of immunologically incompetent lymphocytes. Blood 1967;29(4): 566–584.
2. Caligaris-Cappio F, Ghia P. The normal counterpart to the chronic lymphocytic leukemia B cell. Best Pract Res Clin Haematol 2007;20(3): 385–397.
3. Damle BRN, Wasil T, Fais F, et al. Ig V gene mutation status and CD38 expression as novel prognostic indicators in chronic lymphocytic leukemia. Blood 1999;94(6): 1840–1847.
4. Hamblin BTJ, Davis Z, Gardiner A, Oscier DG, Stevenson FK. Unmutated Ig VH genes are associated with a more aggressive form of chronic lymphocytic leukemia. Blood 1999;94(6): 1848–1854.
5. Doehner H, Stilgenbauer S, Benner A, et al. Genomic aberrations and survival in chronic lymphocytic leukemia. N Engl J Med 2000;343(26): 1910–1916.
6. Delgado J, Doubek M, Baumann T, et al. Chronic lymphocytic leukemia: A prognostic model comprising only two biomarkers (IGHV mutational status and FISH cytogenetics) separates patients with different outcome and simplifies the CLL-IPI. Am J Hematol; publikováno elektronicky 13. února 2017. DOI: 10.1002/ajh.24660.
7. Bird AP. CpG-rich islands and the function of DNA methylation. Nature 1986;321(6067): 209–213.
8. Saxonov S, Berg P, Brutlag DL. A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters. Proc Natl Acad Sci U. S. A. 2006;103(5): 1412–1417.
9. Illingworth RS, Bird AP. CpG islands – “A rough guide.” FEBS Lett 2009;583(11): 1713–1720.
10. Goll MG, Bestor TH. Eukaryotic cytosine methyltransferases. Ann Rev Biochem 2005;74(1): 481–514.
11. Liu L, Wylie RC, Andrews LG, Tollefsbol TO. Aging, cancer and nutrition: the DNA methylation connection. Mech Ageing Dev 2003;124(10–12): 989–998.
12. Oakes CC, Seifert M, Assenov Y, et al. DNA methylation dynamics during B cell maturation underlie a continuum of disease phenotypes in chronic lymphocytic leukemia. Nat Genet 2016;48(3): 253–264.
13. Kulis M, Heath S, Bibikova M, et al. Epigenomic analysis detects widespread gene-body DNA hypomethylation in chronic lymphocytic leukemia. Nat Genet 2012;44(11): 1236–1242.
14. Queirós AC, Villamor N, Clot G, et al. A B-cell epigenetic signature defines three biological subgroups of chronic lymphocytic leukemia with clinical impact. Leukemia 2015;29(29): 598–605.
15. Tobin G, Thunberg U, Karlsson K, et al. Subsets with restricted immunoglobulin gene rearrangement features indicate a role for antigen selection in the development of chronic lymphocytic leukemia. Blood 2004;104(9): 2879–2885; 2017;104(9): 2879–2886.
16. Bhoi S, Mansouri L, Castellano G, et al. DNA methylation profiling in chronic lymphocytic leukemia patients carrying stereotyped B-cell receptors: a different cellular origin for subset. Haematologica 2017;102(S2): 68(Abstract P244).
17. Corcoran M, Parker A, Orchard J, et al. ZAP-70 methylation status is associated with ZAP-70 expression status in chronic lymphocytic leukemia. Haematologica 2005;90 : 1078–1088.
18. Rani L, Mathur N, Gupta R, et al. Genome-wide DNA methylation profiling integrated with gene expression profiling identifies PAX9 as a novel prognostic marker in chronic lymphocytic leukemia. Clin Epigenetics 2017;9(1): 57.
19. Subhash S, Andersson P-O, Kosalai ST, et al. Global DNA methylation profiling reveals new insights into epigenetically deregulated protein coding and long noncoding RNAs in CLL. Clin Epigenetics 2016;8(1): 106.
20. Cortese D, Sutton L, Cahill N, et al. On the way towards a “CLL prognostic index”: focus on TP53, BIRC3, SF3B1, NOTCH1 and MYD88 in a population-based cohort. Leukemia 2014;28(3): 710–713.
21. Cahill N, Bergh A-C, Kanduri M, et al. 450K-array analysis of chronic lymphocytic leukemia cells reveals global DNA methylation to be relatively stable over time and similar in resting and proliferative compartments. Leukemia 2013;27(1): 150–158.
Labels
Haematology Internal medicine Clinical oncology
Article was published inTransfusion and Haematology Today

2017 Issue 4-
All articles in this issue
- 25 let Českého národního registru dárců dřeně (ČNRDD) a Nadace pro transplantace kostní dřeně
- Epigenetické změny jako nový nástroj pro prognostickou stratifikaci pacientů s chronickou lymfocytární leukemií
- Angioimunoblastický T-lymfom: přehled problematiky, zkušenosti centra a kazuistika sekvenčního vzniku difuzního velkobuněčného B-lymfomu
- Sekvenování nové generace u akutní myeloidní leukemie: nový pohled na patogenezi a vývoj leukemických klonů
- Možnost ukončení terapie tyrozin-kinázovými inhibitory u nemocných s chronickou myeloidní leukemií a koncept „treatment-free remission“
- První perorální inhibitor proteazomu v léčbě relabujícího/refrakterního mnohočetného myelomu
- Transfusion and Haematology Today
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Angioimunoblastický T-lymfom: přehled problematiky, zkušenosti centra a kazuistika sekvenčního vzniku difuzního velkobuněčného B-lymfomu
- Sekvenování nové generace u akutní myeloidní leukemie: nový pohled na patogenezi a vývoj leukemických klonů
- Možnost ukončení terapie tyrozin-kinázovými inhibitory u nemocných s chronickou myeloidní leukemií a koncept „treatment-free remission“
- První perorální inhibitor proteazomu v léčbě relabujícího/refrakterního mnohočetného myelomu
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

