-
Medical journals
- Career
Molekulárně genetická studie mykobakteriálních kmenů prevalujících na území České republiky v roce 2014
Authors: M. Pinková; E. Tietzeová; I. Zemanová; V. Dvořáková
Authors‘ workplace: Státní zdravotní ústav Praha – Národní referenční laboratoř pro mykobakterie
Published in: Epidemiol. Mikrobiol. Imunol. 68, 2019, č. 3, s. 131-136
Category: Original Papers
Overview
Cíl práce: Identifikovat kmeny Mycobacterium tuberculosis prevalující na území České republiky v roce 2014.
Materiál a metodika: DNA 269 kmenů M. tuberculosis získaných od pacientů žijících na území ČR v roce 2014. Kmeny byly postupnou analýzou zařazeny do geneticky příbuzných skupin webovým softwarem MIRU-VNTRplus.
Výsledky: Byly identifikovány čtyři globální linie: 1 Indo-Oceánská, 2 Východo-Asijská (13,8 %), 3 Středo-Asijská a 4 Euro-Americká (83,6 %) se subliniemi: Haarlem (44,2 %), H37Rv, Cameroon, Latinská Amerika-Středomoří (LAM), Haarlem / X, NEW-1, URAL, S, X, TUR a UgandaI. Plná identita repetitivních sekvencí byla prokázána u 27,1 % kmenů dvou globálních linií: 2 Východo-Asijská (2 skupiny) a 4 Euro-Americká (21 skupin): sublinie Haarlem (11 skupin), H37Rv (3 skupiny), Cameroon (2 skupiny), Haarlem / X (2 skupiny), X (1 skupina), LAM (1 skupina) a S (1 skupina). V souboru získaných kmenů bylo sedm multirezistentních (MDR) kmenů bez klastrování. Čtyři MDR kmeny globální linie 2 Východo--Asijské (Beijing) a tři MDR kmeny globální linie 4 Euro-Americké (2 kmeny sublinie Latinská Amerika-Středomoří (LAM) a 1 kmen sublinie Haarlem).
Závěry: Jednoletá studie zaměřená na identifikaci mykobakteriálních kmenů prevalujících na celém území České republiky v roce 2014. Studie přináší první geografický obraz o plošném rozložení kmenů M. tuberculosis v populaci.
Klíčová slova:
linie Mycobacterium tuberculosis – repetitivní sekvence – identifikace
ÚVOD
Tuberkulóza (TBC) je zánětlivé onemocnění patřící v České republice k povinně hlášeným nemocem. Onemocnění způsobují acidorezistentní tyčky pomalu rostoucí bakterie Mycobacterium tuberculosis komplex, tzv. Kochův bacil. Nejčastěji je způsobené druhem M. tuberculosis. Celosvětově patří TBC stále k nejsledovanějším nemocem, patří mu druhá příčka mezi úmrtími na infekční nemoci. Ve světě je sociální chorobou značně rozšířenou v rozvojových zemích. Vysoký výskyt tohoto onemocnění je především v Číně, Indii a Ruské federaci. Tyto země se potýkají také s vyšším výskytem rezistence na antibiotika používaná k léčbě TBC. Většina hlášených multirezistentních (MDR) kmenů M. tuberculosis pochází tedy z východní Evropy a centrální Asie. Česká republika patří podle WHO mezi oblasti s nejnižším výskytem tohoto onemocnění na světě, a to i v porovnání s evropskými zeměmi [1]. V roce 2014 byla incidence onemocnění TBC v České republice 4,8/100 000 obyvatel. Vyšší incidenci než republikový průměr mělo hlavní město Praha (7,8/100 000 obyvatel), kraj Středočeský (5,8/100 000 obyvatel), Pardubický (5,4/100 000 obyvatel), Olomoucký (5,2/100 000 obyvatel) a Královehradecký (5,1/100 000 obyvatel). Nejnižší incidence počtu hlášených onemocnění byla v Jihočeském kraji (2,4/100 000 obyvatel). Podíl pacientů s onemocněním TBC, který tvoří cizí státní příslušníci, byl v roce 2014 18,7 % a podle zdravotnických statistik stále mírně narůstá [2].
Sledování neustále probíhajícího evolučního vývoje M. tuberculosis umožňují polymorfismy v DNA. Genotypizace M. tuberculosis je pro mikrobiologii a epidemiologii nepostradatelná. Důležitost genotypizace tuberkulózy potvrdily za posledních 20 let stovky významných publikací [3, 4]. Pro identifikaci a analýzu polymorfismu některých typů opakujících se jednotek v DNA jsou využívány tyto molekulární metody: RFLP (Restriction fragment length polymorphism), spoligotypování a MIRU-VNTR (Mycobacterial Interspersed Repetitive Unit-Variable Number of Tandem Repeats) či celogenové sekvenování [4].
V předkládané studii bylo provedeno molekulárně genetické testování pomocí metody MIRU-VNTR druhu M. tuberculosis s cílem rozčlenit tento druh do linií, sublinií a vytvoření geneticky příbuzných skupin pro potřeby současných i budoucích epidemiologických studií, zejména za účelem zjišťování patogenity.
MATERIÁL A METODIKA
Pro izolaci DNA a identifikaci genotypů byly použity následující kity a reagencie – izolační kitGenoLyse (HAIN), 24 párů primerů [5] (GeneriBiotech), HotStarTaq DNA Polymerasekit (QIAGEN), polymeráza KAPA2G Robust (Elisabeth Pharmacon), PCR dNTP mix (Top-Bio), agaróza (SIGMA), interkalační barvivo SERVA DNA Stain G (SERVA), 50 bp DNA Ladder (NEW ENGLAND BioLabs), 100 bp DNA Ladder (NEW ENGLAND BioLabs).
K identifikaci byl použit referenční kmen M. tuberculosis H37Rv a 269 kmenů M. tuberculosis získaných od pacientů s TBC žijících v roce 2014 na území čtrnácti statutárních celků České republiky (Hlavního města Praha a třinácti krajů: Středočeského, Jihočeského, Plzeňského, Karlovarského, Ústeckého, Libereckého, Královehradeckého, Pardubického, Olomouckého, Moravskoslezského, Jihomoravského, Zlínského a Kraje Vysočina). Mezi analyzovanými kmeny bylo sedm MDR kmenů; čtyři kmeny z hl. m. Prahy a po jednom kmenu ze Středočeského, Libereckého a Moravskoslezského kraje. V souboru 269 kmenů bylo 57 kmenů od pacientů s cizí státní příslušností.
Ze získaných kmenů byla v naší laboratoři provedena izolace DNA pomocí izolačního kitu GenoLyse (HAIN) a zhotoveno 269 izolátů DNA. Z každého izolátu M. tuberculosis a referenčního kmene H37Rv byly polymerázovou řetězovou reakcí (PCR, z anglického Polymerase Chain Reaction) připraveny amplikony (PCR produkty vzniklé metodou rychlého selektivního zmnožení – amplifikací určité oblasti DNA v podmínkách in vitro). Amplifikované úseky DNA byly ohraničeny čtyřiadvaceti páry tzv. primerů (oligonukleotidů), což jsou krátké fragmenty DNA [6] (GeneriBiotech). Syntéza vybraného úseku DNA probíhala od přisedlých primerů termostabilními polymerázami HotStarTaq DNA Polymerasekit (QIAGEN) nebo KAPA2G Robust (Elisabeth Pharmacon). Pro genetickou analýzu lokusů byly PCR produkty nanášeny na 2% agarózový gel (agaróza SIGMA) a vizualizovány interkalačním barvivem SERVA DNA Stain G. Fotodokumentace gelů byla pořizována fotoaparátem Canon PowerShot_SX510_HS. Na fotodokumentaci byl vizuálně odečten počet repetitivních sekvencí podle vyhodnocovací tabulky [6, 7]. K porovnání velikostí fragmentů DNA generovaných v PCR byl použit 50 bp DNA Ladder a 100 bp DNA Ladder (NEW ENGLAND BioLabs).
Mykobakteriální kmeny získané od pacientů v roce 2014 byly označeny pětimístným či šestimístným kódem. První dvě čísla značí rok, kdy byl materiál odebrán. Číslo izolátu představují tři či čtyři následující čísla. Pod těmito kódy byly kmeny řazeny do fylogeneticky příbuzných skupin. Shluková analýza a identifikace linií a sublinií byla provedena online přístupným webovým softwarem MIRU - -VNTRplus [6, 7]. Tento volně přístupný software umožňuje uživatelům analyzovat údaje získané vyšetřením jejich kmenů. Referenční databáze kmenů reprezentuje 186 kmenů MTBC (Mycobacterium tuberculosis komplex).
Panel MIRU-VNTR se skládá z následujících 24 lokusů: 154 - 424 - 577 - 580 - 802 - 960 - 1644 - 1955 - 2059 - 2163b - 2165 - 2347 - 2401 - 2461 - 2531 - 2687 - 2996 - 3007 - 3171 -3192 - 3690 - 4052 - 4156 - 4348. Profily kmenů představují sérii 24 čísel odpovídajících počtu opakování v repetitivních sekvencích v každém lokusu. Například počet repetitivních sekvencí referenčního kmene H37Rv je: 2-2-4-3´-1-3-2-2-2-5-3-4-2-3-6-1-3-3-3-3-5-5-2-2 - [6, 7]. Klastrované izoláty jsou definovány jako dva nebo více izolátů se shodným počtem repetitivních sekvencí u 24 lokusů ve výše uvedeném pořadí.
VÝSLEDKY
Kmeny M. tuberculosis získané od 269 pacientů (z toho 57 cizinců a 7 MDR) žijících v roce 2014 ve čtrnácti územních celcích České republiky (ČR) byly rozděleny do linií a sublinií a zařazeny do geneticky příbuzných skupin.
Z globálních linií byla v našem souboru identifikována linie 1 Indo-Oceánská: EAI n = 4, 2 Východo-Asijská: Beijing n = 37, 3 Středo-Asijská: Delhi/CAS n = 3 a 4 Euro-Americká: n = 225 sublinie: Haarlem n = 119, H37Rv n = 36, Cameroon n = 24, Latinská Amerika-Středomoří (LAM) n = 13, Haarlem/X n = 9, NEW-1 n = 8, URAL n = 6, S n = 5, X n = 3, TUR =1 a Uganda I n = 1. Grafické znázornění kvantitativního zastoupení prevalujících linií/sublinií v ČR představuje graf 1. Rozložení kmenů genotypu Beijing, Haarlem, Latinská Amerika-Středomoří (LAM) a ostatních genotypů Euro-americké linie 4 v jednotlivých krajích ČR znázorňuje graf 2.
Graph 1. Kvantitativní zastoupení linií a sublinií linie 4 M. tuberculosis prevalujících v ČR v roce 2014.
Graph 1. Quantitative distribution of M. tuberculosis lineages and lineage 4 sublineages in the Czech Republic in 2014
Graph 2. Rozložení kmenů genotypu Beijing, Haarlem, LAM a ostatních genotypů 4 Euro- -Americké linie na území ČR v roce 2014.
Graph 2. Distribution of strains of genotypes Beijing, Haarlem, LAM, and other Euro- -American lineage 4 genotypes in the Czech Republic in 2014
Plná identita repetitivních sekvencí 24 lokusů byla zjištěna u dvou globálních linií: 2 Východo-Asijské a 4 Euro-Americké (linie 1 a 3 neklastrovaly). Tabulka 1 uvádí počet klastrů, jejich velikost a poměrné zastoupení v populaci. Grafické vyjádření výsledků shlukové/klastrové analýzy ilustruje dendrogram na obrázku 1.
Table 1. Diferenciace kmenů druhu M. tuberculosis v roce 2014
Table 1. Identification of M. tuberculosis strains in 2014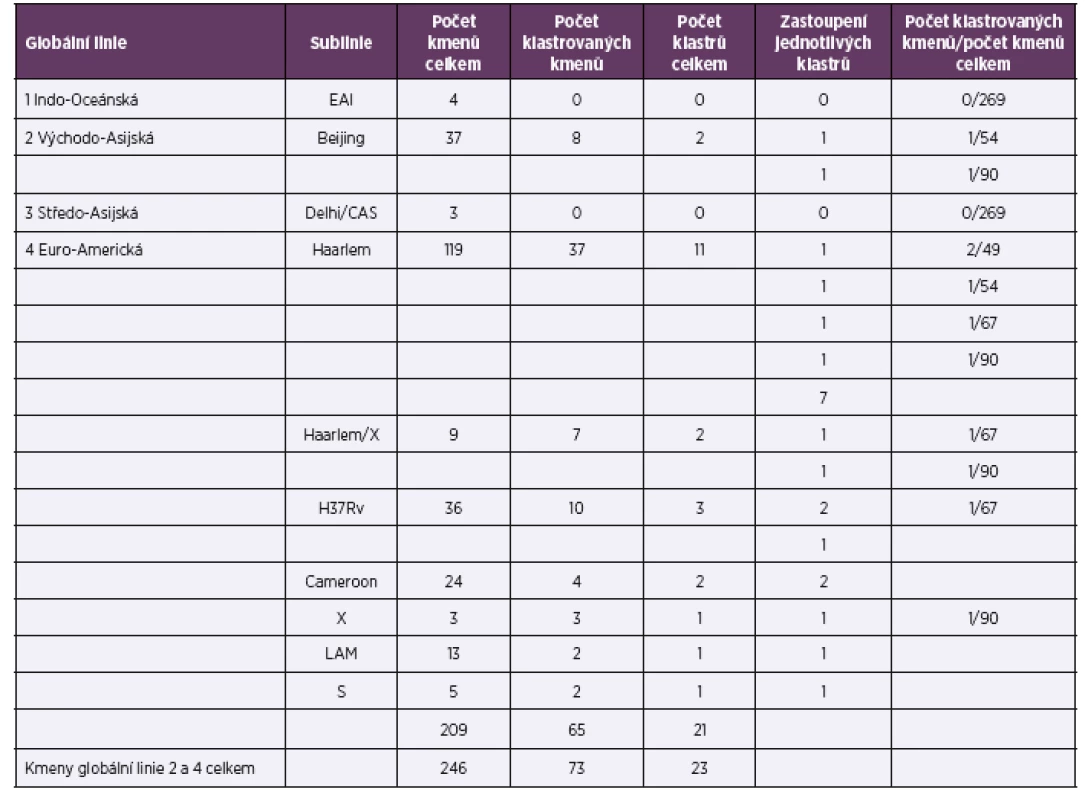
Ze souboru 269 kmenů bylo 6 kmenů od pacientů s cizí státní příslušností součástí šesti klastrových skupin (označeni hvězdičkou *, viz obr. 1). Čtyři kmeny byly zařazeny do globální linie 4 Euro-Americká (sublinie Haarlem, S a H37Rv) a dva kmeny do linie 2 Východo-Asijská (sublinie Beijing; 51 kmenů neklastrovalo).
Image 1. Dendrogram klastrovaných kmenů linie 2 Východo-Asijské a 4 Euro- -Americké na území ČR v roce 2014 Vysvětlivky: *kmen získaný od pacienta s TBC s cizí státní příslušností akmeny získané od matky a dcery bkmeny získané od dvou bratrů
Figure 1. Dendrogram of clustered East-Asian lineage 2 and Euro-American lineage 4 strains the Czech Republic in 2014 Note: *strain obtained from a non-resident TB patient astrains obtained from a mother and her daughter bstrains obtained from two brothers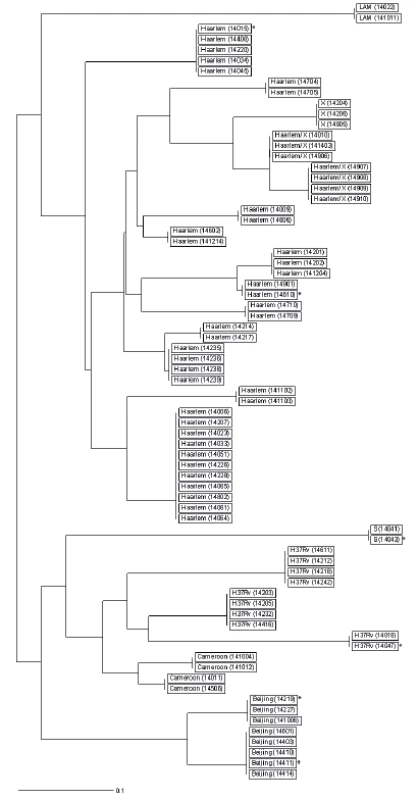
Sublinie Haarlem (linie 4 Euro-Americká) vytváří nejvyšší podíl klastrovaných kmenů v ČR; celkem 11 klastrových skupin. U dvou skupin se dvěma klastrovanými kmeny sublinie Haarlem byl prokázán příbuzenský vztah. U skupiny čtyř klastrovaných kmenů sublinie Haarlem/X byla zjištěna blízkost bydliště (viz tab. 1, obr. 1).
Majoritní linie 4 Euro-Americká prevalovala na celém území ČR (viz graf 2), způsobila onemocnění TBC 83,6 % pacientům (ve 13 % u cizích státních příslušníků). Další významnou sublinií, která byla identifikována u 13,8 % pacientů (z toho 5,6 % cizinců) je linie 2 Východo-Asijská sublinie Beijing. Tato sublinie byla identifikována ve všech krajích kromě kraje Jihočeského, Libereckého, Královéhradeckého a Zlínského (viz graf 2).
Kmeny globální linie 1 Indo-Oceánská, 3 Středo-Asijská sublinie Delhi/CAS, 4 Euro--Americká sublinie NEW-1, URAL, X, TUR a Uganda I byly bez klastrování.
Kmeny MDR (7 kmenů; 2,6 %) nebyly klastrovány. Byla u nich identifikována linie 2 Východo--Asijská sublinie Beijing (4 kmeny) a linie 4 Euro-Americká sublinie LAM (2 kmeny) a sublinie Haarlem (1 kmen). Ze skupiny MDR kmenů patřily 4 kmeny pacientům s cizí státní příslušností.
DISKUSE
V předkládané studii bylo provedeno genotypování pomocí metody MIRU-VNTR repetetivní sekvence kmenů M. tuberculosis u 269 pacientů. Jedná se o molekulárně genetickou metodu navazující na RFLP a spoligotyping. V posledních deseti letech se v rámci TBC tyto metody staly základem pro diferenci genotypů a jejich distribuci v národní i nadnárodní úrovni [4]. Vzhledem k tomu, že je TBC v nadnárodním měřítku vedoucí příčinou úmrtí na infekční onemocnění, v roce 2017 [8] na toto onemocnění celosvětově zemřelo odhadem 1,7 milionů nakažených, je tomuto onemocnění věnována široká vědecká pozornost. Jak již bylo zmíněno výše, ačkoli je incidence onemocnění TBC v ČR pouze 4,8/100 000 obyvatel, představuje toto onemocnění závažnou nákazu, které je třeba věnovat pozornost a intenzivně pracovat na epidemiologických studiích. Vzhledem k tomu, že globální zátěž TBC spadá především na rozvojové země, jsou pro návrh vhodných strategií pro kontrolu nemoci nezbytné přesné odhady přistěhovalectví. Tento trend potvrzuje např. studie [9], z níž je patrno, že z 1 080 pacientů, kteří byli do studie zahrnuti, bylo 614 pacientů (56,9 %) italské národnosti a 466 pacientů (43,1 %) byli cizinci (z toho 27,9 % pocházelo z Afriky, 23,6 % z Asie, 22,3 % z východní Evropy a 11,6 % ze Střední a Jižní Ameriky). V námi předkládané práci bylo zjištěno, že v České republice (ČR) z 269 pacientů, pocházelo pouze 57 kmenů od pacientů s cizí státní příslušností, tedy o polovinu méně, než u výše zmíněné studie v Itálii. V ČR však není tak vysoký výskyt cizinců z Afriky a Jižní Ameriky. Dále bylo zjištěno, že ze skupiny 7 multirezistentních kmenů patřily 4 kmeny pacientům s cizí státní příslušností. Fakt, že v ČR není v současné době tak vysoký podíl cizinců z celkového počtu pacientů, nijak neubírá na důležitosti předkládané studie a lze spíše předpokládat, že v souvislosti s globalizací bude trend incidence TBC v ČR minimálně u cizinců vzrůstající.
V současnosti je M. tuberculosis komplex způsobující onemocnění TBC u člověka řazen do sedmi fylogenetických globálních linií: linie 1 Indo-Oceánská (EAI), linie 2 Východo-Asijská (Beijing), linie 3 Středo-Asijská (CAS), linie 4 Euro-Americká, Africká linie 1, Africká linie 2 a Etiopská linie 7. V předkládané studii byl testován druh M. tuberculosis, u kterého řada autorů popisuje 5 globálních linií: EAI, Beijing, CAS, Euro-Americká linie a poslední identifikovaná linie byla linie Etiopská [6]. V naší studii byly identifikovány 4 linie z 5 a to i u pacientů s cizí státní příslušností (tabulka 1, graf 1; linie 7 nebyla identifikována). Tyto výsledky korespondující s již dříve publikovanými daty získanými v rámci evropských studií, z nich je rovněž patrno, že největší zastoupení má linie 4 Euro-Americká, následuje linie 2 Východo-Asijská (Beijing) a malé nebo žádné zastoupení má linie 1 EAI, linie 3 CAS a linie 7 Etiopská [9]–[11]. Bylo jednoznačně zjištěno, že nejrozšířenějším původcem TBC v populaci je kmen globální linie 4 (Euro-Americká). Prevalujícími subliniemi byly Haarlem a LAM. Tyto výsledky jsou opět v souladu s již dříve publikovanými daty u jiných autorů [12, 13], v naší studii byl největší počet kmenů zařazen do sublinie Haarlem (obrázek 1, graf 1, tabulka 1). Ke stejnému závěru dospěla i naše studie z roku 2013–2014 v Praze [14]. Důvody globálního úspěchu linie 4 a jejích sublinií nejsou zcela známé. Nicméně vzhledem k faktu, že je tato linie geneticky a fenotypově různorodá (fylogeograficky divergentní) s vysokou prevalencí, je díky těmto vlastnostem dobrou charakteristikou pro určování epidemiologie různých subtypů v různých částech světa. Podrobnou studii globální distribuce a geografické členění linie 4 a jejích sublinií uvádí publikace [15] u 3 366 kmenů M. tuberculosis komplex. Autoři uvádějí, že globální úspěch linie 4 je odrazem migrace v populaci člověka. V námi předkládané studii byly identifikovány dva příbuzenské/epidemilogické vztahy mezi pacienty s TBC způsobenou právě linií 4 a její subliníí Haarlem (obrázek 1). Identifikované MDR kmeny linie 4 byly zařazeny do sublinií LAM (2 ze 7 kmenů) a Haarlem (1 ze 7 kmenů), ostatní 3 pacienti byli linie 2 Východo-Asijské–Beijing. Tato data jsou opět v souladu s již dříve publikovanými pracemi. Řada autorů v nich uvádí, že sublinie LAM a sublinie Beijing je spojována s rezistencí na antibiotika [16, 17], proto je těmto kmenům potřeba věnovat zvláštní pozornost. Navíc Merker et al. (2015) uvádějí, že sublinie Beijing vznikla na Dálném východě, odkud se do celého světa rozšířila v několika vlnách, přičemž dochází k postupnému zvětšování velikosti populace této sublinie. Tato zjištění plně korespondují s našimi daty, kdy v ČR v letech 2013–2014 bylo v hlavním městě Praze identifikováno 10 neklastrovaných kmenů linie Beijing [14]. V předkládané studii z let 2014–2015 bylo identifikováno 37 kmenů Linie Beijing z toho 11 kmenů bylo identifikováno v hlavním městě Praze (viz graf 2, tab. 1).
I přes zásadní změny v léčbě TBC spolu s rozvojem zemí a s tím spojenou globalizací, se nedaří snižovat výskyt této nemoci. Naopak přibývá rezistentních kmenů mykobakterií. Tato studie obsahuje zásadní informace o shromažďování dat týkajících se rozšířenosti druhu M. tuberculosis za účelem určení linií (sublinií), pro potřeby identifikace faktorů rezistence a incidence TBC v národní i nadnárodní úrovni.
ZÁVĚR
Předkládáme výsledky studie identifikace a rozložení kmenů prevalujících na území České republiky v roce 2014 pomocí molekulární metody MIRU-VNTR druhu M. tuberculosis. Díky numerickému kódu a programu MIRU-VNTRplus lze kmeny a tedy i výsledky této studie velmi snadno porovnávat a použít pro srovnání v rámci různých studií dalších laboratoří. Toto uplatnění má význam především při identifikaci mikroepidemií a přeshraničního přenosu.
Při víceletém genotypování lze vysledovat původní zdroj infekce a její přenos. Pro prokázání mikroepidemií a nákaz mezi příbuznými je také důležitá spolupráce s epidemiology a klinickými lékaři. Předkládané výsledky mohou být podkladem pro další molekulárně epidemiologické studie doplněné dalšími metodami molekulární typizace.
Podpořeno MZ ČR – RVO („Státní zdravotní ústav – SZÚ, 75010330“).
Do redakce došlo dne 1. 3. 2019.
Adresa pro korespondenci:
RNDr. Martina Pinková
Státní zdravotní ústav
Šrobárova 48
100 00 Praha 10
Ing. Věra Dvořáková, Ph.D.
e-mail: vera.dvorakova@szu.cz
Sources
1. European Centre for Disease Prevention and Control. Tuberculosis surveillance and monitoring in Europe 2016, 17-bře-2016. [Online]. Dostupné na www: http://ecdc.europa.eu/en/publications-data/tuberculosis-surveillance-and-monitoring-europe-2016. [Viděno:15-dub-2019].
2. ÚZIS ČR. Tuberkulóza a respirační nemoci 2013. [Online]. Dostupné na www: https://www.uzis.cz/publikace/tuberkuloza-respiracni-nemoci-2013. [Viděno: 15-dub-2019].
3. Felipe Garcia D., Astudillo M. MIRU-VNTR genotyping of M. tuberculosis in a population of patients from Santiago De Cali, Colombia, 2013-2015, Biomedica, 2019;39.
4. Amlerova J, Bitar I, Hrabak J. Genotyping of Mycobacterium tuberculosis using whole genome sequencing. Folia Microbiol, 2018;63(5):537–545.
5. Supply P, et al. Proposal for Standardization of Optimized Mycobacterial Interspersed Repetitive Unit-Variable-Number Tandem Repeat Typing of Mycobacterium tuberculosis. J Clin Microbiol, 2006, 44(12):4498–4510.
6. Allix-Béguec C, et al. Evaluation and Strategy for Use of MIRU--VNTRplus, a Multifunctional Database for Online Analysis of Genotyping Data and Phylogenetic Identification of Mycobacterium tuberculosis Complex Isolates. J Clin Microbiol, 2008;46(8): 2692–2699.
7. Weniger T, et al. MIRU-VNTRplus: a web tool for polyphasic genotyping of Mycobacterium tuberculosis complex bacteria. Nucleic Acids Res, 2010;38, web server issue:W326–331.
8. WHO. Global tuberculosis report 2018 [online]. Dostupné na www: http://www.who.int/tb/publications/global_report/en/. [Viděno:16-dub-2019].
9. Garzelli C, et al. Impact of immigration on tuberculosis in a low-incidence area of Italy: a molecular epidemiological approach. Clin Microbiol Infect, 2010; 16(11):1691–1697.
10. Gagneux S, Small MP. Global phylogeography of Mycobacterium tuberculosis and implications for tuberculosis product development. Lancet Infect Dis, 2007;7(5):328–337.
11. Nebenzahl-Guimaraes H, et al. Genomic characterization of Mycobacterium tuberculosis lineage 7 and a proposed name: ‘Aethiops vetus’. Microb Genomics, 2016;2(6).
12. Coscolla M, Gagneux S. Consequences of genomic diversity in Mycobacterium tuberculosis. Semin Immunol, 2014;26(6):431–444.
13. Demay C, et al. SITVITWEB – a publicly available international multimarker database for studying Mycobacterium tuberculosis genetic diversity and molecular epidemiology. Infect Genet Evol J Mol Epidemiol Evol Genet Infect Dis, 2012;12(4):755–766.
14. Tietzeová E, Pinková M, Zemanová I. Molekulární epidemiologie tuberkulózy v hlavním městě Praha v letech 2013 a 2014. [Online]. Dostupné na www: https://www.prolekare.cz/casopisy/epidemiologie/2018-2-5/molekularni-epidemiologie-tuberkulozy-v-hlavnim-meste-praha-v-letech-2013-a-2014-104981. [Viděno: 18-dub-2019].
15. Stucki D, et al. Mycobacterium tuberculosis lineage 4 comprises globally distributed and geographically restricted sublineages. Nat Genet, 2016; 48(12):1535–1543.
16. Merker M. et al. Evolutionary history and global spread of the Mycobacterium tuberculosis Beijing lineage. Nat Genet, 2015;47(3):242–249.
17. Valcheva V, et al. Utility of New 24-Locus Variable-Number Tandem-Repeat Typing for Discriminating Mycobacterium tuberculosis Clinical Isolates Collected in Bulgaria. J Clin Microbiol, 2008;46(9):3005–3011.
Labels
Hygiene and epidemiology Medical virology Clinical microbiology
Article was published inEpidemiology, Microbiology, Immunology

2019 Issue 3-
All articles in this issue
- Vysoká prevalence virové hepatitidy C ve vyloučené komunitě brněnských Romů
- Výskyt a analýza případů onemocnění kampylobakteriózou v České republice v letech 1997–2017
- Molekulárně genetická studie mykobakteriálních kmenů prevalujících na území České republiky v roce 2014
- Antivirová adoptivní imunoterapie pomocí antigen specifických lymfocytů T u příjemců alogenního transplantátu krvetvorných buněk
- West Nile virus (linie 2) poprvé detekován v komárech v jižních Čechách: nová endemická oblast?
- Prevalence a role CCR5Δ32 v progresi onemocnění u HIV pozitivních pacientů v České republice
- Epidemiology, Microbiology, Immunology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Výskyt a analýza případů onemocnění kampylobakteriózou v České republice v letech 1997–2017
- Prevalence a role CCR5Δ32 v progresi onemocnění u HIV pozitivních pacientů v České republice
- Antivirová adoptivní imunoterapie pomocí antigen specifických lymfocytů T u příjemců alogenního transplantátu krvetvorných buněk
- Vysoká prevalence virové hepatitidy C ve vyloučené komunitě brněnských Romů
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

