-
Medical journals
- Career
Zapojení PIWI-interagujících RNA do procesů kancerogeneze prostřednictvím regulace genové exprese
Authors: S. Rybecká; K. Štítkovcová; P. Vychytilová-Faltejsková; O. Slabý
Authors‘ workplace: CEITEC – Středoevropský technologický institut, MU, Brno
Published in: Klin Onkol 2016; 29(6): 428-438
Category: Review
doi: https://doi.org/10.14735/amko2016428Overview
Východiska:
Za poslední desetiletí přibyly stovky studií, jež souhlasně prokázaly, že krátké nekódující RNA jsou slibnými diagnostickými, prognostickými a prediktivními biomarkery nádorových onemocnění. Významnou podskupinu těchto molekul představují RNA interagující s PIWI proteiny, označované jako piRNA. Tyto krátké RNA se podílejí na regulaci genové exprese na transkripční nebo post-transkripční úrovni, jejich hlavní úlohou je však epigenetické umlčování transpozibilních elementů LINE a SINE, čímž významně přispívají k udržení genomové stability. Dále se účastní důležitých buněčných procesů, jakými jsou gametogeneze, segregace chromozomů či sebeobnova kmenových buněk. Přestože byly piRNA poprvé detekovány v zárodečných buňkách, bylo zjištěno, že se vyskytují ve vysokých hladinách také v jiných lidských tkáních, přičemž jejich exprese vykazuje tkáňovou specificitu. První studie rovněž prokázaly změněný expresní profil piRNA u pacientů s nádorovými onemocněními. Funkce těchto molekul v procesu kancerogeneze však zatím zůstávají neobjasněné. Do popředí zájmu se v poslední době též dostávají volné cirkulující piRNA v tělních tekutinách, které nabízejí široké využití pro včasný záchyt nádorových onemocnění, predikci léčebné odpovědi či stanovení prognózy pacientů.Cíl:
Tento přehledový článek jako první v českém jazyce shrnuje dosavadní poznatky o biogenezi piRNA, o jejich strategii při umlčování transpozibilních elementů a dalších genů. Poskytuje rovněž ucelený přehled o piRNA s deregulovanou expresí u lidských nádorových onemocnění a klade důraz na jejich potenciální diagnostické a terapeutické využití.Klíčová slova:
PIWI-interagující RNA – piRNA – biogeneze – nádorové onemocnění – umlčování transpozonů – biomarkery – terapeutické cíleÚvod
Nádorová choroba je často vnímána jako onemocnění, jehož podstatou je kumulace genetických mutací, k nimž dochází v důsledku genomové nestability. Mezi typické biologické důsledky těchto mutací potom patří nadměrná proliferace nádorových buněk, rezistence vůči signálům zastavujícím buněčný cyklus, neomezený replikační potenciál, zvýšená invazivita či schopnost zakládat vzdálené metastázy. Jednotlivé mutace mohou dále indukovat angiogenezi, necitlivost k apoptotickým signálům, deregulaci buněčné energetiky, únik před imunitním systémem a zánětlivé procesy. Genomová nestabilita je tedy podmiňujícím znakem vzniku nádorového onemocnění [1]. Ze současných poznatků dále vyplývá, že jednou z příčin genomové nestability jsou rovněž deregulace na epigenetické úrovni. Jako epigenetické jsou označovány dědičné změny v genové expresi, které nesouvisejí se změnami v primárním genetickém kódu. Mezi hlavní mechanizmy, jež regulují genovou expresi na transkripční či post-transkripční úrovni, patří především metylace DNA, modifikace histonů a dále též regulace prostřednictvím nekódujících RNA [2,3]. Nedávné studie odhalily novou skupinu krátkých nekódujících RNA, které hrají významnou roli při udržování stability genomu. Úlohou těchto molekul je zejména umlčování transpozonů a regulace genové exprese na transkripční či post-transkripční úrovni. Od roku 2006 jsou známy pod názvem PIWI-interagující RNA (piRNA). V posledních 10 letech bylo prokázáno, že hladiny těchto nekódujících RNA jsou významně deregulovány rovněž u řady nádorových onemocnění.
PIWI-interagující RNA
PiRNA jsou krátké jednořetězcové RNA o délce 24–32 nukleotidů [4] hrající významnou roli při vývoji zárodečných buněk, sebeobnově kmenových buněk, a především pak při udržování genomové stability v důsledku umlčování transpozibilních elementů (TE) [5]. Tyto repetitivní elementy tvoří asi polovinu genomu člověka a hrají důležitou úlohu ve struktuře chromatinu, čímž ovlivňují i expresi přilehlých genů [6–8]. PiRNA byly pojmenovány na základě schopnosti vázat se na proteiny PIWI patřící do rodiny AGO (Argonaute) proteinů [9]. AGO proteiny se dělí na podrodiny AGO a PIWI. Lidské geny AGO1–4 jsou lokalizovány na chromozomu 1, lidská podrodina PIWI proteinů zahrnuje HIWI1, HIWI2, HIWI3 a HILI, které jsou kódovány geny na chromozomech 12, 11, 22 a 8 [10]. Argonautové proteiny jsou složeny ze dvou hlavních domén – N-terminální PAZ (Piwi-Argonaute-Zwille) domény a C-terminální PIWI domény. Délka PAZ čítá přibližně 120 aminokyselin, PIWI doména je dlouhá přibližně 400 aminokyselin. PIWI doména se dále skládá ze dvou částí, Mid/A a PIWI/B, přičemž PIWI/B katalyzuje aktivitu enzymu Slicer (RNáza H), který se účastní biogeneze piRNA. Mid doména váže 5’ fosfátové konce krátkých RNA a funguje jako kotva vazby RNA na AGO proteiny, zatímco 3’ konce krátkých RNA se vážou na doménu PAZ [11–13]. PIWI proteiny se účastní biogeneze piRNA, vývoje zárodečných buněk, gametogeneze a dalších buněčných pochodů. V důsledku mutací genů pro PIWI vznikají defekty v gametogenezi, dochází ke ztrátě zárodečné linie kmenových buněk, k pozastavení meiózy a blokacím spermatogeneze. Předpokládá se, že piRNA působí jako sekvenčně specifická vodítka PIWI proteinů umožňující regulaci exprese transpozonů a protein kódujících genů [14].
První studie zabývající se regulačním potenciálem piRNA u savců pochází z roku 2006. Aravin et al [15] popsali skupinu malých nekódujících RNA v samčích zárodečných buňkách u Mus musculus, jejichž délka se pohybovala v rozmezí 26–31 nukleotidů. Ve stejné době Girard et al [16] popsali skupinu malých nekódujících RNA, které se vážou na proteiny MIWI z rodiny myších PIWI proteinů a jsou hojně zastoupené ve varlatech myší. Jedna z prvních studií, která propojila piRNA s genomovou nestabilitou, se věnovala mutacím v genech pro Zucchini (ZUC) a Squash (SQU). Produkty těchto genů obsahují doménu homologickou s nukleázou a účastní se procesu RNA interference v zárodečných buňkách u Drosophily melanogaster. Mutantní samičky jsou sterilní a vykazují defekty v dorzoventrální orientaci v průběhu oogeneze. Mutace těchto genů rovněž souvisí se zvýšenou expresí HET-A a TART, dvou telomerově specifických transpozonů. Výsledky této studie naznačují, že k nadměrné aktivitě transpozonů může docházet v důsledku mutací v genech, jež se podílejí na vzniku a správném fungování piRNA [17]. Role piRNA při ochraně stability genomu byla popsána také u kuřat. S využitím sekvenování nové generace byly stanoveny expresní profily vybraných piRNA a několika genů asociovaných s piRNA v primordiálních zárodečných buňkách. Všechny analyzované geny byly u primordiálních zárodečných buněk vysoce exprimované. Zároveň bylo provedeno umlčení dvou známých genů souvisejících s dráhou piRNA – CIWI (chicken PIWI-like protein 1) a CILI (chicken PIWI-like protein 2). Po vyřazení těchto genů došlo ke změnám v expresi několika genů vázaných na piRNA a také ke zvýšenému výskytu dvouvláknových zlomů DNA v primordiálních zárodečných buňkách [18]. Významné funkce piRNA ve spojitosti s ochranou před transpozibilními elementy byly pozorovány rovněž v zárodečných buňkách myší, kde jsou piRNA schopné navodit metylaci DNA v oblasti CpG [19,20]. Kromě toho se tyto malé molekuly podílejí na udržování metylačního stavu histonů (H3K9me3) na repetitivních sekvencích typu LINEs v zárodečných buňkách [21]. Tato represe chromatinu zprostředkovaná pomocí piRNA cílí výhradně na elementy plné délky, které jsou aktivní. Toto zjištění odkrývá pozoruhodnou schopnost piRNA rozpoznávat aktivní elementy z množství fragmentů genomických transpozonů, které buňka obsahuje. Tyto studie a mnohé další potvrzují schopnost piRNA udržovat genomovou stabilitu prostřednictvím tlumení TE.
Přestože prvotní studie předpokládaly, že mechanizmy účinku piRNA jsou spojeny výhradně se zárodečnými buňkami, v roce 2012 Rajasethupathy et al [22] prokázali hojnou expresi piRNA o délce 28 nukleotidů v CNS zeje obrovského (Aplysia depilans). Tyto piRNA jsou lokalizované převážně v jádře a vyznačují se unikátním typem biogeneze. Výsledky studie naznačují, že piRNA se účastní dlouhodobých změn v neuronech, které vedou k udržování paměti. Zvýšená exprese piRNA byla následně popsána i v hipokampu myší, což naznačuje možnou účast těchto molekul na morfogenezi páteře prostřednictvím regulace exprese genů zodpovědných za její vývoj [23]. Nejnovější studie potvrdily, že piRNA jsou abundantně exprimované téměř ve všech lidských tkáních, přičemž jejich exprese vykazuje tkáňovou specificitu [24].
Biogeneze piRNA
Dosavadní studie naznačují, že v biogenezi, tedy v procesech, které vedou ke vzniku maturovaných molekul piRNA, jsou mezidruhové rozdíly a tyto signalizační dráhy jsou navíc odlišné od drah biogeneze miRNA a siRNA [25]. PiRNA původem vychází z mezigenových repetitivních elementů, tzv. piRNA klastrů. Klastry jsou součástí heterochromatinu bohatého na transpozony [26]. Na rozdíl od miRNA a siRNA jsou piRNA generované z jednovláknových prekurzorů, které nevyžadují RNázu III [27]. Dalším rozdílem je asociace s odlišnými členy podrodiny Argonautových proteinů. Zatímco piRNA interagují s podrodinou PIWI, miRNA a siRNA interagují primárně s proteiny podrodiny AGO. Molekuly piRNA se liší od miRNA a siRNA také svou délkou, která čítá o několik nukleotidů více. Mezi různými živočichy, dokonce i mezi příbuznými druhy, jsou však velké rozdíly v maturovaných sekvencích piRNA. Zatímco u Drosophily melanogaster a většiny obratlovců byly identifikovány piRNA o délce 24–32 nukleotidů, u Caenorhabditis elegans se vyskytují piRNA o délce 21 nukleotidů a označují se jako 21U-RNA [28]. U většiny organizmů jsou pak zpravidla popisovány dvě rozdílné dráhy biogeneze piRNA v závislosti na tom, zda se jedná o buňky somatické, nebo zárodečné. Zatímco v somatických buňkách jsou piRNA přepisovány pouze z jednoho vlákna, v zárodečných buňkách probíhá transkripce z obou vláken takzvaného duálního klastru piRNA. V případě zárodečných buněk pak probíhá kromě primární biogeneze i sekundární dráha zpracování piRNA (tzv. ping-pong cyklus).
Primární biogeneze piRNA
PiRNA původem vychází z mezigenových repetitivních elementů, tzv. piRNA klastrů. Klastry jsou součástí heterochromatinu bohatého na transpozony [29]. Tyto sekvence pokrývají velkou část genomu a jejich velikost se pohybuje od 100 kb výše [30]. V průběhu primární biogeneze jsou geny pro piRNA přepisovány pomocí RNA polymerázy II. Na procesu transkripce se rovněž podílí heterochromatinový protein HP1 zvaný Rhino, který se váže na některé lokusy genů kódujících piRNA, a dále na helikázu UAP56, s níž interaguje. Vlastní transkript je vázán proteinem UAP56 na vnitřní straně jaderné membrány a poté je přenesen pomocí RNA helikázy Vasa do cytoplazmatického váčku [31–33]. Zde probíhá sestřih prekurzorového transkriptu pomocí endonukleázy ZUC (analog u myší je PLD6 – fosfolipáza D6), jež je lokalizována na vnější mitochondriální membráně a generuje 5’ konec budoucí piRNA odštěpením fosfátového zbytku z uridinu za vzniku hydroxylového konce, na který se přednostně vážou PIWI proteiny. Vazba Argonautových PIWI/AUB proteinů (MILI a MIWI u Mus musculus) na piRNA je usnadněna díky chaperonovým proteinům HSP83 (heat shock protein 83) a SHU (Shutdown), které se následně odštěpí. Poté je sestřižen 3’ konec enzymem zvaným Trimmer, a to až k místu navázání proteinů PIWI/AUB. Takto upravený 3’ konec je následně metylován pomocí komplexu HEN1/Primet za vzniku 2’-O-metylovaného konce, který zajišťuje zvýšenou stabilitu zralých piRNA. Výstupem primární biogeneze je „primární“ piRNA (antisense vlákno) vázající proteiny PIWI/AUB (obr. 1). Takto vytvořený komplex vstupuje zpět do jádra, kde dochází k transkripčnímu umlčování transpozonů [32,34].
Image 1. Primární dráha biogeneze piRNA. Upraveno dle [4]. ![Primární dráha biogeneze piRNA. Upraveno dle [4].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/82349a5d12d76e3572b9f4a8184a4ff3.jpg)
Geny pro piRNA jsou přepisovány pomocí RNA polymerázy II za účasti proteinu Rhino a helikázy UAP56. Primární transkript je následně prostřednictvím helikázy Vasa přenesen z jádra do cytoplazmy, kde je sestřižen endonukleázou ZUC. Následně dochází k vazbě proteinů PIWI/AUB a sestřižení 3’ konce pomocí enzymu Trimmer, který je dále metylován metyltransferázou HEN1. Sekundární biogeneze piRNA
K sekundární biogenezi piRNA dochází především v zárodečných buňkách a pro iniciaci tohoto tzv. ping-pong cyklu je vyžadována primární piRNA (antisense vlákno) s navázanými proteiny PIWI/AUB, jež je produktem primární biogeneze. Ping-pong cyklus začíná navázáním komplexu „primární“ piRNA/AUB na komplementární vlákno transpozonálního (sense) transkriptu, čímž dochází k jeho umlčení a zároveň k tvorbě „sekundární“ piRNA. Vlákna sense a antisense piRNA jsou vázána prostřednictvím prvních 10 nukleotidů. V této fázi cyklu dochází k tvorbě 5’ konce vlákna piRNA pomocí proteinu AUB, který funguje jako enzym Slicer (opět s pomocí SHU a HSP83), a navázání proteinu AGO3 v místě 10. nukleotidu obsahujícího adenin. Na odstranění 5’ konce po sestřihu vlákna pomocí AUB se pravděpodobně podílí protein SHU. Následně dochází k sestřihu 3’ konce pomocí exonukleázy Trimmer a jeho metylaci pomocí HEN1. Metylovaný fragment v komplexu s AGO3 váže antisense transkript pro piRNA a zajišťuje jeho sestřih. Takto upravený řetězec opětovně váže AUB proteiny, podstupuje sestřih 3’ konce a jeho metylaci, čímž dochází k uzavření celého cyklu a amplifikaci vznikajících piRNA (obr. 2) [31,32,35].
Image 2. Sekundární dráha biogeneze piRNA (ping-pong cyklus). Upraveno dle [4]. ![Sekundární dráha biogeneze piRNA (ping-pong cyklus). Upraveno dle [4].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/6f21da5f13560957bb15ea211bbc8d90.jpg)
Cyklus začíná vazbou primárního komplexu piRNA/AUB na komplementární vlákno transpozonálního transkriptu. Toto vlákno je následně štěpeno a vzniká sekundární piRNA vázající protein AGO3, jež je opět sestřižena pomocí enzymu Trimmer a metylována za účasti metyltransferázy HEN1. Sekundární piRNA poté váže antisense transkript pro piRNA a zajišťuje jeho sestřih. Celý cyklus vede k amplifikaci vznikajících piRNA. Mechanizmy regulace genové exprese prostřednictvím piRNA
Za nejpodstatnější funkci komplexů PIWI/piRNA je považována schopnost represe mobilních elementů DNA. Tento předpoklad je podpořený zejména faktem, že piRNA obsahují komplementární sekvence vůči transpozonům, jež jsou proto považovány za hlavní cíl jejich aktivity. Tato skutečnost byla opakovaně prokázána nezávislými studiemi napříč několika živočišnými druhy, jakými jsou Drosophila melanogaster [27], Mus musculus [15] či Danio rerio [25]. Zajímavé je, že osud komplexu PIWI/piRNA (jeho konkrétní lokalizace v jádře nebo v cytoplazmě a možný mechanizmus účinku umlčování) záleží na podskupině proteinů z rodiny PIWI, se kterou je piRNA asociována. Pokud je piRNA asociována s proteiny PIWI, nachází se komplex v jádře, a může tak docházet k umlčování genové exprese na transkripční úrovni. Pokud je piRNA asociována s proteiny AUB, bude se tento komplex nacházet v perinukleárním prostoru [27]. Podobných závěrů bylo dosaženo v případě myšího modelu, kdy se zralé piRNA asociované s proteiny MILI nacházely v cytoplazmě, zatímco komplexy MIWI/piRNA byly translokovány do jádra [19,36]. Nedávné studie pak prokázaly, že k umlčování TE a regulaci exprese genů prostřednictvím piRNA může docházet především cestou metylace DNA, v důsledku modifikací histonů a chromatinu, a v neposlední řadě též na základě post-transkripčního umlčování genové exprese (obr. 3).
Image 3. Mechanizmy regulace genové exprese prostřednictvím piRNA. 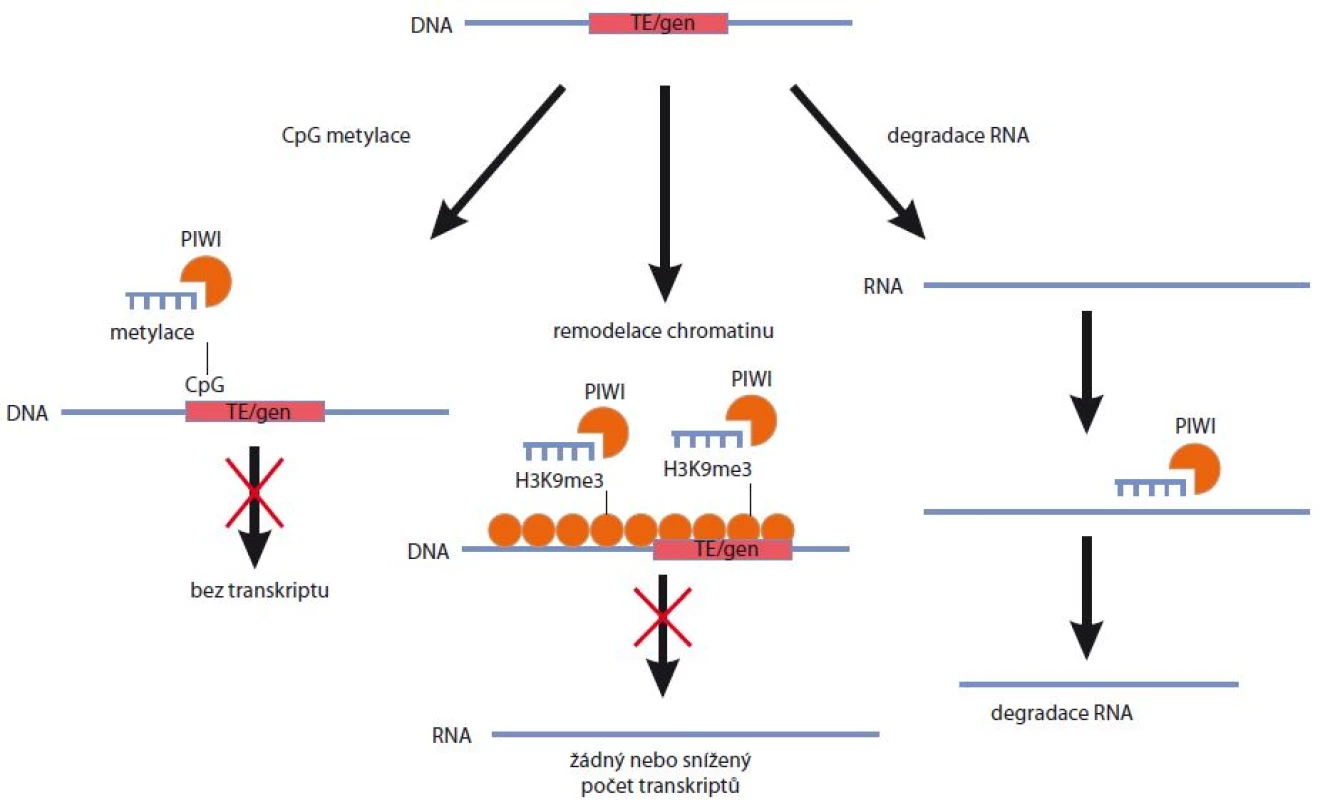
K umlčování transpozibilních elementů (TE) a regulaci genové exprese může docházet prostřednictvím metylace CpG oblastí na DNA, v důsledku remodelace chromatinu či na základě post-transkripčního umlčování vazbou komplexů PIWI/piRNA ke komplementárním sekvencím mRNA. Metylace DNA jako mechanizmus umlčování transpozonů
Kovalentní modifikace DNA prostřednictvím metylace cytozinových zbytků je dědičný, avšak reverzibilní proces. Metylace DNA se podílí na regulaci genové exprese, inaktivaci chromozomu X, genomickém imprintingu a na početných modifikacích chromatinu. Nedávné studie prokázaly zásadní roli piRNA při metylaci mobilních elementů v zárodečných a somatických buňkách.
Kuramochi-Miyagawa et al [20] prokázali spojitost mezi expresí PIWI/piRNA a umlčováním TE v zárodečných buňkách myší, kdy v důsledku delece genů pro myší PIWI proteiny MILI a MIWI2 nedocházelo k tvorbě komplexů MILI/MIWI2/piRNA a následné metylaci TE, jež by utlumila aktivitu těchto mobilních elementů. K podobným výsledkům dospěla další studie provedená na myších s deletovaným genem TDRD1 (protein s doménou Tudor 1) [37]. Autoři zde identifikovali protein TDRD1, jenž je asociovaný s komplexem MILI/piRNP (PIWI ribonukleoprotein) u dospělých samců během spermatogeneze. Stejně jako u mutantů MILI, tak i u myší s chybějícím TDRD1 byla prokázána snížená metylace DNA vedoucí ke zvýšené aktivitě transpozonu L1.
Metylace DNA prostřednictvím komplexů PIWI/piRNA byla prokázána i v CNS zeje obrovského [22]. Expresní profilování malých RNA v mozku dospělých jedinců prokázalo přítomnost hojně se vyskytujících krátkých RNA o délce 28 nukleotidů. Tyto piRNA byly senzitivní na serotonin, jenž je důležitým neurotransmiterem a významně se podílí na mechanizmech dlouhodobé paměti. Komplex PIWI/piRNA v tomto případě ulehčoval na serotoninu závislou metylaci CpG ostrovu v oblasti promotoru CREB2 (cAMP response element binding protein 2), který je inhibitorem dlouhodobé paměti.
Z výše uvedených studií vyplývá, že metylace DNA prostřednictvím komplexu PIWI/piRNA je mezidruhově rozmanitá a představuje důležitý nástroj epigenetické regulace jak v zárodečných, tak somatických buňkách organizmů.
Vliv histonových modifikací a struktury chromatinu na expresi transpozonů a okolních genů
PIWI proteiny v asociaci s piRNA jsou zapojené nejen do metylace DNA, ale také do regulace histonových modifikací, které vedou rovněž k umlčování mobilních elementů. K tomuto umlčování TE dochází zpravidla v důsledku metylace lysinu 9, jenž je součástí histonu 3 (H3K9me3). Tato histonová modifikace vede ke vzniku heterochromatinu a ovlivňuje nejen transkripci vlastních mobilních elementů, ale též expresi přilehlých genů [38].
Le Thomas et al [39] dále prokázali, že přestože je asociace PIWI proteinů s cílovou strukturou chromatinu pouze dočasná, může vést k trvalému umlčení exprese genů či TE v důsledku zvýšené metylace jednotlivých histonů a vytvoření transkripčně inaktivního heterochromatinu. Podobná situace byla popsána i v případě Caenorhabditis elegans, kdy přítomnost komplexu PRG-1/21U RNA umožnila vznik heterochromatinu [40,41]. Zajímavé přitom je, že vazba komplexu PRG-1/21U RNA na cílové oblasti chromatinu hraje klíčovou roli pouze v iniciaci celého procesu, zatímco následná kontinuální represe přetrvávající napříč generacemi závisí na jaderných proteinech HRDE1/WAGO-9 z Argonautové rodiny a histon-metyltransferázách SET-25 a SET32 [42].
Další studie z roku 2014 na myších prokazuje, že přítomnost piRNA je nezbytná pro udržení vysoké úrovně represivní histonové modifikace H3K9me3 u elementů typu LINE v zárodečných buňkách. Tato chromatinová represe zprostředkovaná piRNA je nejvíce selektivní, neboť dochází pouze k umlčování elementů plné délky, které jsou z rodin LINE a jsou schopné aktivní transpozice [21].
Regulace exprese genů a mobilních elementů na post-transkripční úrovni
K post-transkripčnímu umlčování genové exprese dochází zpravidla prostřednictvím vazby piRNA na cílové sekvence retrotranspozonů, jež jsou součástí 3’ nepřekládaných oblastí téměř 30 % myších či lidských mRNA [43]. Zároveň bylo prokázáno, že piRNA mohou přímo regulovat také geny s retrotranspozonní sekvencí v 5’ nepřekládané oblasti [44].
Příkladem post-transkripčního umlčení exprese genů je regulace mRNA prostřednictvím piRNA derivovaných z pseudogenů. Tyto oblasti mohou produkovat tzv. trans-elementy – piRNA, které regulují mRNA z funkčních genů. PiRNA mohou být produkované nejen z nefunkčních pseudogenů, ale také z protismyslného vlákna funkčního genu. V takovém případě mluvíme o cis-přirozených protismyslných transkriptech, které rovněž mohou produkovat piRNA a následně regulovat přepis cílových genů [45]. Příkladem trans regulace z pseudogenu je regulace genu Stellate, který se vyskytuje na chromozomu X. Exprese tohoto genu je u Drosophily melanogaster potlačena v důsledku vazby piRNA, jež je produktem pseudogenu vyskytujícího se na chromozomu Y a označovaného jako SU nebo STE. V případě delece tohoto pseudogenu dochází k přepisu genu pro Stellate, akumulaci cílového proteinu a vytvoření krystalické struktury v spermatocytech, což vede k neplodnosti [46]. Regulace genové exprese na post-transkripční úrovni prostřednictvím piRNA byla rovněž popsána i v případě sexuální determinace bource morušového. Tento organizmus má ptačí typ sexuální determinace ZW (samice ZW, samci ZZ) a jediná piRNA derivovaná z pohlavního chromozomu W má feminizující účinek na celý organizmus [47].
Nedávná studie na dvou kmenech Drosophily melanogaster prokázala, že začlenění TE do euchromatinového lokusu vede k tvorbě nového dvouvláknového klastru piRNA, z něhož mohou následně vznikat funkční piRNA zamezující expanzi TE prostřednictvím post-transkripční regulace exprese těchto mobilních elementů. Zároveň bylo potvrzeno, že produkce malých RNA vyvolaná inzercí TE může rovněž snižovat expresi přilehlých genů [48]. Tato studie je dalším důkazem toho, že malé RNA asociované s TE hrají významnou roli v regulaci genové exprese, a to nejen u zárodečných buněk.
PIWI proteiny a kancerogeneze
První zmínka o souvislosti mezi proteiny z podrodiny PIWI a nádory se týkala proteinu HIWI, jehož exprese byla prokázána v nádorové tkáni seminomů, avšak nikoliv v příslušné nenádorové tkáni [49]. Na tuto studii navázaly další práce dokumentující deregulovanou expresi proteinu HILI u nádorů prsu, vaječníků a děložního hrdla, zvýšené hladiny proteinu HIWI u nádorů jater a u pacientů se sarkomy či pozměněnou expresi proteinů HIWI a HIWI2 u pacientů s nádory žaludku [50].
Další klinické studie naznačují prognostický potenciál některých PIWI proteinů. U gliomů byla popsána pozitivní korelace mezi expresí proteinu HIWI a gradem onemocnění, přičemž pacienti s vysokou hladinou tohoto proteinu měli významně horší prognózu [51]. Následující studie dokumentují úzkou souvislost mezi hladinami proteinu HIWI a vybranými klinicko-patologickými znaky pacientů s nádory slinivky břišní, tlustého střeva a konečníku, endometria, jícnu, jater a žaludku [50]. V případě pacientů s nádory žaludku byla prokázána přímá korelace mezi expresí proteinů HIWI, HILI, PIWIL3 i HIWI2 a klinickým stadiem onemocnění. Zároveň bylo zjištěno, že pacienti se zvýšenými hladinami těchto proteinů mají kratší dobu celkového přežívání [52].
Konkrétní zapojení PIWI proteinů do procesů kancerogeneze zatím není objasněno, avšak na základě dosavadních studií na animálních modelech Mus Musculus a Drosophila melanogaster [19,53] existuje několik možných vysvětlení. Jednou z hypotéz je, že zvýšená exprese těchto proteinů přispívá ke vzniku a progresi nádorových onemocnění prostřednictvím epigenetického umlčování nádorově supresorových genů na transkripční úrovni. Další možností je pak jejich zapojení do umlčování TE [54] či regulace genové exprese prostřednictvím metylace histonů, o čemž svědčí také jejich společný výskyt se skupinou proteinů Polycomb, které jsou známé svou schopností remodelovat chromatin [55]. PIWI proteiny mohou dále fungovat jako aktivátory transkripce prostřednictvím zavádění euchromatických histonových modifikací do oblastí heterochromatinu [56]. V souladu s pozorovanými epigenetickými funkcemi komplexů PIWI/piRNA bylo dále popsáno, že exprese proteinů HIWI koreluje s mírou metylace u pacientů se sarkomy, přičemž snížené hladiny tohoto proteinu byly nalezeny ve tkáních s globální hypometylací DNA a souvisely s prognosticky příznivějším onemocněním [57].
Deregulovaná exprese piRNA u nádorových onemocnění
Vzhledem ke skutečnosti, že se piRNA významně podílejí na regulaci exprese genů a udržování genomové stability, předpokládá se jejich zapojení do procesů kancerogeneze [58]. Přestože v souvislosti s nádory byly nejprve zkoumány pouze jednotlivé PIWI proteiny, prvotní studie již prokázaly význam konkrétních piRNA v patogenezi nádorových onemocnění [50]. Tyto molekuly se podobně jako miRNA mohou podílet na regulaci exprese klíčových nádorových supresorů či onkogenů, a to především prostřednictvím vazby k cílovým mRNA, jež v 3’ nepřekládané oblasti obsahují transpozonální sekvence. Kromě toho narušení fyziologických hladin piRNA, které za normálních okolností potlačují aktivitu TE, může ulehčit retrotranspozici, zvýšit genomovou nestabilitu, a přispět tak ke vzniku či progresi nádorového onemocnění. Přestože dosavadní studie dokumentují celkovou deregulaci hladin piRNA v nádorové tkáni, přesné funkce těchto molekul nebyly dosud objasněny. Zároveň není známo, zda k této deregulaci dochází v důsledku přítomnosti nádorových buněk, nebo ještě před samotným vznikem onemocnění.
Jednou z nejvíce studovaných piRNA u pacientů s nádory je piR-823. Snížené hladiny této piRNA byly zaznamenány u nádorů žaludku [59] či u pacientů s renálním karcinomem [60], přičemž umělé navýšení hladiny piR-823 in vitro v buněčné linii odvozené od nádoru žaludku vedlo k významné inhibici růstu nádorových buněk. Nádorově supresorový efekt této piRNA byl následně potvrzen in vivo s využitím myšího modelu [61]. K opačným výsledkům dospěla studie zabývající se expresí piR-823 u mnohočetného myelomu. Hlavní příčinou rozvoje tohoto onemocnění je hypermetylace DNA vedoucí k potlačení transkripce nádorově supresorových genů. V tomto případě byla exprese piR-823 zvýšená jak u pacientů s mnohočetným myelomem, tak ve stabilních buněčných liniích odvozených od tohoto typu nádoru. Zvýšené hladiny zároveň pozitivně korelovaly s klinickým stadiem onemocnění. Umlčení piR-823 pomocí syntetických oligonukleotidů vedlo k zástavě buněčného cyklu a k expresi proteinů indukujících apoptózu. Autoři této studie tedy předpokládají, že piR-823 funguje jako významný onkogen u pacientů s mnohočetným myelomem, a mohla by proto v budoucnu sloužit jako slibný terapeutický cíl [62]. Tyto výsledky naznačují, že jednotlivé piRNA mohou podobně jako miRNA vystupovat v procesu kancerogeneze buď jako onkogeny, nebo jako nádorové supresory v závislosti na typu nádorového onemocnění a okolním mikroprostředí.
V případě hepatocelulárního karcinomu byla identifikována piR-Hep1, jež byla ve zvýšené míře exprimována v nádorové tkáni necelých 50 % pacientů a jejíž umlčení v nádorových liniích vedlo k inhibici životaschopnosti buněk a snížení jejich pohyblivosti [63]. Cheng et al [64] se ve své práci zabývají piR-651, jež je ve zvýšené míře exprimována u pacientů s nádory žaludku, tračníku, prsu a plic, a funguje tedy pravděpodobně jako významný nádorový onkogen. V nádorové tkáni močového měchýře byla identifikována piR-60152, jejíž exprese byla významně snížená u pacientů s tímto onemocněním. Zvýšení hladin této piRNA ve stabilních buněčných liniích pomocí tranzientní transfekce vedlo k snížené proliferační kapacitě, indukci apoptózy a k poklesu klonogenního potenciálu buněk [65].
Deregulovaná exprese piRNA byla popsána i u pacientů s nádory prsu. Huang et al [66] identifikovali zvýšenou expresi pěti piRNA v nádorové tkáni pacientů, přičemž piR-4987 byla asociována s přítomností metastáz v regionálních lymfatických uzlinách, a mohla by proto sloužit jako slibný prognostický marker. Nedávné studie dále prokázaly, že přibližně 40 % piRNA exprimovaných u pacientů s nádory prsu má intragenový původ. Předpokládá se tedy, že transkripty některých genů by mohly sloužit jako prekurzory piRNA a zároveň by jejich exprese mohla být prostřednictvím těchto piRNA zpětně regulována. Dalším zajímavým zjištěním je pak skutečnost, že transkripce genů pro některé piRNA je závislá na estrogenu a na transkripčním faktoru Erβ. Exprese tří piRNA (DQ597945, DQ570994 a DQ598651) je odlišná u buněk s estrogenovým receptorem a u buněk bez tohoto receptoru. Tyto výsledky naznačují, že by piRNA mohly sloužit jako významné prediktivní biomarkery u pacientů s nádory prsu [67].
Expresním profilováním piRNA v somatických nádorových buňkách se zabývala studie Martineze et al [68]. Z popisovaných 20 831 piRNA bylo v nemaligních somatických buňkách exprimováno 273 piRNA (1,3 %), zatímco v nádorových buňkách bylo detekováno 522 různých piRNA (2,5 %), což je téměř 2krát více. Na základě exprese konkrétních piRNA pak bylo možné spolehlivě odlišit nádorovou tkáň od nenádorové sliznice, zároveň pak byla prokázána vysoká tkáňová specificita některých piRNA.
Tabulka 1 shrnuje výsledky několika dosavadních studií, jež popisují významnou deregulaci konkrétních piRNA u jednotlivých onkologických diagnóz. Tyto kandidátní piRNA by se po dalších validacích mohly stát slibnými diagnostickými, prognostickými či prediktivními biomarkery u pacientů s nádorovými onemocněními.
Table 1. Přehled piRNA s deregulovanou expresí v nádorové tkáni pacientů s nádorovým onemocněním. 
RLU – regionální lymfatické uzliny, DNMT3A/B – DNA metyltransferáza 3A/B Cirkulující piRNA jako nové nádorové biomarkery
Do popředí zájmu se v posledních letech dostaly volné cirkulující piRNA v tělních tekutinách, které nabízejí široké využití pro včasný neinvazivní záchyt nádorových onemocnění [69] a také pro predikci léčebné odpovědi či stanovení prognózy pacientů. Bylo prokázáno, že cirkulující piRNA, podobně jako miRNA, jsou v tělních tekutinách vysoce stabilní a mají schopnost odolávat celé řadě i extrémních fyzikálních vlivů uměle vytvořených v laboratorních podmínkách [70].
Jedním z míst výskytu piRNA je např. nebuněčná frakce lidských slin. Bahn et al [71] detekovali 109 různých piRNA přítomných v lidských slinách, přičemž nejvíce exprimovanou byla piR-018570. Cui et al [72] následně analyzovali expresi piR-823 a piR-651 v periferní krvi pacientů s nádorem žaludku. Obě piRNA byly ve zvýšené míře detekovány v krevním séru zdravých kontrol a mohly by v budoucnu sloužit jako nové markery pro neinvazivní diagnostiku tohoto onemocnění. Autoři zároveň uvádějí, že piR-823 a piR-651 jsou citlivějšími indikátory karcinomu žaludku než miR-106a a miR-17, které byly dosud popisovány jako vhodné diagnostické biomarkery těchto nádorů [73]. Na základě exprese piR-823 a piR-651 bylo dále možné zachytit časná stadia onemocnění, a to s vyšší senzitivitou a specificitou, než jakou nabízejí současně používané markery CEA (karcinoembryonální antigen) a CA 19-9 [74].
PiRNA jako nové terapeutické cíle
Identifikace interakčních partnerů piRNA a pochopení principu těchto interakcí nejenže významně rozšíří poznání v oblasti nádorové biologie, ale představuje potenciálně velice slibný terapeutický přístup u nádorových onemocnění. Použití synteticky vyrobených piRNA pro blokaci syntézy cílových proteinů prostřednictvím vazby na mRNA bez nutnosti využití enzymu Dicer je zvažováno jako potenciální terapeutická aplikace. Předpokládá se, že jednou z výhod piRNA jako terapeutického cíle je vysoká specificita (na rozdíl od miRNA). V léčbě by mohly být rovněž využívány PIWI antigeny pro doručení léčiv do míst nádoru [75]. Mezi další výhody piRNA jako potenciálního terapeutického cíle patří účast na degradaci RNA na post-transkripční úrovni a schopnost metylace DNA vedoucí k umlčování genů a inhibici exprese určitých onkogenů [76].
Závěr
Nedávno objevená třída krátkých nekódujících RNA označovaných jako piRNA se stává předmětem výzkumu v mnoha oblastech biologie, vč. biologie nádorové. Tento přehledový článek shrnuje základní poznatky týkající se biogeneze piRNA a mechanizmů jejich funkčního uplatnění v rámci udržování stability genomu. Skutečnost, že piRNA jsou schopné epigeneticky kontrolovat expresi genů a jsou hojně a specificky exprimované ve všech lidských tkáních, jsou hlavními důvody, pro které se staly předmětem biomedicínského výzkumu. V současnosti již víme, že hladiny některých piRNA jsou v nádorové tkáni významně deregulované v porovnání s tkání nenádorovou. Zatím však není zcela jasné, zda tato deregulace vede ke vzniku a progresi onemocnění nebo je spíše důsledkem změn, jež kancerogenezi provázejí. Zatímco některé studie poukazují na skutečnost, že piRNA umlčují expresi nádorových supresorů na transkripční i post-transkripční úrovni, a tím přispívají ke vzniku nádoru, jiné studie předpokládají, že v důsledku globálně snížených hladin piRNA v organizmu dochází k nedostatečné inhibici transkripce některých onkogenů, jejichž exprese je následně zodpovědná za rozvoj nádorového onemocnění. Lepší porozumění biologické funkce těchto molekul by v budoucnosti mohlo vést k jejich možnému využití v terapii nádorových onemocnění a ke zdokonalení molekulární typizace nádorů. Velkým příslibem do budoucna jsou rovněž volné cirkulující piRNA přítomné v tělních tekutinách, jež by mohly představovat slibné biomarkery pro časnou neinvazivní diagnostiku a zároveň by mohly přispět k lepší stratifikaci pacientů na základě prognózy či předpokládané léčebné odpovědi.
Poděkování
Autoři děkují Mgr. Andrejovi Bešše za přípravu ilustrací pro publikaci.
Výsledky tohoto výzkumu byly získány v rámci projektu CEITEC 2020 (LQ1601) za finančního přispění Ministerstva školství, mládeže a tělovýchovy České republiky v rámci účelové podpory z prostředků Národního programu udržitelnosti II.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
doc. RNDr. Ondřej Slabý, Ph.D.
CEITEC – Středoevropský technologický institut
Masarykova univerzita
Kamenice 753/5
625 00 Brno
e-mail: ondrej.slaby@ceitec.muni.cz
Obdrženo: 23. 11. 2016
Přijato: 5. 12. 2016
Sources
1. Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell 2011; 144 (5): 646–674. doi: 10.1016/j.cell. 2011.02.013.
2. Sabin LR, Delás MJ, Hannon GJ. Dogma derailed: the many influences of RNA on the genome. Mol Cell 2013; 49 (5): 783–794. doi: 10.1016/j.molcel.2013.02.010.
3. Baylin SB, Jones PA. A decade of exploring the cancer epigenome – biological and translational implications. Nat Rev Cancer 2011; 11 (10): 726–734. doi: 10.1038/nrc3130.
4. Luteijn MJ, Ketting RF. PIWI-interacting RNAs: from generation to transgenerational epigenetics. Nat Rev Genet 2013; 14 (8): 523–534. doi: 10.1038/nrg3495.
5. Sana J, Faltejskova P, Svoboda M et al. Novel classes of non-coding RNAs and cancer. J Transl Med 2012; 10 : 103. doi: 10.1186/1479-5876-10-103.
6. Chinwalla AT, Cook LL, Delehaunty KD et al. Initial sequencing and comparative analysis of the mouse genome. Nature 2002; 420 (6915): 520–562.
7. Slotkin RK, Martienssen R. Transposable elements and the epigenetic regulation of the genome. Nat Rev Genet 2007; 8 (4): 272–285.
8. Goodier JL, Kazazian HH. Retrotransposons revisited: the restraint and rehabilitation of parasites. Cell 2008; 135 (1): 23–35. doi: 10.1016/j.cell.2008.09.022.
9. Lim RS, Kai T. A piece of the pi (e): the diverse roles of animal piRNAs and their PIWI partners. Semin Cell Dev Biol 2015; 47–48 : 17–31. doi: 10.1016/j.semcdb.2015.10.025.
10. Höck J, Meister G. The Argonaute protein family. Genome Biol 2008; 9 (2): 210. doi: 10.1186/gb-2008-9-2-210.
11. Kim VN, Han J, Siomi MC. Biogenesis of small RNAs in animals. Nat Rev Mol Cell Biol 2009; 10 (2): 126–139. doi: 10.1038/nrm2632.
12. Parker JS, Barford D. Argonaute: a scaffold for the function of short regulatory RNAs. Trends Biochem Sci 2006; 31 (11): 622–630.
13. Zhai L, Wang L, Teng F et al. Argonaute and argonaute-bound small RNAs in stem cells. Int J Mol Sci 2016; 17 (2): 208. doi: 10.3390/ijms17020208.
14. Ku HY, Lin H. PIWI proteins and their interactors in piRNA biogenesis, germline development and gene expression. Natl Sci Rev 2014; 1 (2): 205–218.
15. Aravin A, Gaidatzis D, Pfeffer S et al. A novel class of small RNAs bind to MILI protein in mouse testes. Nature 2006; 442 (7099): 203–207.
16. Girard A, Sachidanandam R, Hannon GJ et al. A germline-specific class of small RNAs binds mammalian Piwi proteins. Nature 2006; 442 (7099): 199–202.
17. Pane A, Wehr K, Schüpbach T. Zucchini and squash encode two putative nucleases required for rasiRNA production in the Drosophila germline. Dev Cell 2007; 12 (6): 851–862.
18. Rengaraj D, Lee S, Park T et al. Small non-coding RNA profiling and the role of piRNA pathway genes in the protection of chicken primordial germ cells. BMC Genomics 2014; 15 : 757. doi: 10.1186/1471-2164-15-757.
19. Aravin AA, Sachidanandam R, Bourc’his D et al. A piRNA pathway primed by individual transposons is linked to de novo DNA methylation in mice. Mol Cell 2008; 31 (6): 785–799. doi: 10.1016/j.molcel.2008.09.003.
20. Kuramochi-Miyagawa S, Watanabe T, Gotoh K et al. DNA methylation of retrotransposon genes is regulated by Piwi family members MILI and MIWI2 in murine fetal testes. Genes Dev 2008; 22 (7): 908–917. doi: 10.1101/gad.1640708.
21. Pezic D, Manakov SA, Sachidanandam R et al. piRNA pathway targets active LINE1 elements to establish the repressive H3K9me3 mark in germ cells. Genes Dev 2014; 28 (13): 1410–1428. doi: 10.1101/gad.240895.114.
22. Rajasethupathy P, Antonov I, Sheridan R et al. A role for neuronal piRNAs in the epigenetic control of memory-related synaptic plasticity. Cell 2012; 149 (3): 693–707. doi: 10.1016/j.cell.2012.02.057.
23. Lee EJ, Banerjee S, Zhou H et al. Identification of piRNAs in the central nervous system. RNA 2011; 17 (6): 1090–1099. doi: 10.1261/rna.2565011.
24. Yan Z, Hu HY, Jiang X et al. Widespread expression of piRNA-like molecules in somatic tissues. Nucleic Acids Res 2011; 39 (15): 6596–6607. doi: 10.1093/nar/gkr298.
25. Houwing S, Kamminga LM, Berezikov E et al. A role for Piwi and piRNAs in germ cell maintenance and transposon silencing in Zebrafish. Cell 2007; 129 (1): 69–82.
26. Khurana JS, Theurkauf W. piRNAs, transposon silencing, and Drosophila germline development. J Cell Biol 2010; 191 (5): 905–913. doi: 10.1083/jcb.201006034.
27. Brennecke J, Aravin AA, Stark A et al. Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell 2007; 128 (6): 1089–1103.
28. Goh WS, Seah JW, Harrison EJ et al. A genome-wide RNAi screen identifies factors required for distinct stages of C. elegans piRNA biogenesis. Genes Dev 2014; 28 (7): 797–807. doi: 10.1101/gad.235622.113.
29. Khurana JS, Theurkauf W. piRNAs, transposon silencing, and Drosophila germline development. J Cell Biol 2010; 191 (5): 905–913. doi: 10.1083/jcb.201006034.
30. Ishizu H, Siomi H, Siomi MC. Biology of PIWI-interacting RNAs: new insights into biogenesis and function inside and outside of germlines. Genes Dev 2012; 26 (21): 2361–2373. doi: 10.1101/gad.203786.112.
31. Czech B, Hannon GJ. One loop to rule them all: the ping-pong cycle and piRNA-guided silencing. Trends Biochem Sci 2016; 41 (4): 324–337. doi: 10.1016/j.tibs.2015.12.008.
32. Luteijn MJ, Ketting RF. PIWI-interacting RNAs: from generation to transgenerational epigenetics. Nat Rev Genet 2013; 14 (8): 523–534. doi: 10.1038/nrg3495.
33. Ketting RF. The many faces of RNAi. Dev Cell 2011; 20 (2): 148–161. doi: 10.1016/j.devcel.2011.01.012.
34. Guzzardo PM, Muerdter F, Hannon GJ. The piRNA pathway in flies: highlights and future directions. Curr Opin Genet Dev 2013; 23 (1): 44–52. doi: 10.1016/j.gde.2012.12.003.
35. Siomi MC, Sato K, Pezic D et al. PIWI-interacting small RNAs: the vanguard of genome defence. Nat Rev Mol Cell Bio 2011; 12 (4): 246–258. doi: 10.1038/nrm3089.
36. Kuramochi-Miyagawa S, Kimura T, Yomogida K et al. Two mouse piwi-related genes: miwi and mili. Mech Dev 2001; 108 (1–2): 121–133.
37. Reuter M, Chuma S, Tanaka T et al. Loss of the Mili-interacting tudor domain-containing protein-1 activates transposons and alters the Mili-associated small RNA profile. Nat Struct Mol Biol 2009; 16 (6): 639–646. doi: 10.1038/nsmb.1615.
38. Sienski G, Dönertas D, Brennecke J. Transcriptional silencing of transposons by Piwi and maelstrom and its impact on chromatin state and gene expression. Cell 2012; 151 (5): 964–980. doi: 10.1016/j.cell.2012.10.040.
39. Le Thomas A, Rogers AK, Webster A et al. Piwi induces piRNA-guided transcriptional silencing and establishment of a repressive chromatin state. Genes Dev 2013; 27 (4): 390–399. doi: 10.1101/gad.209841.112.
40. Bagijn MP, Goldstein LD, Sapetschnig A et al. Function, targets, and evolution of caenorhabditis elegans piRNAs. Science 2012; 337 (6094): 574–578. doi: 10.1126/science.1220952.
41. Ashe A, Sapetschnig A, Weick EM et al. piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell 2012; 150 (1): 88–99. doi: 10.1016/j.cell.2012.06.018.
42. Shirayama M, Seth M, Lee HC et al. piRNAs initiate an epigenetic memory of nonself RNA in the C. elegans germline. Cell 2012; 150 (1): 65–77. doi: 10.1016/j.cell. 2012.06.015.
43. Faulkner GJ, Kimura Y, Daub CO et al. The regulated retrotransposon transcriptome of mammalian cells. Nat Genet 2009; 41 (5): 563–571. doi: 10.1038/ng.368.
44. Lim AK, Lorthongpanich C, Chew TG et al. The nuage mediates retrotransposon silencing in mouse primordial ovarian follicles. Development 2013; 140 (18): 3819–3825. doi: 10.1242/dev.099184.
45. Watanabe T, Lin H. Posttranscriptional regulation of gene expression by Piwi proteins and piRNAs. Mol Cell 2014; 56 (1): 18–27. doi: 10.1016/j.molcel.2014.09.012.
46. Kotelnikov RN, Klenov MS, Rozovsky YM et al. Peculiarities of piRNA-mediated post-transcriptional silencing of Stellate repeats in testes of Drosophila melanogaster. Nucleic Acids Res 2009; 37 (10): 3254–3263. doi: 10.1093/nar/gkp167.
47. Kiuchi T, Koga H, Kawamoto M et al. A single female-specific piRNA is the primary determiner of sex in the silkworm. Nature 2014; 509 (7502): 633–636. doi: 10.1038/nature13315.
48. Shpiz S, Ryazansky S, Olovnikov I et al. Euchromatic transposon insertions trigger production of novel Pi - and Endo-siRNAs at the target sites in the drosophila germline. PLoS Genet 2014; 10 (2): e1004138. doi: 10.1371/journal.pgen.1004138.
49. Qiao D, Zeeman AM, Deng W et al. Molecular characterization of hiwi, a human member of the piwi gene family whose overexpression is correlated to seminomas. Oncogene 2002; 21 (25): 3988–3999.
50. Suzuki R, Honda S, Kirino Y. PIWI expression and function in cancer. Front Genet 2012; 3 : 204. doi: 10.3389/fgene.2012.00204.
51. Sun G, Wang Y, Sun L et al. Clinical significance of Hiwi gene expression in gliomas. Brain Res 2011; 1373 : 183–188. doi: 10.1016/j.brainres.2010.11.097.
52. Wang Y, Liu Y, Shen X et al. The PIWI protein acts as a predictive marker for human gastric cancer. Int J Clin Exp Pathol 2012; 5 (4): 315–325.
53. Saito K, Nishida KM, Mori T et al. Specific association of Piwi with rasiRNAs derived from retrotransposon and heterochromatic regions in the Drosophila genome. Genes Dev 2006; 20 (16): 2214–2222.
54. Cox DN, Chao A, Baker J et al. A novel class of evolutionarily conserved genes defined by piwi are essential for stem cell self-renewal. Genes Dev 1998; 12 (23): 3715–3727.
55. Grimaud C, Bantignies F, Pal-Bhadra M et al. RNAi components are required for nuclear clustering of Polycomb group response elements. Cell 2006; 124 (5): 957–971.
56. Yin H, Lin H. An epigenetic activation role of Piwi and a Piwi-associated piRNA in Drosophila melanogaster. Nature 2007; 450 (7167): 304–308.
57. Siddiqi S, Matushansky I. Piwis and piwi-interacting RNAs in the epigenetics of cancer. J Cell Biochem 2012; 113 (2): 373–380. doi: 10.1002/jcb.23363.
58. Esteller M. Non-coding RNAs in human disease. Nat Rev Genet 2011; 12 (12): 861–874. doi: 10.1038/nrg3074.
59. Cheng J, Deng H, Xiao B et al. piR-823, a novel non-coding small RNA, demonstrates in vitro and in vivo tumor suppressive activity in human gastric cancer cells. Cancer Lett 2012; 315 (1): 12–17. doi: 10.1016/j.canlet.2011.10.004.
60. Iliev R, Stanik M, Fedorko M et al. Decreased expression levels of PIWIL1, PIWIL2, and PIWIL4 are associated with worse survival in renal cell carcinoma patients. Oncotargets Ther 2016; 9 : 217–222. doi: 10.2147/OTT.S91 295.
61. Iliev R, Fedorko M, Machackova T et al. Expression levels of PIWI-interacting RNA piR-823 are deregulated in tumor tissue blood serum and urine of patients with renal cell carcinoma. Anticancer Res 2016; 36 (12): 6419–6423.
62. Yan H, Wu QL, Sun CY et al. piRNA-823 contributes to tumorigenesis by regulating de novo DNA methylation and angiogenesis in multiple myeloma. Leukemia 2015; 29 (1): 196–206. doi: 10.1038/leu.2014.135.
63. Cuiyun Y, Ning Q, Li ZP et al. Non-coding RNAs: new therapeutic targets and opportunities for hepatocellular carcinoma. Adv Mod Oncol Res 2016; 2 (1): 5–17.
64. Cheng J, Guo JM, Xiao BX et al. piRNA, the new non-coding RNA, is aberrantly expressed in human cancer cells. Clin Chim Acta 2011; 412 (17–18): 1621–1625. doi: 10.1016/j.cca.2011.05.015.
65. Chu H, Hui G, Yuan L et al. Identification of novel piRNAs in bladder cancer. Cancer Lett 2015; 356 (2): 561–567. doi: 10.1016/j.canlet.2014.10.004.
66. Huang G, Hu H, Xue X et al. Altered expression of piRNAs and their relation with clinicopathologic features of breast cancer. Clin Transl Oncol 2013; 15 (7): 563–568. doi: 10.1007/s12094-012-0966-0.
67. Hashim A, Rizzo F, Marchese G et al. RNA sequencing identifies specific PIWI-interacting small non-coding RNA expression patterns in breast cancer. Oncotarget 2014; 5 (20): 9901–9910.
68. Martinez VD, Enfield KS, Rowbotham DA et al. An atlas of gastric PIWI-interacting RNA transcriptomes and their utility for identifying signatures of gastric cancer recurrence. Gastric Cancer 2016; 19 (2): 660–665. doi: 10.1007/s10120-015-0487-y.
69. Kishikawa T, Otsuka M, Ohno M et al. Circulating RNAs as new biomarkers for detecting pancreatic cancer. World J Gastroenterol 2015; 21 (28): 8527–8540. doi: 10.3748/wjg.v21.i28.8527.
70. Yang X, Cheng Y, Lu Q et al. Detection of stably expressed piRNAs in human blood. Int J Clin Exp Med 2015; 8 (8): 13353–13358.
71. Bahn JH, Zhang Q, Li F et al. The landscape of microRNA, Piwi-interacting RNA, and circular RNA in human saliva. Clin Chem 2015; 61 (1): 221–230. doi: 10.1373/clinchem.2014.230433.
72. Cui L, Lou Y, Zhang X et al. Detection of circulating tumor cells in peripheral blood from patients with gastric cancer using piRNAs as markers. Clin Biochem 2011; 44 (13): 1050–1057. doi: 10.1016/j.clinbiochem.2011.06.004.
73. Zhou H, Guo JM, Lou YR et al. Detection of circulating tumor cells in peripheral blood from patients with gastric cancer using microRNA as a marker. J Mol Med 2010; 88 (7): 709–717. doi: 10.1007/s00109-010-0617-2.
74. Li PF. Non-coding RNAs and gastric cancer. World J Gastroenterol 2014; 20 (18): 5411–5419. doi: 10.3748/wjg.v20.i18.5411.
75. Assumpção CB, Calcagno DQ, Araújo TM et al. The role of piRNA and its potential clinical implications in cancer. Epigenomics 2015; 7 (6): 975–984. doi: 10.2217/epi.15.37.
76. Mei Y, Clark D, Mao L. Novel dimensions of piRNAs in cancer. Cancer Lett 2013; 336 (1): 46–52. doi: 10.1016/j.canlet.2013.04.008.
77. Martinez VD, Vucic EA, Thu KL et al. Unique somatic and malignant expression patterns implicate PIWI-interacting RNAs in cancer-type specific biology. Sci Rep 2015; 5 : 10423. doi: 10.1038/srep10423.
78. Müller S, Raulefs S, Bruns P et al. Next-generation sequencing reveals novel differentially regulated mRNAs, lncRNAs, miRNAs, sdRNAs and a piRNA in pancreatic cancer. Mol Cancer 2015; 14 (1): 94. doi: 10.1186/s12943-015-0358-5.
79. Mei YP, Liao JP, Shen J et al. Small nucleolar RNA 42 acts as an oncogene in lung tumorigenesis. Oncogene 2012; 31 (22): 2794–2804. doi: 10.1038/onc.2011.449.
80. Busch J, Ralla B, Jung M et al. Piwi-interacting RNAs as novel prognostic markers in clear cell renal cell carcinomas. J Exp Clin Cancer Res 2015; 34 (1): 61. doi: 10.1186/s13046-015-0180-3.
81. Li Y, Wu X, Gao H et al. Piwi-interacting RNAs (piRNAs) are dysregulated in renal cell carcinoma and associated with tumor metastasis and cancer-specific survival. Mol Med Camb Mass 2015; 21 (1): 381–388. doi: 10.2119/molmed.2014.00203.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2016 Issue 6-
All articles in this issue
- Molekulárně genetické vyšetření u akutní myeloidní leukemie
- Molekulární genetika kolorektálního karcinomu
- Zapojení PIWI-interagujících RNA do procesů kancerogeneze prostřednictvím regulace genové exprese
- Klinický a funkční význam vybraných polymorfizmů CASP8 a CASP9 u karcinomu prsu
- Pacientka s primárním intraventrikulárním gliosarkomem s dlouhodobým přežíváním – kazuistika
- Intervenční cvičební program pro onkologické pacientky s karcinomem prsu
- Kvalita života, úzkost a deprese u pacientů s diferencovaným karcinomem štítné žlázy během krátkodobé hypotyreózy indukované vysazením levothyroxinu
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Molekulárně genetické vyšetření u akutní myeloidní leukemie
- Molekulární genetika kolorektálního karcinomu
- Pacientka s primárním intraventrikulárním gliosarkomem s dlouhodobým přežíváním – kazuistika
- Kvalita života, úzkost a deprese u pacientů s diferencovaným karcinomem štítné žlázy během krátkodobé hypotyreózy indukované vysazením levothyroxinu
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

