-
Medical journals
- Career
Co může přinést studium oligomerizace proteinů v procesu onkogeneze?
Authors: D. Coufalová; B. Vojtěšek; L. Hernychová
Authors‘ workplace: Regionální centrum aplikované molekulární onkologie, Masarykův onkologický ústav, Brno
Published in: Klin Onkol 2015; 28(Supplementum 2): 6-10
doi: https://doi.org/10.14735/amko20152S6Overview
Mnoho buněčných proteinů tvoří oligomery. Rovnováha mezi monomerním a oligomerním stavem těchto proteinů hraje důležitou roli v regulaci proteinové aktivity. Ovlivnění oligomerizace se tudíž nabízí jako zajímavý přístup při vývoji nových terapeutických látek. Cílem tohoto přehledového článku je shrnout informace o procesu proteinové oligomerizace a možnostech její cílené modulace. Role oligomerizace v onkogenezi je prezentována na příkladu nádorového supresorového proteinu p53, u kterého jsou v současné době zkoumány látky stabilizující jeho tetramerní strukturu. Z metod pro studium oligomerizace je zde představena metoda vodík/ deuteriové výměny ve spojení s hmotnostní spektrometrií, která je vhodná pro detekci protein‑proteinových interakcí a analýzu dynamiky oligomerizace.
Klíčová slova:
proteomika – vývoj nových léků – nádorový supresorový protein p53 – oligomerizace – vodík/ deuteriová výměnaÚvod
Proteiny se schopností oligomerizace reprezentují významný podíl všech buněčných proteinů [1]. Oligomery složené z identických proteinových podjednotek se nazývají homooligomery a oligomery tvořené interakcí mezi různými proteinovými podjednotkami jsou označovány jako heterooligomery. Oligomery většinou obsahují sudý počet podjednotek a tvoří symetrické struktury [2]. Síla interakce mezi podjednotkami a stabilita oligomerů se u různých komplexů liší a je závislá na koncentraci, teplotě a pH.
Procesu oligomerizace se účastní celá řada regulačních faktorů, které udržují dynamickou rovnováhu mezi jednotlivými oligomerními stavy proteinů v buňce. Navázání regulačních faktorů na podjednotky oligomerů může způsobovat změny ve struktuře podjednotek, a tím i dramatické změny ve struktuře a funkci celého oligomeru. Dříve se předpokládalo, že v důsledku strukturních změn se mění pouze struktura oligomeru, ale ne jeho stechiometrie (obr. 1) [3]. V roce 2005 byla publikována nová teorie oligomerizace, která ukázala na příkladu porfobilinogen syntázy (porphobilinogen synthase – PBGS; EC 4.2.1.24), že struktura proteinových podjednotek oligomeru stechiometrii oligomerizace ovlivňuje [4]. Dimery PBGS na základě alosterické modulace hořčíkem oligomerizují v inaktivní hexamer nebo v aktivní oktamer [5]. Je pravděpodobné, že podobná regulace bude nalezena i u dalších proteinů [6].
Image 1. Teorie oligomerizace. 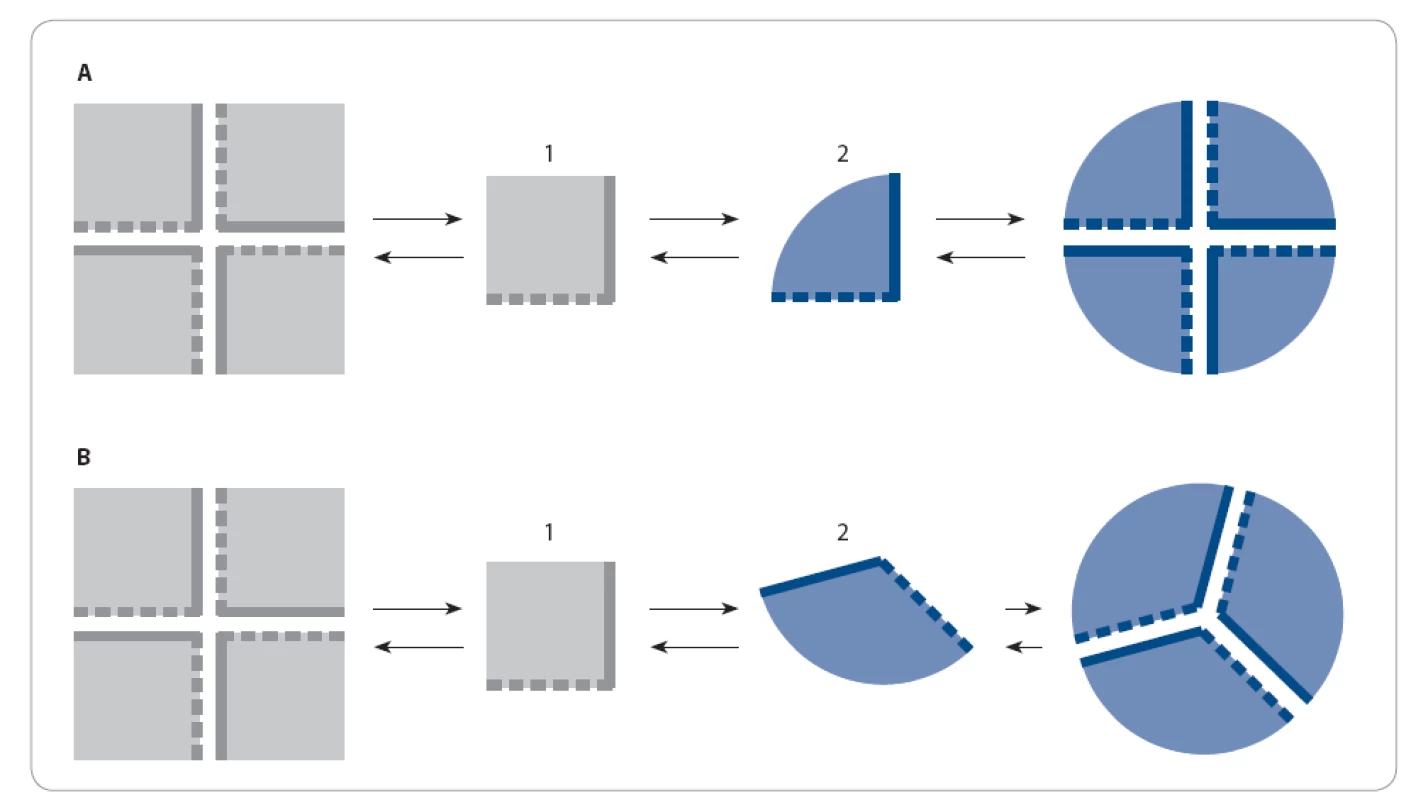
A. Klasická teorie: aktuální konformace proteinu ovlivňuje strukturu výsledného oligomeru, ale nemá vliv na jeho techiometrii. Protein v konformaci 1 i v konformaci 2 tvoří tetramer. B. Nová teorie: aktuální konformace proteinu určuje strukturu i stechiometrii oligomeru. Protein v konformaci 1 tvoří tetramer, avšak změní-li se jeho struktura v konformaci 2, tvoří trimer. Proč spolu proteiny tvoří stabilní oligomerní komplexy? Tato schopnost jim přináší řadu funkčních výhod [1]. Jednou z výhod je, že exprese podjednotek o nižší molekulové hmotnosti snižuje pravděpodobnost chyby při ribozomální syntéze. Tudíž oligomer složený z malých proteinových podjednotek bude syntetizován bezchybně s vyšší pravděpodobností než protein se srovnatelnou velikostí. Další výhodou je možnost modulace aktivity proteinů ovlivněním jejich oligomerizace. Je‑li protein aktivní pouze v oligomerní formě, lze iniciací oligomerizace protein aktivovat a její inhibicí naopak inaktivovat bez zásahů do proteinové exprese. V neposlední řadě je výhodou oligomerů jejich odolnost vůči denaturaci a následné degradaci.
Oligomery v onkogenezi
Jedním z nejstudovanějších proteinů tvořících oligomery je nádorový supresorový protein p53. Protein p53 je sekvenčně specifický transkripční faktor, který může během buněčného stresu zastavit buněčný cyklus, aktivovat mechanizmy pro opravu DNA, vyvolat senescenci, nebo apoptózu buněk [7]. Vzhledem k antionkogenním vlastnostem proteinu p53 jsou předmětem mnoha výzkumů faktory, které aktivitu tohoto proteinu regulují. Protein p53 se na DNA váže prostřednictvím DNA vazebné domény ve formě dimerů. Tyto dimery po vazbě k DNA vzájemně oligomerizují a dávají vzniknout aktivnímu tetrameru, který spouští transkripci [8,9]. Největší množství mutací vedoucích k inaktivaci proteinu p53 bylo popsáno právě v jeho DNA vazebné doméně [10]. Avšak i mutace v oligomerizační doméně, které blokují oligomerizaci, mohou způsobovat snížení nebo až zastavení jeho transaktivační aktivity [9]. Mutace v oligomerizační doméně nepůsobí pouze na aktivitu proteinu p53, ale také mají vliv na jeho lokalizaci v buňce. V oligomerizační doméně se nachází signální sekvence pro export proteinu z jádra do cytoplazmy. U tetramerů je tato sekvence na rozdíl od dimerů a monomerů skrytá [11]. Monomery a dimery proteinu p53 jsou transportovány z jádra do cytoplazmy, kde mohou být degradovány ubikvitin‑proteazomovou cestou, zatímco aktivní tetramery zůstávají v jádře. Vzhledem k významnosti proteinu p53 a důležitosti jeho tetramerizace roste počet studií, které se zabývají hledáním nízkomolekulárních ligandů stabilizujících jeho tetramerní formu [12– 15]. Tyto ligandy by v budoucnu mohly dát základ novým terapeutickým látkám. Kromě proteinu p53 je proces oligomerizace zkoumán i u dalších proteinů spojovaných s rakovinou, a to např. u proteinů AGR2 [16], MDM2 [17] a pyruvát kinázy M2 [18].
Ovlivňování oligomerizace a jeho přínos
Modulace oligomerizace je velice zajímavý přístup ve vývoji nových terapeutických látek. Jejím cílem může být inhibice oligomerizace anebo naopak stabilizace vznikajících oligomerů. Inhibovat oligomerizaci je možné za použití kompetitivních molekul, které se vážou přímo na oligomerizační místa proteinů, čímž zabraňují jejich vzájemné interakci [19,20]. Ke stabilizaci oligomerních molekul jsou využívány ligandy, které přemosťují vazbu mezi podjednotkami oligomeru a tím ji upevňují [14], nebo se vážou na jednotlivé monomery a cestou alosterických strukturních změn proteinů zvyšují jejich afinitu [21]. Jedním z dnes již používaných léků, který ovlivňuje oligomerizaci proteinů, je paclitaxel. Tento lék je užíván samostatně nebo v kombinaci s dalšími léky jako cytostatikum k léčbě rakoviny vaječníků, prsu, plic a dalších tkání [22]. Paclitaxel se váže na dimery tubulinu, stabilizuje vznikající mikrotubuly a tím inhibuje dělení buněk [23,24].
Metody studia oligomerů
Řada látek ovlivňujících oligomerizaci proteinů byla objevena náhodou [25]. Avšak díky rozvoji nových metod množství publikací zabývajících se hledáním látek zacílených na modulaci oligomerizace proteinů v posledních letech stoupá. K analýze struktury proteinů a jejich oligomerních komplexů jsou používány metody rentgenové krystalografie a nukleární magnetické rezonance [26]. Zatímco při rentgenové krystalografii je hlavní nevýhodou nutnost krystalizace proteinů, nukleární magnetická rezonance sice proteiny měří v roztoku, ale nelze ji použít pro analýzu velkých komplexů. Vzhledem k těmto omezením jsou k určení oligomerního stavu používány také metody gelové chromatografie a analytické ultracentrifugace, které navíc dokáží určit i relativní zastoupení jednotlivých oligomerů [27,28]. S uvedenými technikami se lze v literatuře setkat nejčastěji, a ačkoliv jsou schopny poskytnout detailní informace o struktuře oligomerních komplexů, nedokáží měřit molekulární dynamiku a drobné konformační změny proteinů.
Vodík/ deuteriová výměna
Metoda vodík/ deuteriové (H/ D) výměny dokáže nejen detekovat koexistující monomerní a oligomerní formy proteinů, ale také analyzovat dynamiku oligomerizace a najít vazebná místa proteinů. H/ D výměna je s oblibou spojována s hmotnostní spektrometrií [29], ale k detekci vyměněných vodíků je možné použít i nukleární magnetickou rezonanci [30]. Metoda je založena na principu výměny amidových vodíků proteinů za deuteria z okolního roztoku [31]. Vodíky, které tvoří vodíkové můstky, případně nepřístupné vodíky uvnitř složeného proteinu, jsou vůči výměně chráněny. Naopak snadno přístupné vodíky na povrchu proteinu jsou vyměňovány za deuteria s vysokou rychlostí. Jelikož je deuterium těžší než vodík, způsobuje jeho zabudování posun v hmotnostních spektrech k vyšším hmotnostem. Díky tomu je možné v hmotnostních spektrech od sebe odlišit různé konformace proteinu, které se liší v počtu zabudovaných deuterií.
Při H/ D výměně existují dva základní typy analýz – lokální a globální [32]. U obou analýz je protein inkubován v pufru s D2O a výměnná reakce je zastavována snížením pH a snížením teploty (obr. 2). U lokální analýzy je protein následně štěpen pepsinem nebo jinou proteázou štěpící v oblasti kyselého pH. Vzniklé peptidy jsou separovány pomocí kapalinové chromatografie on‑line spojené s hmotnostním spektrometrem. Naopak u globální analýzy je štěpení proteinu vynecháno a protein je analyzován v hmotnostním spektrometru v nativní formě. Globální analýza je užitečná k celkovému náhledu na chování proteinů, k detekci protein‑proteinových interakcí a k odhalení přítomnosti oligomerních forem proteinů ve vzorku. Lokální analýza poskytuje informace na úrovni peptidů a je schopna lokalizovat místa protein‑proteinových interakcí a alosterických strukturních změn proteinů. Ke studiu oligomerizace proteinů je využívána globální i lokální analýza [33,34].
Image 2. H/D výměna – globální a lokální analýza. 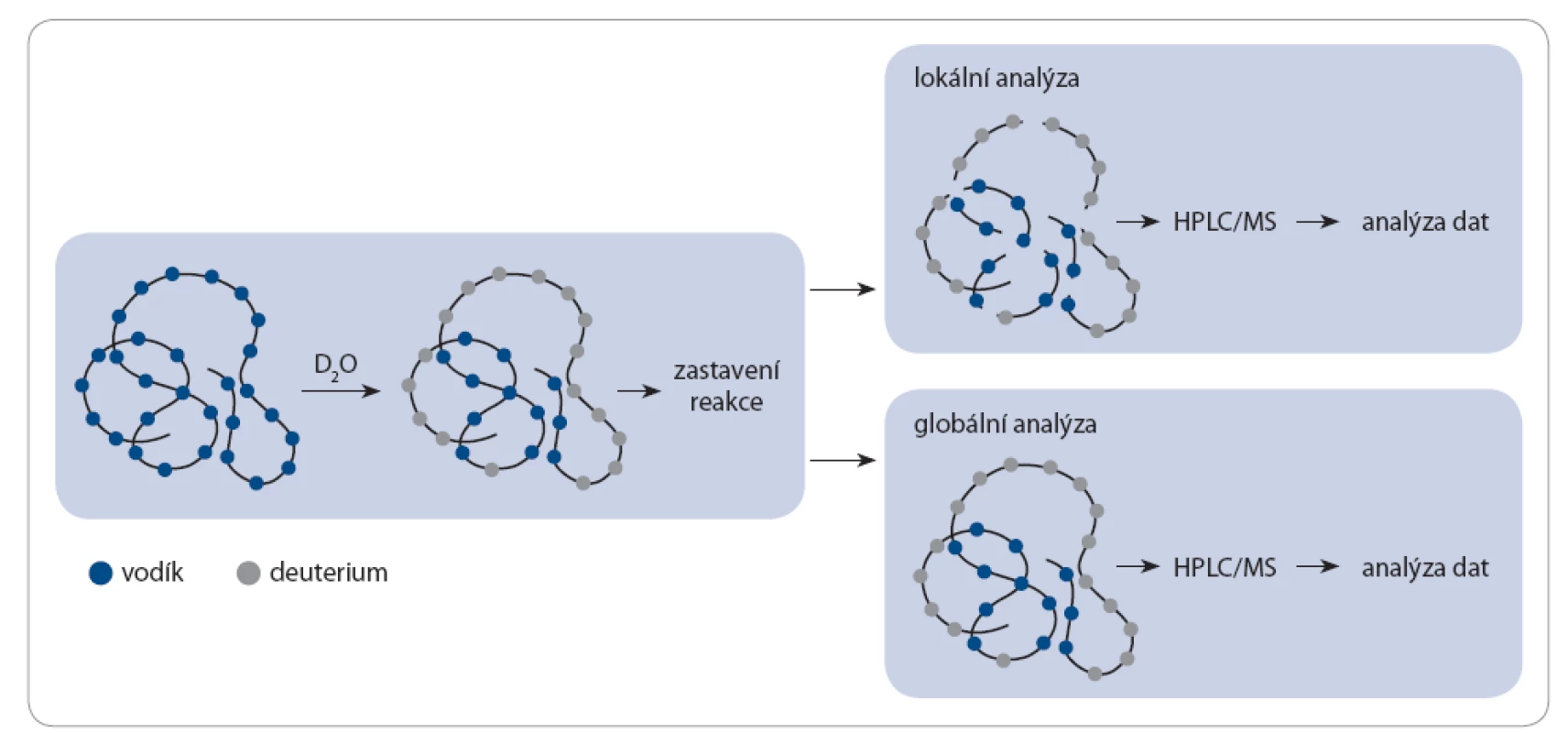
H/D výměna je zahájena inkubací proteinu v pufru s D2O, kdy dochází k výměně amidových vodíků za deuteria z pufru. Při lokální analýze je po zastavení reakce protein štěpen a výsledné peptidy jsou separovány kapalinovou chromatografi í (high performance liquid chromatography – HPLC) a měřeny hmotnostním spektrometrem (mass spectrometry – MS). Globální analýza vynechává krok štěpení a protein měří pomocí HPLC/MS v nativní formě. Je‑li v analyzovaném vzorku protein přítomen v monomerní i oligomerní formě, je možné oba tyto stavy sledovat zároveň v jednom hmotnostním spektru. U monomerního proteinu lze očekávat vyšší rychlost výměny vodíků za deuteria než u proteinu v oligomerní formě, jelikož je celý jeho povrch přístupný okolnímu roztoku. V oligomerním proteinu si podjednotky vzájemně stíní, což způsobuje snížení deuterace ve stíněných místech. Díky tomu jsou ve výsledném hmotnostním spektru přítomné dva píky, které náleží proteinu v monomerní a oligomerní formě.
Metodu H/ D výměny je tedy možné využít k analýze struktury oligomerních komplexů, ale je také vhodná ke studiu látek ovlivňujících oligomerizaci proteinů, které mohou mít terapeutický potenciál.
Závěr
Detailní porozumění protein‑proteinovým interakcím, mezi které patří i tvorba oligomerních struktur, je nezbytné k důkladnému pochopení biologických dějů a může být klíčem k návrhu nových terapeutických látek. Pro studium protein‑proteinových interakcí je unikátní metoda H/ D výměna, která lokalizuje vazebná místa proteinů a analyzuje jejich alosterické strukturní změny. Pomocí této metody lze studovat i interakce mezi potenciálními léčivy a proteiny a vliv těchto léčiv na změnu proteinové struktury a na změny v interakcích mezi studovanými proteiny.
Práce byla podpořena Evropským fondem pro regionální rozvoj a státním rozpočtem České republiky (RECAMO, CZ.1.05/2.1.00/03.0101), projektem MŠMT – NPU I – LO1413, MZ ČR –RVO (MOÚ, 00209805) a BBMRI_CZ (LM2010 - 004).
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
Mgr. Dominika Coufalová
Regionální centrum aplikované molekulární onkologie
Masarykův onkologický ústav
Žlutý kopec 7
656 53 Brno
e-mail: dominika.coufalova@mou.cz
Obdrženo: 9. 4. 2015
Přijato: 15. 6. 2015
Sources
1. Ali MH, Imperiali B. Protein oligomerization: how and why. Bioorganic Med Chem 2005; 13(17): 5013 – 5020.
2. Goodsell DS, Olson AJ. Structural symmetry and protein function. Annu Rev Biophys Biomol Struct 2000; 29 : 105 – 153.
3. Koshland DE, Némethy G, Filmer D. Comparison of experimental binding data and theoretical models in proteins containing subunits. Biochemistry 1966; 5(1): 365 – 385.
4. Jaffe EK. Morpheeins – a new structural paradigm for allosteric regulation. Trends Biochem Sci 2005; 30(9): 490 – 497.
5. Jaffe EK, Lawrence SH. Allostery and the dynamic oligomerization of porphobilinogen synthase. Arch Biochem Biophys 2012; 519(2): 144 – 153. doi: 10.1016/ j.abb.2011.10.010.
6. Selwood T, Jaffe EK. Dynamic dissociating homo ‑ oligomers and the control of protein function. Arch Biochem Biophys 2012; 519(2): 131 – 143. doi: 10.1016/ j.abb.2011.11.020.
7. Šmardová J, Koptíková J. Brazilský příběh mutace p53 R337H. Klin Onkol 2014; 27(4): 247 – 254. doi: 10.14735/amko2014247.
8. Kitayner M, Rozenberg H, Kessler N et al. Structural basis of DNA recognition by p53 tetramers. Mol Cell 2006; 22(6): 741 – 753.
9. Kawaguchi T, Kato S, Otsuka K et al. The relationship among p53 oligomer formation, structure and transcriptional activity using a comprehensive missense mutation library. Oncogene 2005; 24(46): 6976 – 6981.
10. Olivier M, Eeles R, Hollstein M et al. The IARC TP53 database: new online mutation analysis and recommendations to users. Hum Mutat 2002; 19(6): 607 – 614.
11. Stommel JM, Marchenko ND, Jimenez GS et al. A leucine ‑ rich nuclear export signal in the p53 tetramerization domain: regulation of subcellular localization and p53 activity by NES masking. EMBO J 1999; 18(6): 1660 – 1672.
12. Salvatella X, Martinell M, Gairí M et al. A tetraguanidinium ligand binds to the surface of the tetramerization domain of protein p53. Angew Chem Int Ed Engl 2004; 43(2): 196 – 198.
13. Martinell M, Salvatella X, Fernández ‑ Carneado J et al. Synthetic ligands able to interact with the p53 tetramerization domain. Towards understanding a protein surface recognition event. Chembiochem 2006; 7(7): 1105 – 1113.
14. Kamada R, Yoshino W, Nomura T et al. Enhancement of transcriptional activity of mutant p53 tumor suppressor protein through stabilization of tetramer formation by calix[6]arene derivatives. Bioorg Med Chem Lett 2010; 20(15): 4412 – 4415. doi: 10.1016/ j.bmcl.2010.06.053.
15. Gabizon R, Brandt T, Sukenik S et al. Specific recognition of p53 tetramers by peptides derived from p53 interacting proteins. PLoS One 2012; 7(5): e38060. doi: 10.1371/ journal.pone.0038060.
16. Gray TA, Murray E, Nowicki MW et al. Development of a fluorescent monoclonal antibody‑based assay to measure the allosteric effects of synthetic peptides on self ‑ oligomerization of AGR2 protein. Protein Sci 2013; 22(9): 1266 – 1278. doi: 10.1002/ pro.2299.
17. Graves B, Thompson T, Xia M et al. Activation of the p53 pathway by small‑molecule‑induced MDM2 and MDMX dimerization. Proc Natl Acad Sci U S A 2012; 109(29): 11788 – 11793. doi: 10.1073/ pnas.1203789109.
18. Anastasiou D, Yu Y, Israelsen WJ et al. Pyruvate kinase M2 activators promote tetramer formation and suppress tumorigenesis. Nat Chem Biol 2012; 8(10): 839 – 847.
19. Miller GD, Woessner DW, Sirch MJ et al. Multidomain targeting of Bcr ‑ Abl by disruption of oligomerization and tyrosine kinase inhibition: toward eradication of CML. Mol Pharm 2013; 10(9): 3475 – 3483. doi: 10.1021/ mp400323c.
20. He MM, Smith AS, Oslob JD et al. Small‑molecule inhibition of TNF‑alpha. Science 2005; 310(5750): 1022 – 1025.
21. Teufel DP, Freund SM, Bycroft M et al. Four domains of p300 each bind tightly to a sequence spanning both transactivation subdomains of p53. Proc Natl Acad Sci U S A 2007; 104(17): 7009 – 7014.
22. Rowinsky EK. The development and clinical utility of the taxane class of antimicrotubule chemotherapy agents. Annu Rev Med 1997; 48 : 353 – 374.
23. Mitra A, Sept D. Taxol allosterically alters the dynamics of the tubulin dimer and increases the flexibility of microtubules. Biophys J 2008; 95(7): 3252 – 3258. doi: 10.1529/ biophysj.108.133884.
24. Xiao H, Verdier ‑ Pinard P, Fernandez ‑ Fuentes N et al.Insights into the mechanism of microtubule stabilization by Taxol. Proc Natl Acad Sci U S A 2006; 103(27): 10166 – 10173.
25. Gabizon R, Friedler A. Allosteric modulation of protein oligomerization: an emerging approach to drug design. Front Chem 2014; 2(9): 1 – 15. doi: 10.3389/ fchem.2014.00009.
26. Veldkamp CT, Seibert C, Peterson FC et al. Structural basis of CXCR4 sulfotyrosine recognition by the chemokine SDF ‑ 1/ CXCL12. Sci Signal 2008; 1(37): ra4. doi: 10.1126/ scisignal.1160755.
27. Lee CC, Lin TW, Ko TP et al. The hexameric structures of human heat shock protein 90. PLoS One 2011; 6(5): e19961. doi: 10.1371/ journal.pone.0019961.
28. Aprile FA, Dhulesia A, Stengel F et al. Hsp70 oligomerization is mediated by an interaction between the interdomain linker and the substrate‑binding domain. PLoS One 2013; 8(6): e67961. doi: 10.1371/ journal.pone.0067961.
29. Hamuro Y, Coales SJ, Southern MR et al. Rapid analysis of protein structure and dynamics by hydrogen/ deuterium exchange mass spectrometry. J Biomol Tech 2003; 14(3): 171 – 182.
30. Dempsey CE. Hydrogen exchange in peptides and proteins using NMR spectroscopy. Prog Nucl Magn Reson Spectrosc 2001; 39(2): 135 – 170.
31. Hvidt A, Nielsen SO. Hydrogen exchange in proteins. Adv Protein Chem 1966; 21 : 287 – 386.
32. Wales TE, Eggertson MJ, Engen JR. Mass spectrometry data analysis in proteomics. Methods Mol Biol 2013; 1007 : 263 – 288.
33. Paslawski W, Mysling S, Thomsen K et al. Co ‑ existence of two different α ‑ synuclein oligomers with different core structures determined by hydrogen/ deuterium exchange mass spectrometry. Angew Chem Int Ed Engl 2014; 53(29): 7560 – 7563. doi: 10.1002/ anie.201400491.
34. Mobley JA, Poliakov A. Detection of early unfolding events in a dimeric protein by amide proton exchange and native electrospray mass spectrometry. Protein Sci 2009; 18(8): 1620 – 1627. doi: 10.1002/pro.176.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2015 Issue Supplementum 2-
All articles in this issue
- Nádory u adolescentů
- Bioinformatika a sekvenování nové generace
- Co může přinést studium oligomerizace proteinů v procesu onkogeneze?
- Analýza změn fosfoproteomu nádorové buněčné linie MDA‑ MB‑ 468 v odpovědi na expresi izoforem p63 pomocí hmotnostní spektrometrie
- „Cukry zasahují“ aneb glykomika na poli nádorových biomarkerů
- Nrf2 – dve tváre regulátora antioxidačného systému
- Polo‑like kináza 1 jako cíl protinádorové terapie
- PDLIM2 a jeho role v onkogenezi – tumor supresor nebo onkoprotein?
- Profil aktivace receptorových tyrozinkináz a mitogenem aktivovaných proteinkináz v terapii Maffucciho syndromu
- Rekombinantní protilátky a jejich využití v protinádorové terapii
- Imunologické aspekty v onkologii – cirkulující γδ T lymfocyty
- Cirkulující nádorová DNA v krvi a její využití jako potenciálního biomarkeru nádorových onemocnění
- Adenovírusové vektory v génovej terapii
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Adenovírusové vektory v génovej terapii
- Nrf2 – dve tváre regulátora antioxidačného systému
- Rekombinantní protilátky a jejich využití v protinádorové terapii
- Co může přinést studium oligomerizace proteinů v procesu onkogeneze?
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

