-
Medical journals
- Career
Potenciál volné cirkulující DNA v diagnostice nádorových onemocnění
Authors: V. Kubaczkova 1,2; L. Sedlaříková 1,2; L. Bešše 1; M. Almaši 1,2; Hájek R. 1–3; S. Ševčíková 1,2
Authors‘ workplace: Babákova myelomová skupina, Ústav patologické fyziologie, LF MU, Brno 1; Oddělení klinické hematologie, FN Brno 2; Klinika hematoonkologie LF OU a FN Ostrava 3
Published in: Klin Onkol 2015; 28(4): 251-259
Category: Review
doi: https://doi.org/10.14735/amko2015251Overview
Volná cirkulující DNA (cf ‑ DNA) je charakterizována jako extracelulární DNA, která může být přítomna v nízkých koncentracích v krvi zdravých jedinců. Cf ‑ DNA je do krevního řečiště uvolňována apoptózou, ale i nekrózou. Zvýšené hladiny se vyskytují u patologických stavů, jako je zánět, stres či autoimunitní onemocnění. Výrazně zvýšené hodnoty cf ‑ DNA jsou patrné zejména u pacientů s malignitami, a to především v pokročilých stadiích nemoci. V takovémto případě je nádorově specifická cf‑DNA uvolňována nekrózou z buněk primárního nádoru a metastáz. V poslední době se hodně studií zabývá tzv. liquid biopsy (tekutou biopsií), která umožňuje poměrně snadnou detekci cirkulujících nádorových buněk i cirkulujících molekul nukleových kyselin z periferní krve k diagnostice nádorových onemocnění. Kvantitativní stanovení a detekce genetických a epigenetických změn v cf‑DNA u pacientů s různými malignitami má potenciální využití v molekulární diagnostice, prognóze, monitorování průběhu nemoci a odpovědi na léčbu. Tento článek je zaměřen na potenciální využití cf‑DNA jako krevního biomarkeru u vybraných solidních nádorů a hematologických malignit.
Klíčová slova:
volná cirkulující DNA – nádorový marker – solidní nádory – hematologické malignityÚvod a historie
Volná cirkulující DNA, z anglického výrazu cell‑free DNA (cf‑DNA), představuje extracelulární krátké fragmenty dvouřetězcových molekul DNA o délce 180 bp vyskytující se v krevním oběhu a jiných tělních tekutinách člověka [1].
Cf‑DNA v krevní plazmě zdravých jedinců byla poprvé popsána v roce 1948 [2]. V roce 1977 Leon et al popsali výskyt cf‑DNA v souvislosti s nádory. Pomocí radio ‑ imuno ‑ analýzy bylo otestováno celkem 173 vzorků sér pacientů s maligními nádory (plic, střev, ledvin, prostaty, dělohy, vaječníků, prsu aj.) a 55 vzorků sér zdravých osob. Zjistili, že pacienti s nádorovým onemocněním vykazovali v plazmě významně vyšší hladiny cf‑DNA oproti zdravým kontrolám. V případě zdravých dárců se průměrná koncentrace cf‑DNA pohybovala kolem 13 ng/ ml, zatímco u pacientů s maligními nádory naměřené hodnoty dosahovaly rozmezí 0 – 5 000 ng/ ml s průměrnou koncentrací cf‑DNA 180 ng/ ml. Výrazně vyšší koncentrace cf-DNA byly zjištěny v séru pacientů s metastázami ve srovnání s pacienty s lokalizovaným nádorem. Dále po radioterapii byl prokázán pokles hladiny cf‑DNA přibližně u 66 – 90 % pacientů s několika různými malignitami, jako např. nádory plic, vaječníků, dělohy či lymfomy. Snížení hladiny cf‑DNA korelovalo se zlepšením stavu pacienta (snížení velikosti nádoru a bolesti). Přetrvávající vysoké koncentrace cf‑DNA pak byly asociovány s rezistencí k léčbě a špatnou prognózou [3].
Vlastnosti a biologie cf‑DNA
V periferní krvi zdravých jedinců se cf‑DNA vyskytuje ve velmi nízkých koncentracích (10 – 100 ng/ ml). K uvolnění fragmentů molekul DNA z buněk dochází apoptózou, tedy programovanou buněčnou smrtí [4]. V takovémto případě fragmenty DNA dosahují délky kolem 180 bp a v periferní krvi jsou navázány na erytrocyty [5]. Fragmenty cf‑DNA v krevním oběhu mají krátký poločas rozpadu, zhruba za 10 – 15 min jsou zejména činností jater odstraněny z krevního řečiště [6].
Některé studie ukázaly, že buňky mohou cf‑DNA uvolňovat aktivně. Tato frakce DNA může být zodpovědná za některé buněčné funkce, jako je transkripce, případně se může podílet na komunikaci mezi buňkami a tkáněmi [5].
Ke změnám v kvalitě a také v množství cf‑DNA v oběhu dochází v souvislosti s patologickým stavem jedince. Vyšší koncentrace cf‑DNA nacházíme u stavů, jako je zánět, trauma, operační zákrok, infarkt či autoimunitní onemocnění [7 – 10]. Zvýšené hladiny cf‑DNA v krevním řečišti jsou výsledkem nadměrného uvolnění fragmentů molekul DNA, které byly způsobeny masivním úmrtím buněk či nedostatečným odstraněním uvolněné DNA z mrtvých buněk činností jater [6].
Vlastnosti cf‑DNA u nádorových onemocnění
Výrazně vyšší hladiny cf‑DNA, dosahující koncentrace až 1 000 ng/ ml, jsou patrné především u různých druhů malignit a zejména pak u pacientů s metastázemi [11]. Nádorově specifická cf‑DNA je do oběhu uvolňována nekrózou nádorových buněk (obr. 1). Do krevního oběhu je DNA také uvolňována buňkami zdravé tkáně, které se nacházejí v blízkosti nádoru [1]. V krevním oběhu se obvykle vyskytuje ve formě velkých fragmentů delších než 180 bp, které s proteiny vytvářejí komplexy připomínající nukleozom [12].
Image 1. Uvolňování cf-DNA nekrózou z nádorových buněk do krevního řečiště. Do krevního oběhu je také uvolňována cf-DNA z buněk v okolí nádoru. 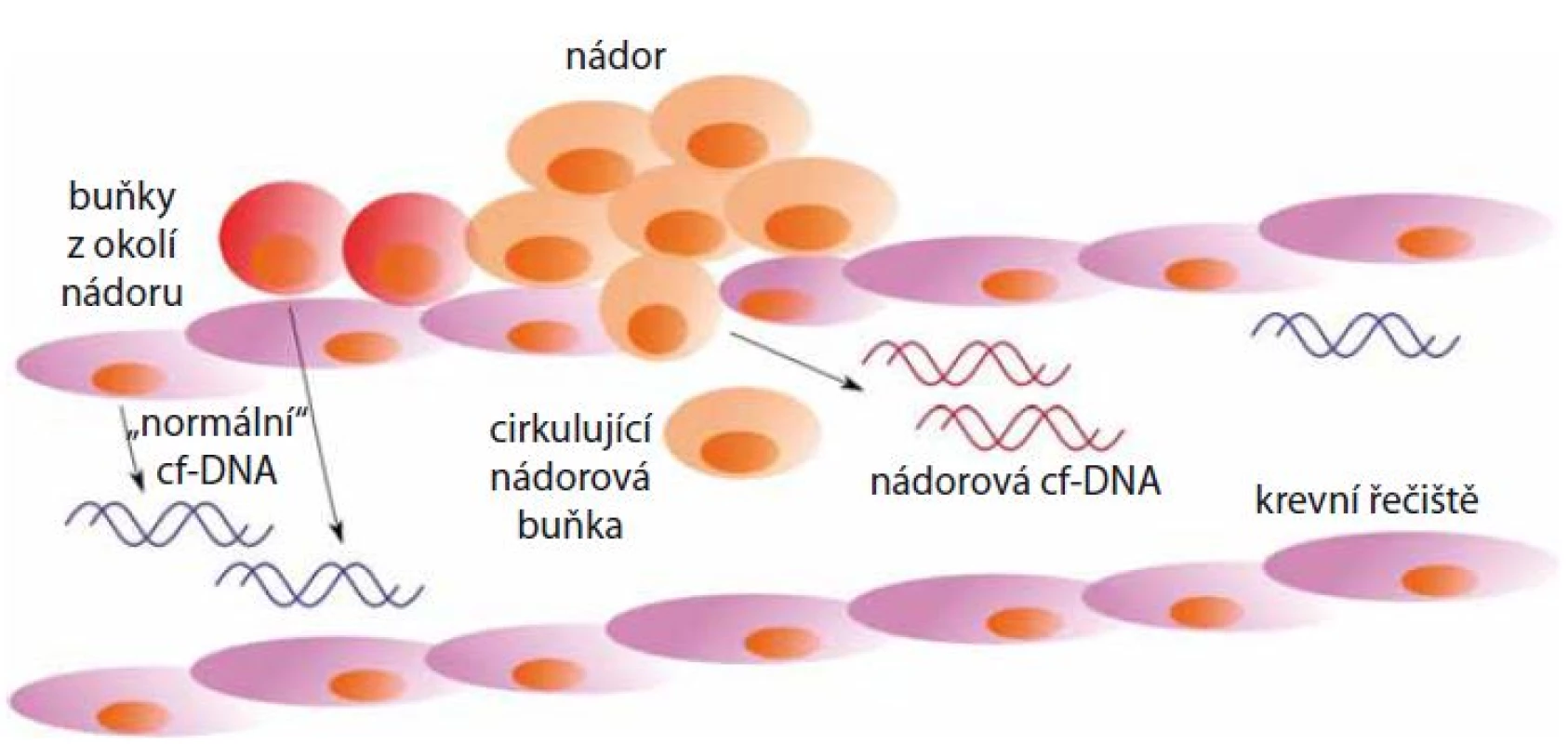
Diehl et al zjistili, že u pacientů s nádorem o velikosti 100 g (který je přibližně tvořen 3 × 1010 nádorovými buňkami) se denně do krevního řečiště uvolní 3,3 % nádorové cf‑DNA [13]. Nádorová DNA je z 10 – 90 % tvořena cf‑DNA, tedy fragmenty molekul DNA, které jsou apoptózou uvolněny z buněk v okolí nádoru. Množství nádorové cf‑DNA závisí na typu nádoru. Nízký podíl nádorové frakce může být způsoben tím, že nádorové buňky jsou oddělovány od nádoru a do krevního oběhu jsou uvolňovány až během procesu epiteliálně‑mezenchymálního přechodu [14].
Praktické využití cf‑DNA
Přestože byla cf‑DNA poprvé identifikována před více než 60 lety, výzkum cf‑DNA značně zaostal z důvodu chybějících citlivých a specifických analytických metod. V důsledku těchto laboratorních omezení došlo k přenesení poznatků základního výzkumu do klinické praxe s mnoholetým zpožděním. Teprve vývoj efektivních izolačních postupů a citlivých metod využívajících nová a specifická fluorescenční barviva a zdokonalené PCR techniky umožnil v průběhu posledních dvou desetiletí postup v oblasti výzkumu cf‑DNA [15].
Kvantitativní a kvalitativní analýza cf‑DNA se v poslední době stala jedním ze slibných diagnostických přístupů. Jedním ze zásadních faktorů, které ovlivňují hodnocení cf‑DNA, je standardizace metodických postupů a definování optimálního typu analyzovaného vzorku (sérum nebo plazma) tak, aby bylo dosaženo konzistentních a srovnatelných dat mezi jednotlivými laboratořemi. V současné době se jednotlivé metodické postupy liší v preanalytické fázi, izolaci DNA, kvantifikaci a analýze, a tím i v interpretaci výsledků [16].
Praktické využití u nádorových onemocnění
Vědci zjistili, že prakticky všechny druhy nádorů jsou zapříčiněny genetickými změnami v DNA, které u pacientů mohou být detekovány a dále sledovány. Přestože sám nádor je hlavním zdrojem nádorové DNA, získání DNA pomocí biopsie představuje pro pacienta invazivní a riskantní postup, který v některých případech není možné provést [16].
Detekce nádorově specifické cf‑DNA, nazývaná také tekutá biopsie (liquid biopsy), se stává nadějným nástrojem pro minimálně invazivní monitoring pacientů s maligním onemocněním. Frakce DNA pocházející z nádorové tkáně se volně vyskytují v periferní krvi těchto pacientů (zejména v pokročilých stadiích nemoci). Nedávné studie také ukázaly, že cf‑DNA odvozená od nádorových buněk může být použita pro monitorování nádorové masy a odpovědi na léčbu u pacientů se solidními nádory, ale také u pacientů s hematologickými malignitami. Hladiny cf‑DNA v plazmě pacientů korelují s progresí nemoci, umožňují kontinuálně sledovat minimální reziduální chorobu, určit molekulární relaps onemocnění a zahájit tak včasnou léčbu [17].
V cf‑DNA můžeme detekovat nádorově specifické genetické a epigenetické změny, které jsou přítomny rovněž v buňkách primárního nádoru [18]. Těmito genetickými změnami jsou bodové mutace v důležitých genech, chromozomové přestavby, ztráta heterozygozity (loss of heterozygosity – LOH), změny v mikrosatelitech a v metylaci DNA. Detekce těchto charakteristických změn odliší specifickou nádorovou cf‑DNA od pozadí „normální“ cf‑DNA v krevním oběhu a tím umožňuje vyšší diagnostickou specificitu ve srovnání s pouhým kvantitativním stanovením celkového množství cf‑DNA [19].
Solidní nádory
Kolorektální karcinom
V ČR je kolorektální karcinom (colorectal cancer – CRC) u obou pohlaví druhým nejčastějším nádorovým onemocněním. Ročně je nově diagnostikováno kolem 8 000 pacientů a zhruba polovina z nich na něj umírá. U mužů je incidence i mortalita tohoto karcinomu vyšší než u žen [20]. V tab. 1 jsou uvedeny vybrané studie zabývající se využitím cf‑DNA u tohoto onemocnění [21 – 40].
Table 1. Vybrané studie zabývající se kvantitativní a kvalitativní analýzou cf-DNA u kolorektálního karcinomu. 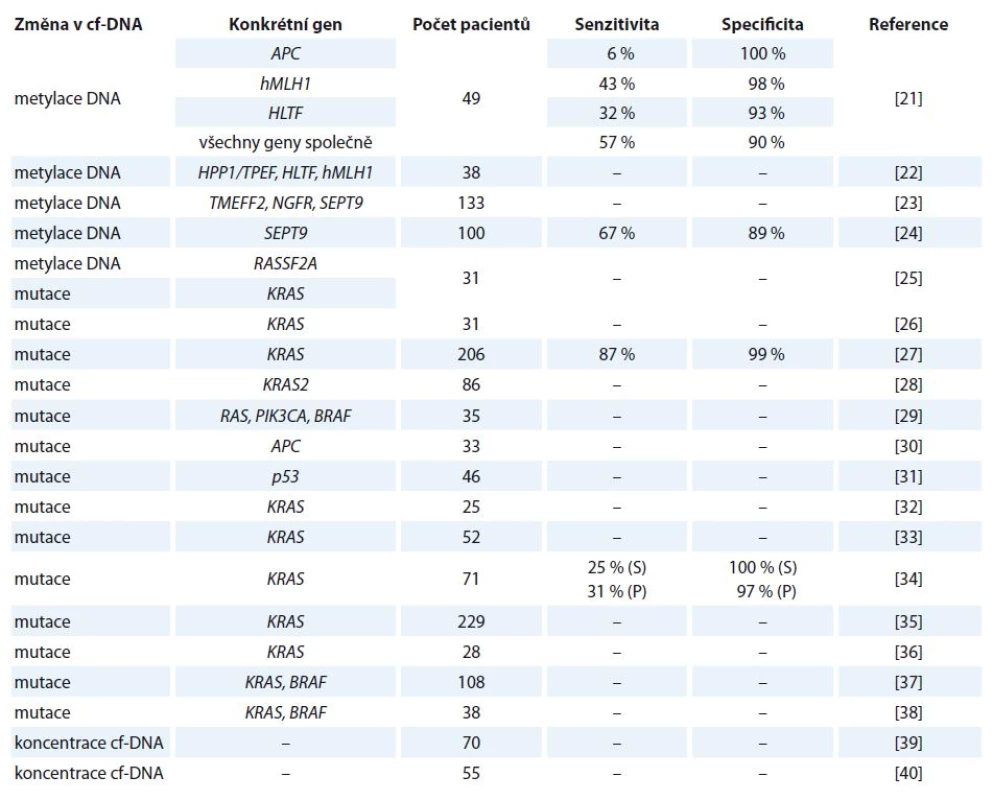
S – hodnoty vztažené pro vzorky séra, P – hodnoty vztažené ke vzorkům plazmy Kvantifikace nádorově specifické cf‑DNA může představovat užitečný nástroj pro monitorování pacientů s CRC a také může sloužit při rozpoznávání vysoce rizikových pacientů. Frattini a jeho výzkumný tým zjistili, že u pacientů s CRC byly hladiny cf‑DNA v periferní krvi výrazně vyšší oproti zdravým jedincům. Dále zjistili, že hladiny cf‑DNA po operačním zákroku postupně klesaly a naopak jejich zvyšování bylo zaznamenáno u pacientů s metastázemi či u relapsu onemocnění [39]. Tyto výsledky jsou v souladu s poznatky jiné studie, ve které Schwarzenbach a jeho výzkumný tým uvedli, že u pacientů ve IV. stadiu CRC byly hladiny cf‑DNA výrazně vysoké a během 1. linie chemoterapie docházelo k jejich poklesu [40]. Toto potvrzuje i studie, ve které bylo zjištěno, že detekovatelné množství cf‑DNA v periferní krvi koreluje se stadiem nádoru. Pouze u 47 % pacientů s CRC I. stupně byly měřitelné hodnoty cf‑DNA. Se zvyšujícím se stupněm progrese nemoci se hladiny cf‑DNA zvyšovaly, např. u II. stupně byly hladiny měřitelné u 55 % pacientů, u IV. stupně až u 82 % z nich. Toto zjištění tedy poukazuje na fakt, že pouze měření hladiny cf‑DNA v krvi pacienta by mohlo být v budoucnu použito k určení stadia progrese rakoviny. Když byly měřeny koncentrace cf‑DNA u 206 pacientů v pokročilém stadiu CRC s metastázemi, bylo zjištěno, že pacienti s nižšími krevními hladinami cf‑DNA dosahovali delší doby přežití [27].
Významnou genetickou změnou, kterou můžeme u pacientů s CRC v cf‑DNA sledovat, jsou somatické mutace v genu K RAS. Tento gen je charakteristický vysokou četností mutací, které přispívají k progresi nádoru [41]. Terapeutickou možností pro pacienty s CRC je cílená biologická léčba monoklonálními protilátkami (cetuximab a panitumumab) založená na inhibici EGFR a signálních drah navozujících nádory. Negativním prediktorem léčebné odpovědi na anti‑EGFR terapii u pacientů s metastatickým kolorektálním karcinomem (metastatic colorectal cancer – mCRC) jsou aktivační somatické mutace v kodonech 12. a 13. genu K RAS, jenž se účastní signálních kaskád spouštěných EGFR. V současné době je podmínkou pro indikaci cílené anti‑EGFR terapie průkaz nemutovaného genu K RAS [34,36].
Přítomnost aktivačních mutací v genu K RAS lze také detekovat v cf‑DNA odvozené z primárního nádoru a metastáz. Výrazná výhoda této metody je především v jejím minimálně invazivním přístupu, neboť získání vzorků nádorové tkáně biopsií pro účely genetické analýzy je obtížné [36]. V nedávné studii Bettegowda et al detekovali u 206 pacientů s mCRC přítomnost mutace v kodonu 12. nebo 13. genu K RAS v cf‑DNA ze séra a tkání primárního nádoru. U 33 % pacientů byla v cf‑DNA detekována mutovaná varianta genu, tyto mutace byly nalezeny i v primárním nádoru. Procentuální shoda mezi K RAS mutacemi v séru a nádorové tkáni byla 95 %. U provedeného testování detekce mutací v klinicky důležitém genu K RAS byla stanovena 87% senzitivita a 99% specificita [27]. Dále bylo zjištěno, že u 24 pacientů, kteří nejprve vykazovali dobrou reakci na léčbu anti‑EGFR monoklonálními protilátkami, došlo později k relapsu onemocnění. U 96 % z nich došlo k rozvinutí jedné nebo více mutací v genu K RAS [27].
Trevisiol et al se ve své studii zabývali prognostickým významem stanovení mutace v kodonu 12. genu K RAS2 v cf‑DNA ze séra 86 pacientů s CRC, kteří podstoupili léčbu. Zjištěné údaje byly následně porovnány se stavem mutací v genu K RAS2 u primárních nádorů [28]. Pacienti s pokročilou formou nemoci a detekovanou mutací v genu K RAS2 v séru zemřeli do 24 měsíců od provedení chirurgického zákroku. U těchto pacientů byla po chirurgické resekci detekovatelná reziduální choroba. Doba přežití byla výrazně lepší u pacientů s pokročilou formou nemoci bez mutace v genu K RAS2, 56 % z nich vykazovalo dobu přežití více než 24 měsíců a jeden pacient byl naživu po uplynutí 44 měsíců [25].
Pomocí PCR bylo testováno 31 sér pacientů s pokročilou formou CRC a 24 sér zdravých osob. Bodová mutace v genu K RAS byla detekována v cf‑DNA u 12 (39 %) pacientů s CRC a ani v jednom případě u zdravých jedinců. Výsledky ukazují, že mutace v genu K RAS jsou snadno zjistitelné PCR technikami v plazmě/ séru pacientů s pokročilým CRC. Toto testování může představovat možný způsob detekce a monitorování CRC [26].
Karcinom plic
Karcinom plic představuje významnou skupinu nádorů u mužů v české populaci [20]. Již Leon et al ve své práci zjistili, že vyšší hodnoty cf‑DNA byly patrné u pacientů s metastázemi oproti pacientům s lokalizovaným nádorem plic. Po léčbě došlo k poklesu hladin cf‑DNA u 75 % pacientů [3]. Také Sozzi et al uvedli, že po operačním zákroku došlo u pacientů k postupnému poklesu hladin cf‑DNA, které pak byly srovnatelné se zdravými kontrolami. Kvantifikace cf‑DNA může představovat nový postup monitorování úspěšnosti chirurgických zákroků a hodnocení účinnosti chemo/ radioterapie [42].
Nejčastěji jsou u karcinomu plic v cf‑DNA vyšetřovány mutace v genu pro K RAS, EGFR a p53. Gautschi et al analyzovali celkem 180 vzorků plazmy pacientů s plicním karcinomem a zjistili významnou korelaci mezi přítomností mutace v genu K RAS v cf‑DNA a špatnou prognózou [43]. Mutace v genu pro EGFR hrají významnou roli u nemalobuněčného karcinomu plic (non‑small cell lung cancer – NSCLC), který představuje až 80 % všech plicních malignit [44]. K prodloužení přežití pacientů s pokročilým stadiem NSCLC významně přispívá zavedení molekulárně cílené léčby tyrozinkinázovými inhibitory (TKI), které blokují aktivaci kaskády genu EGFR. Detekce aktivačních mutací genu EGFR, delece na 19. exonu a bodová mutace na 21. exonu jsou užitečné v předpovědi dobré odpovědi na léčbu TKI a progrese celkového přežití pacientů [45]. V léčbě NSCLC jsou běžně užívány TKI erlotinib a gefitinib.
Detekce mutací v genu EGFR v primárním nádoru vyžaduje biopsii nádorové tkáně, což v mnohých případech není proveditelné. Z těchto důvodů se do popředí zájmu dostává využití tekuté biopsie, detekce mutací v genu EGFR v cf‑DNA z periferní krve, jenž představuje minimálně invazivní přístup [46]. Využitím cf‑DNA se ve své studii zabývali Bai et al, kteří u 81 z 230 pacientů ve III. a IV. stadiu NSCLC detekovali přítomnost mutací v genu EGFR. Pacienti, u nichž byly přítomny mutace, na léčbu inhibitorem gefitinib reagovali mnohem lépe a vykazovali delší dobu přežití oproti pacientům, u kterých mutace v genu EGFR nebyly přítomny [46]. Toto zjištění potvrzuje Kuang et al, kteří prováděli studie u pacientů léčených gefitinibem a erlotinibem [47]. Mack et al se zabývali účinností léčby erlotinibem a docetaxelem u pacientů s aktivačními mutace v genu EGFR a zjistili, že docetaxel nesnižoval účinnost erlotinibu [48]. V další studii bylo uvedeno, že hladiny mutantních sekvencí v genu EGFR korelují s klinickou odpovědí pacienta. Vymizení mutace bylo detekováno u všech pacientů s částečnou nebo kompletní klinickou remisí, zatímco přetrvání mutace bylo zaznamenáno u pacientů s progresí nemoci [49]. Z uvedených poznatků tedy vyplývá, že přesná kvantifikace a citlivá detekce mutací v genu EGFR v cf‑DNA z periferní krve by byla užitečná pro predikci léčebné odpovědi, monitorování progrese nemoci a pro včasné odhalení selhání léčby spojené se získanou rezistencí. Přehled vybraných studií zabývající se kvantitativní a kvalitativní analýzou cf‑DNA u karcinomu plic je uveden v tab. 2 [42,43,46–49,50–62].
Table 2. Vybrané studie zabývající se kvantitativní a kvalitativní analýzou cf-DNA u karcinomu plic. 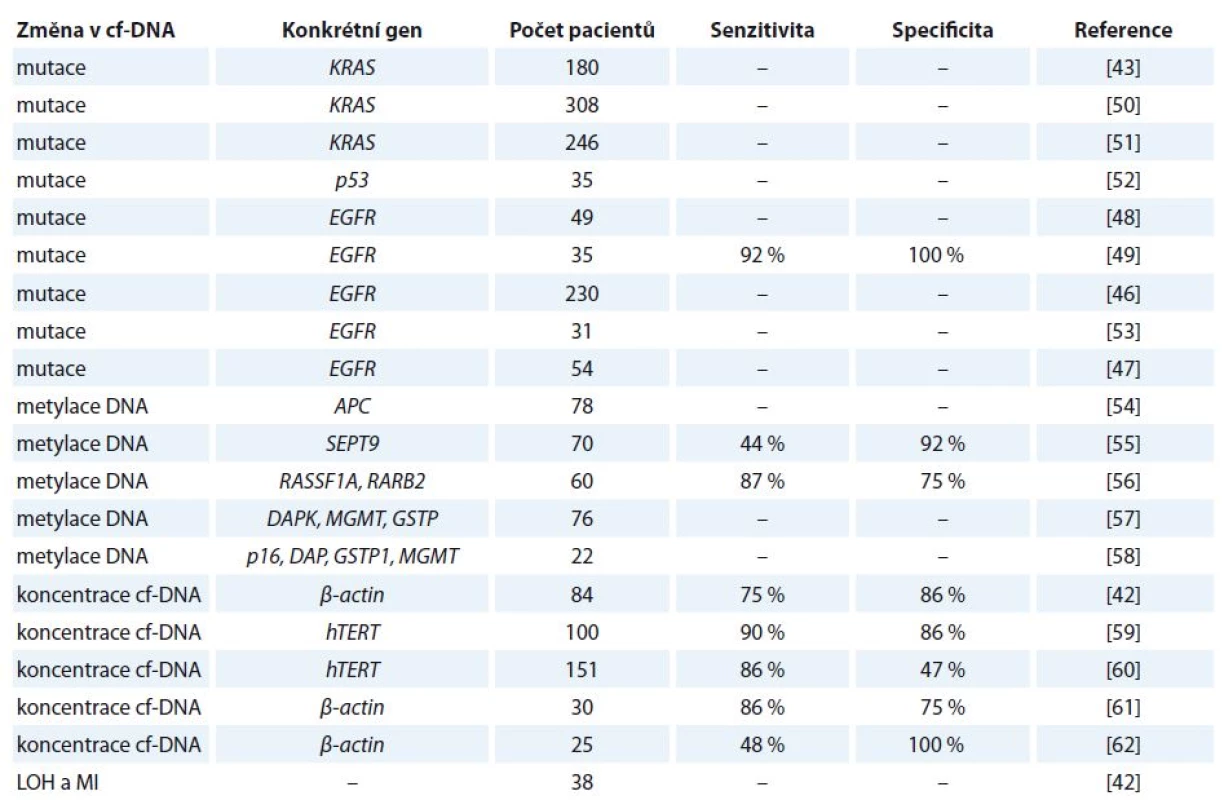
LOH – ztráta heterozygozity, MI – instabilita mikrosatelitů Karcinom prsu
V ČR je karcinom prsu nejčastějším zhoubným nádorem žen, jehož četnost se za posledních 20 let zdvojnásobila [20]. Přehled vybraných studií zabývající se využitím cf‑DNA u tohoto onemocnění je uveden v tab. 3 [63 – 83].
Table 3. Vybrané studie zabývající se kvantitativní a kvalitativní analýzou cf-DNA u karcinomu prsu. 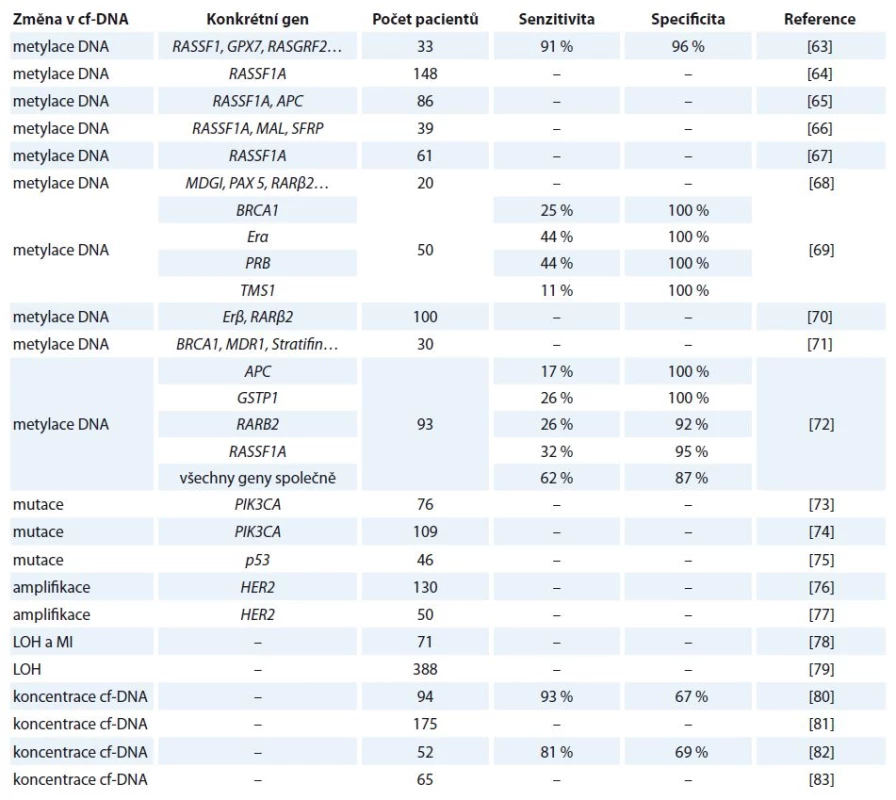
LOH – ztráta heterozygozity, MI – instabilita mikrosatelitů Kohler et al ve své studii uvedli, že hladiny cf‑DNA u pacientek s maligním nádorem prsu dosahovaly výrazně vyšších hodnot oproti pacientkám s benigním nádorem a zdravým ženám. Tento kvantitativní přístup by mohl sloužit k odlišení pacientek s maligním nádorem [82]. Catarino et al kvantifikovali cf‑DNA pomocí PCR v reálném čase a zjistili, že naměřené koncentrace byly vyšší před operací oproti naměřeným hodnotám po operačním zákroku. Tyto výsledky prokazují, že cf‑DNA by byla vhodným markerem monitorování nemoci. Hladiny navíc korelovaly se stadiem nemoci, velikostí nádoru a metastázemi do lymfatických uzlin [81].
Uvádí se, že onkogen PIK3CA je nejčastěji mutován u karcinomu prsu a nachází se u 40 % pacientek s touto diagnózou [84]. Ve své studii Board et al srovnávali přítomnost této mutace v cf‑DNA u 46 pacientek s metastázemi a u 30 pacientek s lokalizovaným nádorem. Zjistili, že tato mutace byla nalezena v 13 ze 46 vzorků plazmy (28 %) a v 10 ze 46 vzorků sér (21 %) pacientek s metastázemi, ale ani v jednom případě u pacientek s lokalizovaným operabilním nádorem [73]. V jiné studii byla tato mutace v cf‑DNA detekována téměř u 30 % pacientek v pokročilém stadiu karcinomu prsu [74]. Tyto poznatky jasně ukazují, že detekce mutace v genu pro PIK3CA v cf‑DNA je podstatně vyšší u pokročilejších forem nemoci.
K dalším genetickým změnám s potenciálním uplatněním při vyšetřování karcinomu prsu patří nestabilita mikrosatelitů a ztráta heterozygotnosti DNA [78,79]. V nedávné studii bylo prokázáno, že změny v nádorově supresorových genech TIG1, PTEN, CCND2, RB1 a BRCA1 v cf‑DNA jsou spojovány s agresivnější formou karcinomu. Zejména LOH markeru D12S1725 v genu CCND2 se může stát významným indikátorem nepříznivé prognózy [79]. Dehan et al ve své práci uvedli, že epigenetické změny jsou v nádorových buňkách změnami konstantními a mají podstatný vliv na časnou kancerogenezi a progresi nemoci [85]. Nejvíce zkoumanou epigenetickou změnou je metylace DNA, např. hypermetylace v genu RASSF1A a APC jsou spojovány s horší prognózou [41]. Detekce metylovaných míst v cf‑DNA představuje potenciální možnost posouzení stupně rizika pacientek s karcinomem prsu.
Hematologické malignity
V případě hematologických malignit jsou poznatky ohledně využití cf‑DNA na experimentální úrovni. Přehled studií zabývající se využitím cf‑DNA u vybraných hematologických malignit je uveden v tab. 4 [86 – 98].
Table 4. Vybrané studie zabývající se analýzou cf-DNA u pacientů s hematologickými malignitami. 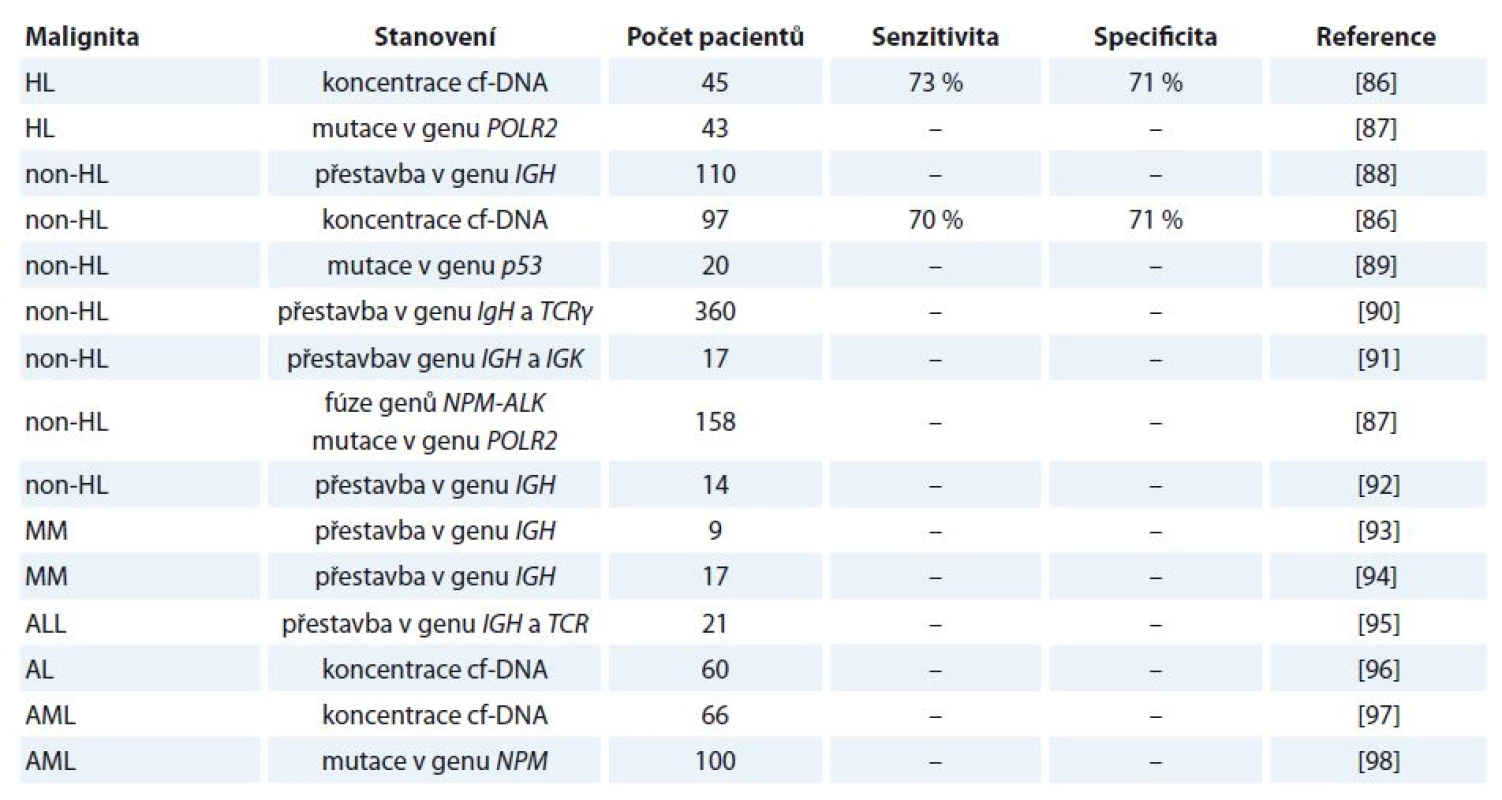
HL – Hodgkinův lymfom, non-HL – non-Hodgkinův lymfom, MM – mnohočetný myelom, ALL – akutní lymfoblastická leukemie, AL – akutní leukemie, AML – akutní myeloidní leukemie Několik studií bylo provedeno např. u B buněčných malignit. U pacientů s non‑Hodgkinovým lymfomem a akutní lymfoblastickou leukemií byla v cf‑DNA detekována přestavba těžkého imunoglobulinového řetězce. Nádorově odvozený klon DNA CDRIII byl nalezen v periferní krvi 52 (47 %) ze 110 pacientů. Z následně provedených odběrů byla patrná korelace mezi přetrvávajícím nádorově specifickým klonem DNA v séru/ plazmě a rezistencí k léčbě nebo brzkým relapsem nemoci. Výsledky ukazují, že nádorově specifická cf‑DNA může být detekována z periferní krve většiny pacientů s B buněčnými malignitami a může sloužit jako nový marker monitorování odpovědi na léčbu [88].
Také v jiné studii byla stanovena nádorová cf‑DNA u pacientů s agresivním B buněčným non‑Hodgkinovým lymfomem. U všech pacientů byl nalezen alespoň jeden dominantní nádorový klon v době diagnózy. S využitím nové techniky sekvenování byli autoři schopni identifikovat nádorový klon u 94 % pacientů s velkým difúzním B buněčným lymfomem a velkým mediastinálním B buněčným lymfomem. U 81 % těchto pacientů byla detekovatelná hladina cf‑DNA nádorového klonu v séru či mononukleárních buňkách periferní krve [91].
Další hematologickou malignitou, u které bylo testováno využití cf‑DNA, je mnohočetný myelom (multiple myeloma – MM). O tomto onemocnění byl na konferenci American Society of Hematology (ASH) prezentován abstrakt, který pojednával o cf‑DNA nesoucí specifickou sekvenci IgH klonu odvozeného od plazmatických buněk kostní dřeně. U pacientů byly detekovány přestavby v cf‑DNA v periferní krvi. Bylo zjištěno, že po nasazení kombinované chemoterapie hladiny cf‑DNA klesaly, hodnoty byly však v krvi tak nízké, že výsledky byly limitované citlivostí testu [93].
I přes významné pokroky v léčebných postupech dochází u nejméně 50 % pacientů s MM k relapsu onemocnění. Z těchto důvodů je hledán vhodný minimálně invazivní marker z periferní krve umožňující časnou detekci molekulárního relapsu, a tím i zahájení léčby. I na našem pracovišti probíhá výzkum klinického využití cf‑DNA v monitorování minimální reziduální choroby u pacientů s MM. Výsledky studie byly prezentovány na mezinárodní konferenci ASH v roce 2014. Kromě jiného se ukázalo, že nejpočetněji byly v séru zastoupeny DNA frakce o délce 180 – 220 bp, zatímco delší fragmenty byly častěji přítomny ve vzorcích odebraných v různých časových bodech léčby [94].
Závěr
Detekce cf‑DNA neboli tzv. tekutá biopsie (liquid biopsy) představuje nový, minimálně invazivní nástroj nabízející nové možnosti v diagnostice různých druhů maligních nádorů. Cf‑DNA skýtá obrovský potenciál v časném rozpoznání nádorů, v prognóze a monitorování nemoci. Představuje zejména možnost vytvoření personalizované medicíny, a tím zlepšení kvality života pacientů. Stále je však nutné provedení dalších studií s větší kohortou pacientů, které povedou ke zlepšení klinického využití cf‑DNA.
Práce byla podpořena grantem IGA MZ ČR NT14575.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
Obdrženo: 13. 3. 2015
Přijato: 18. 5. 2015
RNDr. Sabina Ševčíková, Ph.D.
Babákova myelomová skupina
Ústav patologické fyziologie
LF MU
Kamenice 5, A3
625 00 Brno
e-mail: sevcik@med.muni.cz
Sources
1. Jahr S, Hentze H, Englisch S et al. DNA fragments in the blood plasma of cancer patients: quantitations and evidence for their origin from apoptotic and necrotic cells. Cancer Res 2001; 61(4): 1659 – 1665.
2. Mandel P, Metais P. Les acides nucleiques du plasma sanguin chezl‘homme. C R Seances Soc Biol Fil 1948; 142(3 – 4): 241 – 243.
3. Leon SA, Shapiro B, Sklaroff DM et al. Free DNA in the serum of cancer patients and the effect of therapy. Cancer Res 1977; 37(3): 646 – 650.
4. Anker P, Stroun M. Circulating DNA in plasma or serum, Medicina (B Aires) 2000; 60(5 Pt 2): 699 – 702.
5. Stroun M, Lyautey J, Lederrey C et al. About the possible origin and mechanism of circulating DNA: apoptosis and active DNA release. Clin Chim Acta 2001; 313(1 – 2): 139 – 142.
6. Zeerleder S. The struggle to detect circulating DNA. Crit Care 2006; 10(3): 142.
7. Rumore PM, Steinman CR. Endogenous circulating DNA in systemic lupus erythematosus. Occurrence as multimeric complexes bound to histone. J Clin Invest 1990; 86(1): 69 – 74.
8. Zeerleder S, Zwart B, Wuillemin WA et al. Elevated nucleosome levels in systemic inflammation and sepsis. Crit Care Med 2003; 31(7): 1947 – 5191.
9. Lo YMD, Rainer TH, Chan LYS et al. Plasma DNA as a prognostic marker in trauma patients. Clin Chem 2000; 46(3): 319 – 323.
10. Rainer TH, Wong LK, Lam W et al. Prognostic use of circulating plasma nucleic acid concentrations in patients with acute stroke. Clin Chem 2003; 49(4): 562 – 569.
11. Shapiro B, Chakrabarty M, Cohn EM et al. Determination of circulating DNA levels in patients with benign or malignant gastrointestinal disease. Cancer 1983; 51(11): 2116 – 2120.
12. Holdenrieder S, Stieber P, Chan LY et al. Cell‑free DNA in serum and plasma:comparison of ELISA and quantitative PCR. Clin Chem 2005; 51(8): 1544 – 1546.
13. Diehl F, Li M, Dressman D et al. Detection and quantification of mutations in the plasma of patients with colorectal tumors. Proc Natl Acad Sci U S A 2005; 102(45): 16368 – 16373.
14. Stroun M, Maurice P, Vasioukhin V et al. The origin and mechanism of circulating DNA. Ann NY Acad Sci 2000; 906 : 161 – 168.
15. Jung K, Fleischhacker M, Rabien A. Cell‑free DNA in the blood as a solid tumor biomarker – a critical appraisal of the literature. Clin Chim Acta 2010; 411(21 – 22): 1611 – 1624.
16. Esposito A, Bardelli A, Criscitiello C et al. Monitoring tumor ‑ derived cell‑free DNA in patients with solid tumors: clinical perspectives and research opportunities. Cancer Treat Rev 2014; 40(5): 648 – 655. doi: 10.1016/ j.ctrv.2013.10.003.
17. Fiegl H, Millinger S, Mueller ‑ Holzner E et al. Circulating tumor ‑ specific DNA: a marker for monitoring efficacy of adjuvant therapy in cancer patients. Cancer Res 2005; 65(4): 1141 – 1145.
18. Gall TM, Frampton AE, Krell J et al. Cell‑free DNA for the detection of pancreatic, liver and upper gastrointestinal cancers: has progress been made? Future Oncol 2013; 9(12): 1861 – 1869. doi: 10.2217/ fon.13.152.
19. Diehl F, Schmidt K, Choti MA et al. Circulating mutant DNA to assess tumor dynamics. Nat Med 2008; 14(9): 985 – 990. doi: 10.1038/ nm.1789.
20. Dušek L, Mužík J, Kubásek M et al (eds). Epidemiologie zhoubných nádorů v České republice [monografie na Internetu]. Brno: Masarykova univerzita; 2005 [citováno 1. března 2015]. Dostupný z: http:/ / www.svod.cz.
21. Leung WK, To KF, Man EP et al. Quantitative detection of promoter hypermethylation in multiple genes in the serum of patients with colorectal cancer. Am J Gastroenterol 2005; 100(10): 2274 – 2279.
22. Wallner M, Herbst A, Behrens A et al. Methylation of serum DNA is an independent prognostic marker in colorectal cancer. Clin Cancer Res 2006; 12(24): 7347 – 7352.
23. Lofton ‑ Day C, Model F, Devos T et al. DNA methylation biomarkers for blood‑based colorectal cancer screening. Clin Chem 2008; 54(2): 414 – 423.
24. deVos T, Tetzner R, Model F et al. Circulating methylated SEPT9 DNA in plasma is a biomarker for colorectal cancer. Clin Chem 2009; 55(7): 1337 – 1346. doi: 10.1373/ clinchem.2008.115808.
25. Lefebure B, Charbonnier F, Di Fiore F et al. Prognostic value of circulating mutant DNA in unresectable metastatic colorectal cancer. Ann Surg 2010; 251(2): 275 – 280. doi: 10.1097/ SLA.0b013e3181c35c87.
26. Kopreski MS, Benko FA, Kwee C et al. Detection of mutant K ‑ ras DNA in plasma or serum of patients with colorectal cancer. Br J Cancer 1997; 76(10): 1293 – 1299.
27. Bettegowda C, Sausen M, Leary RJ et al. Detection of circulating tumor DNA in early ‑ and late ‑ stage human malignancies. Sci Transl Med 2014; 6(224): 224ra24. doi: 10.1126/ scitranslmed.3007094.
28. Trevisiol C, Di Fabio F, Nascimbeni R et al. Prognostic value of circulating KRAS2 gene mutations in colorectal cancer with distant metastases. Int J Biol Markers 2006; 21(4): 223 – 228.
29. Wong AL, Lim JS, Sinha A et al. Tumour pharmacodynamics and circulating cell free DNA in patients with refractory colorectal carcinoma treatedwith regorafenib. J Transl Med 2015; 13(1): 57. doi: 10.1186/ s12967 ‑ 015 ‑ 0405 ‑ 4.
30. Diehl F, Li M, Dressman D et al. Detection and quantification of mutations in the plasma of patients with colorectal tumors. Proc Natl Acad Sci U S A 2005; 102(45): 16368 – 16373.
31. Ito T, Kaneko K, Makino R et al. Clinical significance in molecular detection of p53 mutation in serum of patients with colorectal carcinoma. Oncol Rep 2003; 10(6): 1937 – 1942.
32. Lindforss U, Zetterquist H, Papadogiannakis N et al. Persistence of K ‑ ras mutations in plasma after colorectal tumor resection. Anticancer Res 2005; 25(1B): 657 – 661.
33. Kuo YB, Chen JS, Fan CW et al. Comparison of KRAS mutation analysis of primary tumors and matched circulating cell‑free DNA in plasmas of patients with colorectal cancer. Clin Chim Acta 2014; 433 : 284 – 289.
34. Morgan SR, Whiteley J, Donald E et al. Comparison of KRAS mutation assessment in tumor DNA and circulating free DNA in plasma and serum samples. Clin Med Insights Pathol 2012; 5 : 15 – 22. doi: 10.4137/ CPath.S8798.
35. Spindler KL, Pallisgaard N, Andersen RF et al. Circulating free DNA as biomarker and source for station detection in metastatic colorectal cancer. PLoS One 2015; 10(4): e0108247. doi: 10.1371/ journal.pone.0108247.
36. Diaz LA Jr, Williams RT, Wu J et al. The molecular evolution of acquired resistance to targeted EGFR blockade in colorectal cancers. Nature 2012; 486(7404): 537 – 540. doi: 10.1038/ nature11219.
37. Spindler KL, Pallisgaard N, Vogelius I et al. Quantitative cell‑free DNA, KRAS, and BRAF mutations in plasma from patients with metastatic colorectal cancerduring treatment with cetuximab and irinotecan. Clin Cancer Res 2012; 18(4): 1177 – 1185. doi: 10.1158/ 1078 - 0432.CCR ‑ 11 ‑ 0564.
38. Mouliere F, El Messaoudi S, Gongora C et al. Circulating cell‑free DNA from colorectal cancer patients may reveal high KRAS or BRAF mutation load. Transl Oncol 2013; 6(3): 319 – 328.
39. Frattini M, Gallino G, Signoroni S. Quantitative analysis of plasma DNA in colorectal cancer patients: a novel prognostic tool. Ann NY Acad Sci 2006; 1075 : 185 – 190.
40. Schwarzenbach H, Stoehlmacher J, Pantel K et al. Detection and monitoring of cell‑free DNA in blood of patients with colorectal cancer. Ann N Y Acad Sci 2008; 1137 : 190 – 196. doi: 10.1196/ annals.1448.025.
41. De Roock W, Biesmans B, De Schutter J et al. Clinical biomarkers in oncology: focus on colorectal cancer. Mol Diagn Ther 2009; 13(2): 103 – 114. doi: 10.2165/ 01250444 ‑ 200913020 ‑ 00004.
42. Sozzi G, Conte D, Mariani L et al. Analysis of circulating tumor DNA in plasma at diagnosis and during follow‑up of lung cancer patients. Cancer Res 2001; 61(12): 4675 – 4678.
43. Gautschi O, Huegli B, Ziegler A et al. Origin and prognostic value of circulating KRAS mutations in lung cancer patients. Cancer Lett 2007; 254(2): 265 – 273.
44. Govindan R, Page N, Morgensztern D et al. Changing epidemiology of small‑cell lung cancer in the United States over the last 30 years: analysis of the surveillance, epidemiologic, and end results database. J Clin Oncol 2006; 24(28): 4539 – 4544.
45. Gazdar AF. Activating and resistance mutations of EGFR in non‑small‑cell lung cancer: role in clinical response to EGFR tyrosine kinase inhibitors. Oncogene 2009; 28 (Suppl 1): S24 – S31. doi: 10.1038/ onc.2009.198.
46. Bai H, Mao L, Wang HS et al. Epidermal growth factor receptor mutations in plasma DNA samples predict tumor response in Chinese patients with stages IIIB to IV non‑small‑cell lung cancer. J Clin Oncol 2009; 27(16): 2653 – 2659. doi: 10.1200/ JCO.2008.17.3930.
47. Kuang Y, Rogers A, Yeap BY et al. Noninvasive detection of EGFR T790M in gefitinib or erlotinib resistant non‑small cell lung cancer. Clin Cancer Res 2009; 15(8): 2630 – 2636. doi: 10.1158/ 1078 ‑ 0432.CCR ‑ 08 ‑ 2592.
48. Mack PC, Holland WS, Burich RA et al. EGFR mutations detected in plasma are associated with patient outcomes in erlotinib plus docetaxel‑treated non‑small cell lung cancer. J Thorac Oncol 2009; 4(12): 1466 – 1472. doi: 10.1097/ JTO.0b013e3181bbf239.
49. Young TK, Chan KC, Mok TS et al. Single‑molecule detection of epidermalgrowth factor receptor mutations in plasma by microfluidics digital PCR in non‑small cell lung cancer patients. Clin Cancer Res 2009; 15(6): 2076 – 2084. doi: 10.1158/ 1078 ‑ 0432.CCR ‑ 08 ‑ 2622.
50. Camps C, Jantus ‑ Lewintre E, Cabrera A et al. The identification of KRAS mutations at codon 12 in plasma DNA is not a prognostic factor in advanced non‑small cell lung cancer patients. Lung Cancer 2011; 72(3): 365 – 369. doi: 10.1016/ j.lungcan.2010.09.005.
51. Nygaard AD, Garm Spindler KL, Pallisgaard N et al. The prognostic value of KRAS mutated plasma DNA in advanced non‑small cell lung cancer. Lung Cancer 2013; 79(3): 312 – 317. doi: 10.1016/ j.lungcan.2012.11.016.
52. Gonzalez R, Silva JM, Sanchez A et al. Microsatellite alterations and TP53 mutations in plasma DNA of small‑cell lung cancer patients: follow‑up study and prognostic significance. Ann Oncol 2000; 11(9): 1097 – 1104.
53. Brevet M, Johnson ML, Azzoli CG et al. Detection of EGFR mutations in plasma DNA from lung cancer patients by mass spectrometry genotyping is predictive of tumor EGFR status and response to EGFR inhibitors. Lung Cancer 2011; 73(1): 96 – 102. doi: 10.1016/ j.lungcan.2010.10.014.
54. Pan SY, Xie EF, Shu YQ et al. Methylation quantification of adenomatous polyposis coli (APC) gene promoter in plasma of lung cancerpatients. Ai Zheng 2009; 28(4): 384 – 389.
55. Powrózek T, Krawczyk P, Kucharczyk T et al. Septin 9 promoter region methylation in free circulating DNA ‑ potential role in noninvasive diagnosis of lung cancer: preliminary report. Med Oncol 2014; 31(4): 917. doi: 10.1007/ s12032 ‑ 014 ‑ 0917 ‑ 4.
56. Ponomaryova AA, Rykova EY, Cherdyntseva NV et al. Potentialities of aberrantly methylated circulating DNA for diagnostics and post‑treatment follow‑up of lung cancer patients. Lung Cancer 2013; 81(3): 397 – 403. doi: 10.1016/ j.lungcan.2013.05.016.
57. Hoffmann AC, Kaifi JT, Vallböhmer D et al. Lack of prognostic signifikance of serum DNA methylation of DAPK, MGMT, and GSTPI in patients with non‑small cell lung cancer. J Surg Oncol 2009; 100(5): 414 – 417. doi: 10.1002/ jso.21348.
58. Esteller M, Sanchez ‑ Cespedes M, Rosell R et al. Detection of aberrant promoter hypermethylation of tumor suppressor genes in serum DNA from non‑small cell lung cancer patients. Cancer Res 1999; 59(1): 67 – 70.
59. Sozzi G, Conte D, Leon M et al. Quantification of free circulating DNA as a diagnostic marker in lung cancer. J Clin Oncol 2003; 21(21): 3902 – 3908.
60. Paci M, Maramotti S, Bellesia E et al. Circulating plasma DNA as diagnostic biomarker in non‑small cell lung cancer. Lung Cancer 2009; 64(1): 92 – 97. doi: 10.1016/ j.lungcan.2008.07.012.
61. Szpechcinski A, Dancewicz M, Kopinski P et al. Real ‑ time PCR quantification of plasma DNA in non‑small cell lung cancer patients and healthy controls. Eur J Med Res 2009; 14 (Suppl 4): 237 – 240.
62. Herrera LJ, Raja S, Gooding WE et al. Quantitative analysis of circulating plasma DNA as a tumor marker in thoracic malignancies. Clin Chem 2005; 51(1): 113 – 118.
63. Fackler MJ, Lopez Bujanda Z, Umbricht C et al. Novel methylated biomarkers and a robust assay to detect circulating tumor DNA in metastatic breast. Cancer Res 2014; 74(8): 2160 – 2170.
64. Fiegl H, Millinger S, Mueller ‑ Holzner E et al. Circulating tumor ‑ specific DNA: a marker for monitoring efficacy of adjuvant therapy in cancer patients. Cancer Res 2005; 65(4): 1141 – 1145.
65. Muller HM, Widschwendter A, Fiegl H et al. DNA methylation in serum of breast cancer patients: an independent prognostic marker. Cancer Res 2003; 63(22): 7641 – 7645.
66. Agostini M, Enzo MV, Bedin C et al. Circulating cell‑free DNA: a promising marker of regional lymphonode metastasis in breast cancer patients. Cancer Biomark 2012; 11(2 – 3): 89 – 98.
67. Yazici H, Terry MB, Cho YH et al. Aberrant methylation of RASSF1A in plasma DNA before breast cancer diagnosis in the Breast Cancer Family Registry. Cancer Epidemiol Biomarkers Prev 2009; 18(10): 2723 – 2725. doi: 10.1158/ 1055 ‑ 9965.EPI ‑ 08 ‑ 1237.
68. Liggett TE, Melnikov AA, Marks JR et al. Methylation patterns in cell‑free plasma DNA reflect removal of the primary tumor and drug treatment of breast cancer patients. Int J Cancer 2011; 128(2): 492 – 499. doi: 10.1002/ ijc.25363.
69. Mirza S, Sharma G, Prasad CP et al. Promoter hypermethylation of TMS1, BRCA1, ERalpha and PRB in serum and tumor DNA of invasive ductal breast carcinoma patients. Life Sci 2007; 81(4): 280 – 287.
70. Mirza S, Sharma G, Parshad R et al. Clinical significance of promoter hypermethylation of ERβ and RARβ2 in tumor and serum DNA in Indian breast cancer patients. Ann Surg Oncol 2012; 19(9): 3107 – 3115. doi: 10.1245/ s10434 ‑ 012 ‑ 2323 ‑ 5.
71. Sharma G, Mirza S, Parshad R et al. DNA methylation of circulating DNA: a marker for monitoring efficacy of neoadjuvant chemotherapy in breast cancer patients. Tumour Biol 2012; 33(6): 1837 – 1843. doi: 10.1007/ s13277 ‑ 012 ‑ 0443 ‑ y.
72. Hoque MO, Feng Q, Toure P et al. Detection of aberrant methylation of four genes in plasma DNA for the detection of breast cancer. J Clin Oncol 2006; 24(26): 4262 – 4269.
73. Board RE, Wardley AM, Dixon JM et al. Detection of PIK3CA mutations in circulating free DNA in patients with breast cancer. Breast Cancer Res Treat 2010; 120(2): 461 – 467. doi: 10.1007/ s10549 ‑ 010 ‑ 0747 ‑ 9.
74. Higgins MJ, Jelovac D, Barnathan E et al. Detection of tumor PIK3CA status in metastatic breast cancer using peripheral blood. Clin Cancer Res 2012; 18(12): 3462 – 3469.
75. Shao ZM, Wu J, Shen ZZ et al. p53 mutation in plasma DNA and its prognostic value in breast cancer patients. Clin Cancer Res 2001; 7(8): 2222 – 2227.
76. Page K, Hava N, Ward B et al. Detection of HER2 amplification in circulating free DNA in patients with breast cancer. Br J Cancer 2011; 104(8): 1342 – 1348. doi: 10.1038/ bjc.2011.89.
77. Bechmann T, Andersen RF, Pallisgaard N et al. Plasma HER2 amplification in cell‑free DNA during neoadjuvant chemotherapy in breast cancer. J Cancer Res Clin Oncol 2013; 139(6): 995 – 1003. doi: 10.1007/ s00432 ‑ 013 ‑ 1413 ‑ 5.
78. Shaw JA, Smith BM, Walsh T et al. Microsatellite alterations plasma DNA of primary breast cancer patients. Clin Cancer Res 2000; 6(3): 1119 – 1124.
79. Schwarzenbach H, Eichelser C, Kropidlowski J et al. Loss of heterozygosity at tumor suppressor genes detectable on fractionated circulating cell‑free tumor DNA as indicator of breast cancer progression. Clin Cancer Res 2012; 18(20): 5719 – 5730. doi: 10.1158/ 1078 ‑ 0432.CCR ‑ 12 ‑ 0142.
80. Huang ZH, Li LH, Hua D. Quantitative analysis of plasma circulating DNA at diagnosis and during follow‑up of breast cancer patients. Cancer Lett 2006; 243(1): 64 – 70.
81. Catarino R, Ferreira MM, Rodrigues H et al. Quantification of free circulating tumor DNA as a diagnostic marker for breast cancer. DNA Cell Biol 2008; 27(8): 415 – 421. doi: 10.1089/ dna.2008.0744.
82. Kohler C, Radpour R, Barekati Z et al. Levels of plasma circulating cell free nuclear and mitochondrial DNA as potential biomarkers for breast tumors. Mol Cancer 2009; 8 : 105. doi: 10.1186/ 1476 ‑ 4598 ‑ 8 ‑ 105.
83. Nicolini C, Ens C, Cerutti T et al. Elevated level of cell free plasma DNA is associated with advanced stage breast cancer and metastasi. Clin Chem Lab Med 2013; 51(11): 277 – 278. doi: 10.1515/ cclm ‑ 2013 ‑ 0120.
84. Campbell IG, Russell SE, Choong DY et al. Mutation of the PIK3CA gene in ovarian and breast cancer. Cancer Res 2004; 64(21): 7678 – 7681.
85. Dehan P, Kustermans G, Guenin S et al. DNA methylation and cancer diagnosis: new methods and applications. Expert Rev Mol Diagn 2009; 9(7): 651 – 657. doi: 10.1586/ erm.09.53.
86. Hohaus S, Giachelia M, Massini G et al. Cell‑free circulating DNA in Hodgkin‘s and non‑Hodgkin‘s lymphomas. Ann Oncol 2009; 20(8): 1408 – 1413. doi: 10.1093/ annonc/ mdp006.
87. Mussolin L, Burnelli R, Pillon M et al. Plasma cell‑free DNA in paediatric lymphomas. J Cancer 2013; 4(4): 323 – 329. doi: 10.7150/ jca.6226.
88. Frickhofen N, Müller E, Sandherr M et al. Rearranged Ig heavy chain DNA is detectable in cell‑free blood samples of patients with B ‑ cell neoplasia. Blood 1997; 90(12): 4953 – 4960.
89. Hosny G, Farahat N, Hainaut P. TP53 mutations in circulating free DNA from Egyptian patients with non‑Hodgkin‘s lymphoma. Cancer Lett 2009; 275(2): 234 – 239. doi: 10.1016/ j.canlet.2008.10.029.
90. Zhong L, Huang WF. Better detection of Ig heavy chain and TCRγ gene rearrangement in plasma cell‑free DNA from patients with non‑Hodgkin lymphoma. Neoplasma 2010; 57(6): 507 – 511.
91. Armand P, Oki Y, Neuberg DS et al. Detection of circulating tumour DNA in patients with aggressive B ‑ cell non‑Hodgkin lymphoma. Br J Haematol 2013; 163(1): 123 – 126. doi: 10.1111/ bjh.12439.
92. He J, Wu J, Jiao Y et al. IgH gene rearrangements as plasma biomarkers in non ‑ Hodgkin‘s lymphoma patients. Oncotarget 2011; 2(3): 178 – 185.
93. Kurlander R, LI Y, Stetler ‑ Stevenson M et al. Evaluation of circulating cell ‑ free VDJ DNA as a marker for monitoring patients with multiple myeloma (MM) during treatment with carfilzomib, lenalidomide and dexamethasone. In: American Society of Hematology 2013 Annual Meeting. Abstr. 1868 (Poster Presentation).
94. Kubiczkova ‑ Besse L, Drandi D, Sedlarikova L et al. Cell‑free DNA for minimal residual disease monitoring in multiple myeloma. In: American Society of Hematology 2014 Annual Meeting. Abstract 3423 (Poster Presentation).
95. Schwarz AK, Stanulla M, Cario G et al. Quantification of free total plasma DNA and minimal residual disease detection in the plasma of children with acute lymphoblastic leukemia. Ann Hematol 2009; 88(9): 897 – 905. doi: 10.1007/ s00277 ‑ 009 ‑ 0698 ‑ 6.
96. Gao YJ, He YJ, Yang ZL et al. Increased integrity of circulating cell‑free DNA in plasma of patients with acute leukemia. Clin Chem Lab Med 2010; 48(11): 1651 – 1656. doi: 10.1515/ CCLM.2010.311.
97. Jiang Y, Pan SY, Xia WY et al. Dynamic monitoring of plasma circulating DNA in patients with acute myeloid leukemia and its clinical significance. Zhongguo Shi Yan Xue Ye Xue Za Zhi 2012; 20(1): 53 – 56.
98. Quan J, Gao YJ, Yang ZL et al. Quantitative detection of circulating nucleophosmin mutations DNA in the plasma of patients with acute myeloidleukemia. Int J Med Sci 2015; 12(1): 17 – 22. doi: 10.7150/ ijms.10144.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2015 Issue 4-
All articles in this issue
- Moderní nanomedicína v léčbě karcinomů plic
- Potenciál volné cirkulující DNA v diagnostice nádorových onemocnění
- Možnosti inhibice receptoru pro epidermální růstový faktor u karcinomu anu
- Analýza nákladové efektivity porovnávající panitumumab plus mFOLFOX6 a bevacizumab plus mFOLFOX6 v první linii metastatického kolorektálního karcinomu s expresí nemutovaného typu onkogenu RAS – adaptace modelu na podmínky České republiky
- Extraoseální Ewingův sarkom, primární postižení děložního čípku – kazuistika
- Embryonálne tumory s mnohovrstvovými rozetami – vzácne tumory centrálneho nervového systému v detskom veku
- Antikoagulační léčba a trombembolizmus při léčbě bevacizumabem – opatrnost, či obavy?
- Odhadovaná glomerulární filtrace u onkologických pacientů před chemoterapií cisplatinou
- Incidencia a prognostický význam známych genetických aberácií u pacientov s diagnózou akútnej myeloblastovej leukémie – dvojročná štúdia
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Extraoseální Ewingův sarkom, primární postižení děložního čípku – kazuistika
- Embryonálne tumory s mnohovrstvovými rozetami – vzácne tumory centrálneho nervového systému v detskom veku
- Potenciál volné cirkulující DNA v diagnostice nádorových onemocnění
- Odhadovaná glomerulární filtrace u onkologických pacientů před chemoterapií cisplatinou
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

