-
Medical journals
- Career
Využití metylačního profilu v diagnostice a prognostice nádorových onemocnění CNS
Authors: Aleš Vícha 1,4; Lucie Štolová 1; Pavla Jenčová 1; Michal Zápotocký 1,4; David Sumerauer 1,4; Adéla Mišove 1,4; Miroslav Koblížek 2,4; Petr Brož 1,3; Josef Zámečník 2,4; Martin Kynčl 5; Petr Libý 6; Lenka Krsková 2,4
Authors‘ workplace: Klinika dětské hematologie a onkologie 2. LF UK a FN Motol, Praha 1; Ústav patologie a molekulární medicíny 2. LF UK a FN Motol, Praha 2; Bioxsys, Ústí nad Labem 3; Prague Brain Tumour Research Group 4; Klinika zobrazovacích metod 2. LF UK a FN Motol, Praha 5; Neurochirurgická klinika dětí a dospělých 2. LF UK a FN Motol, Praha 6
Published in: Čes.-slov. Patol., 57, 2021, No. 3, p. 154-160
Category: Reviews Article
Overview
Vyšetření změny v metylačním profilu DNA u nádorových onemocnění je v současné době využíváno ke stanovení diagnózy nebo prognostických a prediktivních biomarkerů. Doplňuje významně histologická nebo molekulárně biologická vyšetření. Zároveň napomáhá při stanovení přesnějších diagnostických skupin a podskupin. V současné době je tato diagnostika nejvíce rozšířena u nádorů CNS, kde se již stala rutinním vyšetřením. Stanovení metylačního profilu může napomoci i tam, kde nelze jiným způsobem stanovit diagnózu nebo podskupinu onemocnění, jak je tomu například u meduloblastomu.
Klíčová slova:
metylace – SNP array – nádory CNS – klasifikace
Přesná patologická diagnostika je základem léčby pacientů s nádorovým onemocněním. Morfologické vyšetření v kombinaci s imunohistochemickým vyšetřením je v patologické diagnostice klíčové, ale čím dál častěji jsou využívány i metody molekulární biologie a genetiky. Takto rozšířené spektrum analýzy onemocnění napomáhá především tam, kde může být přesné stanovení diagnózy na základě histologického vyšetření obtížné a také ke stanovení podskupiny daného onemocnění, které je založeno na jeho molekulární klasifikaci, případně pro stanovení prognostických znaků, nebo terapeutických cílů (1). Molekulární biologie tím napomáhá ke zpřesnění diagnostiky a prognostiky nádorových onemocnění. Stále častěji se do této mozaiky vyšetření zařazuje i zhodnocení epigenetických změn (1–10).
Epigenetické změny, jako je metylace DNA, acetylace a metylace histonů, jsou příčinou přirozených biologických procesů, jako je stárnutí nebo diferenciace buněk. Buňky jednotlivých tkání mají, až na výjimky, stejnou genetickou výbavu, ačkoliv vykonávají specifickou funkci. Rozdílná exprese pro - teinů v jednotlivých typech tkání je regulována především pomocí epigenetických změn. Změny celkového epigenetického profilu, spolu s genetickými alteracemi, jsou prokazovány u některých onemocnění, včetně těch nádorových (11–14). Mezi tyto epigenetické změny patří metylace DNA. Regulace genové exprese proteinů na základě metylace je zajištěna změnou metylace v regulačních oblastech genů nazývaných CpG ostrůvky (obr. 1). Profily metylace DNA během embryonálního vývoje se liší od profilů metylace v dospělosti. Podobně vzorec metylace DNA normálních buněk dané tkáně se liší od metylačního profilu získaného z nádorových buněk této tkáně (15). Nádorový metylační profil je tedy kombinací somaticky získaných změn a změn, které jsou charakteristické pro danou tkáň organismu (15–17). Metylační profil získaný při vyšetření nádoru může stanovit jednotlivé typy nebo podtypy nádorových onemocnění, která ve tkáni vznikají (1,4,6, 7,17–26). Sledování změn v profilu metylace DNA u nádorů je v současné době využíváno při diagnostice nádorových onemocnění v kombinaci s průkazem změn chromozomálních (1,3,4,6–9,17–30). Takto lze prokázat například kodeleci 1p/19q (obr. 2), amplifikaci MYC, MYCN, EGFR, PDGFRA, MDM2, nebo deleci CDKN2A/2B, Rb1 a další typické změny v nádorech. Lze také stanovit metylaci na jednotlivých genech např. metylaci promotorové oblasti genu MGMT, která se využívá k výběru terapie (29). Vyšetření metylačního profilu, při kterém zároveň vyšetříme chromozomální změny (delece, zmnožení a amplifikace), může nahradit další vyšetření, jako jsou FISH, MLPA, aCGH nebo SNP array a přinést více informací. Výhodou je i to, že u postižení CNS lze vyšetření metylačního profilu využít i pro určení původu metastáz tam, kde to nelze histologicky spolehlivě stanovit (31). V současnosti se k vyšetření metylačního profilu užívají nejčastěji techniky metylační NGS a metylační SNP (SNP array, technika založená na vyšetření jednotlivých snp polymorfismů a metylačního stavu na těchto polymorfismech). Metylační SNP se v klinické praxi používá déle a je metodou vhodnou pro vyšetření metylačního profilu proto, že se jedná o techniku velice robustní, kterou lze použít i u malých vzorků se zhoršenou kvalitou materiálu (FFPE vzorky) (17). Tato technika umožňuje vyšetření až 450 tisíc (450K) nebo 850 tisíc (EPIC) metylačních míst podle typu zvoleného kitu (K450 nebo EPIC, Illumina).
Image 1. Ovlivnění exprese prostřednictvím metylace CpG ostrůvků. 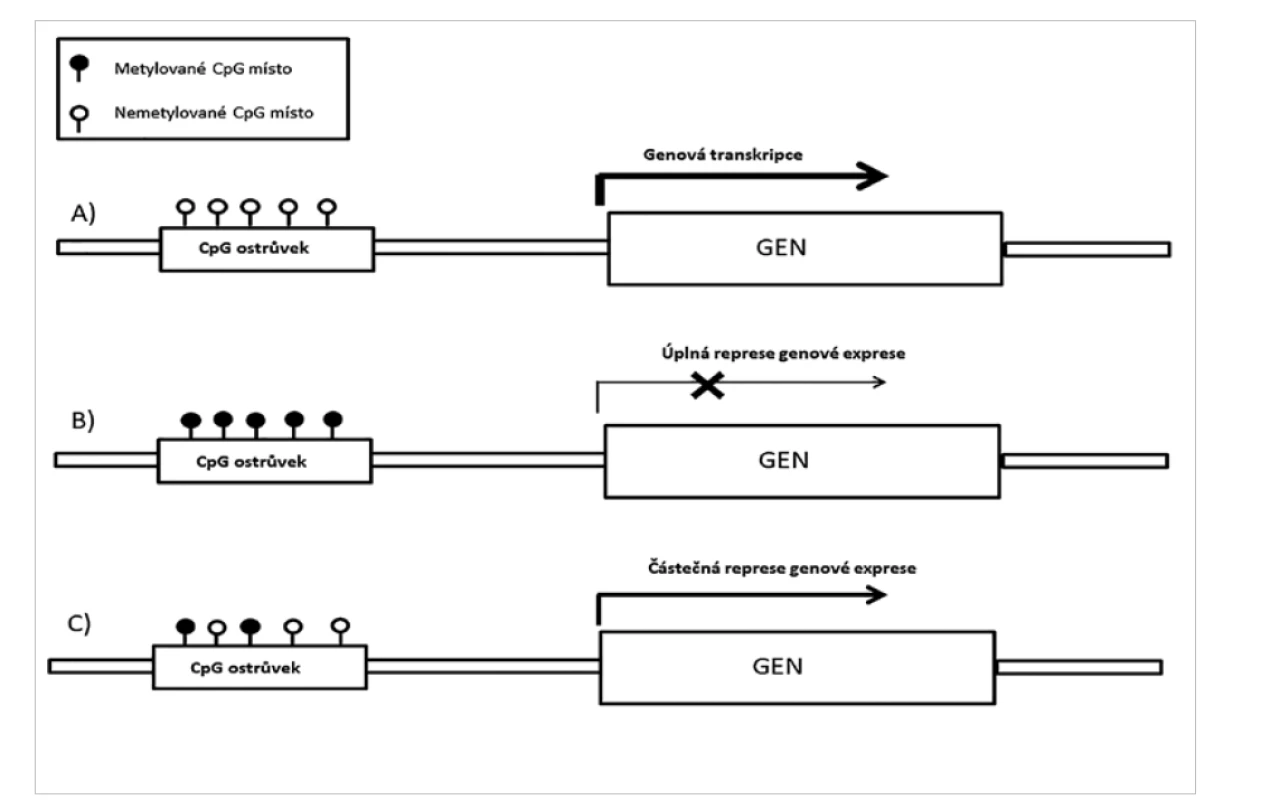
Image 2. Výsledek vyšetření metylační SNP s průkazem kodelece1p/19q. Vyšetření metylační SNP (illumina EPIC) hodnocenoBrain_v11b4_ sample Version 2.0 určený pro nádory CNS na (www.molecularneuropathology.org), které prokázalo deleci 1p, deleci 14q, deleci 19q a gain 22q.
Červeně jsou značeny delece, zeleně zisky.
VYUŽITÍ METYLAČNÍHO PROFILU V DIAGNOSTICE NÁDORŮ CNS
Morfologická diagnostika nádorů CNS může být velice obtížná, často ani imunohistochemie nepomůže tumor přesně zařadit. Proto se diagnostika nádorů CNS stále častěji skládá, podobně jako mozaika, z jednotlivých vyšetření, která kromě morfologického a imunohistochemického vyšetření zahrnuje i zobrazovací vyšetření. Nejčastěji je to MRI, která poskytuje informaci o lokalizaci a charakteru tumoru, dále posouzení klinického obrazu a v poslední době se stále častěji v rutinní praxi používají i metody molekulární biologie a genetiky. Nově zaváděnou technikou je stanovení metylačního profilu nádoru (3,6,7,10,17–20,23,24,29,30,32–45). K zařazení nádoru pomocí stanoveného metylačního profilu a CNV se u mozkových nádorů nejčastěji využívá klasifikátor Brain_v11b4_sample Version 2.0 určený pro nádory CNS (www.molecularneuropathology. org) (3,29,45). Tento klasifikátor vznikl na základě práce Capper a kol. (29). Jejich snahou bylo vytvořit komplexní přístup ke klasifikaci nádorů centrálního nervového systému na základě metylace DNA, v korelaci s histologickým obrazem, klinickými parametry, jako je věk, pohlaví atd., který by bylo možné využít v rutinní klinické praxi (3,29). Proto autoři vybrali do referenční kohorty 2800 nádorů (nejméně 8 z každé skupiny), které odpovídají jednotkám téměř všech neuroektodermálních nádorů a nádorům v oblasti sely podle WHO klasifikace (46) a stanovili u nich metylační profil (3). Následně ověřili, zda se jednotlivé, předem definované jednotky, shlukují (klastrují) podle svého metylačního profilu na základě bioinformatického klastrování, které přiřadí vzorky do jednotlivých skupin na základě podobnosti metylačních profilů. Tak byl vytvořen základ dnes používaného klasifikátoru Brain_v11b4_sample Version 2.0. Tento systém není uzavřen a přidáváním dalších metylačních profilů se klasifikátor nadále zpřesňuje. Díky více než 7 tisícům metylačních profilů, které jsou nyní obsaženy v klasifikátoru, je možné rozlišit 81 typů nádorů CNS (www.molecularneuropathology. org). V budoucnu se předpokládá zařazení dalších jednotek tak, jak tomu bylo u meduloblastomu (45).
VYUŽITÍ METYLAČNÍHO PROFILU V DIAGNOSTICE MEDULOBLASTOMU
Meduloblastom patří mezi nádorová onemocnění, u nichž diagnostika, stratifikace na podkladě rizikových faktorů a klinických parametrů, vykazuje velmi rychlý rozvoj. Meduloblastom je solidním nádorem, který ukazuje, jak efektivní může být spojení morfologie a molekulárně genetických dat ke stanovení klinicko-patologického rizika onemocnění, jež se uplatní při přípravě inovativních klinických studií, včetně cílené terapie (47). Již v roce 2005 byla prokázána přítomnost patogenní změny v genu pro beta-katenin (CTNNB1), která je spojena s delecí 6. chromozomu, většinou bez přítomnosti dalších chromozomálních změn (48). V dalším období byla snaha rozčlenit meduloblastomy do podskupin pomocí expresních profilů. Tyto studie na základě expresního profilu prokazovaly přítomnost 4 až 6 podskupin meduloblastomu (49–52). V roce 2012 bylo dosaženo mezinárodního konsensu a byly ustanoveny 4 skupiny meduloblastomu, které zahrnují skupinu WNT, SHH (TP53 pozitivní/negativní), non WNT/SHH (skupinu 3 a skupinu 4) meduloblastomu (charakteristika skupin viz tab. 1; upraveno podle (53))(48,54). V následných studiích byl stanoven biologický a klinický význam této nové klasifikace, zároveň byly stanoveny molekulárně biologické techniky vhodné pro zařazení meduloblastomu do jedné ze stanovených skupin (tab 1.), ať už se jedná o expresní stanovení na úrovni mRNA nebo pomocí stanovení metylačního profilu na úrovni DNA (17, 21, 55–58). Skupina 3 a 4 se dále dělí do 8 podskupin (45, 59). Ty je možné vyšetřit pomocí klasifikátoru medulloblastomaclassifiergroup 3/4 v.1.0 (https://www.molecularneuropathology.org/mnp/ classifier/7).
Table 1. Charakteristika skupin meduloblastomu. 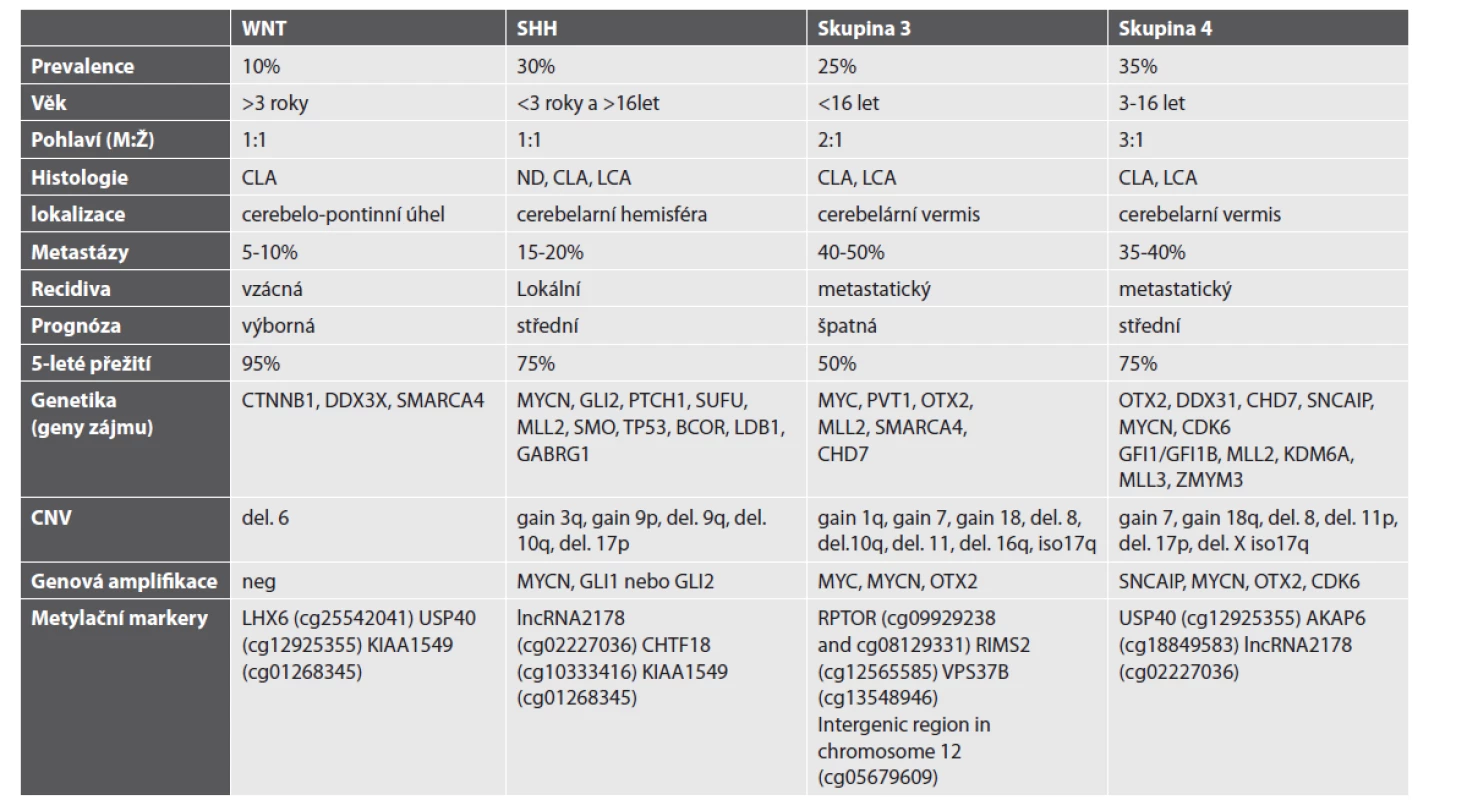
Použité zkratky: CLA – klasický typ, ND – nodulární desmoplastický typ, LCA – velkobuněčný anaplastický typ, del. – delece. Upraveno podle (53) Využití metylační SNP prezentujeme na příkladu čtyř pacientů s meduloblastomem (WNT, SHH, skupina 3 a skupina 4) léčených ve FN v Motole. Histologický a molekulárně biologický nález je shrnut v tabulce 2. Rentgenologický nález (MRI) je prezentován na obr. 3 a histologický nález pak na obr. 4.
Table 2. Klinický, histologický a molekulárně biologický nález pacientů s meduloblastomem. 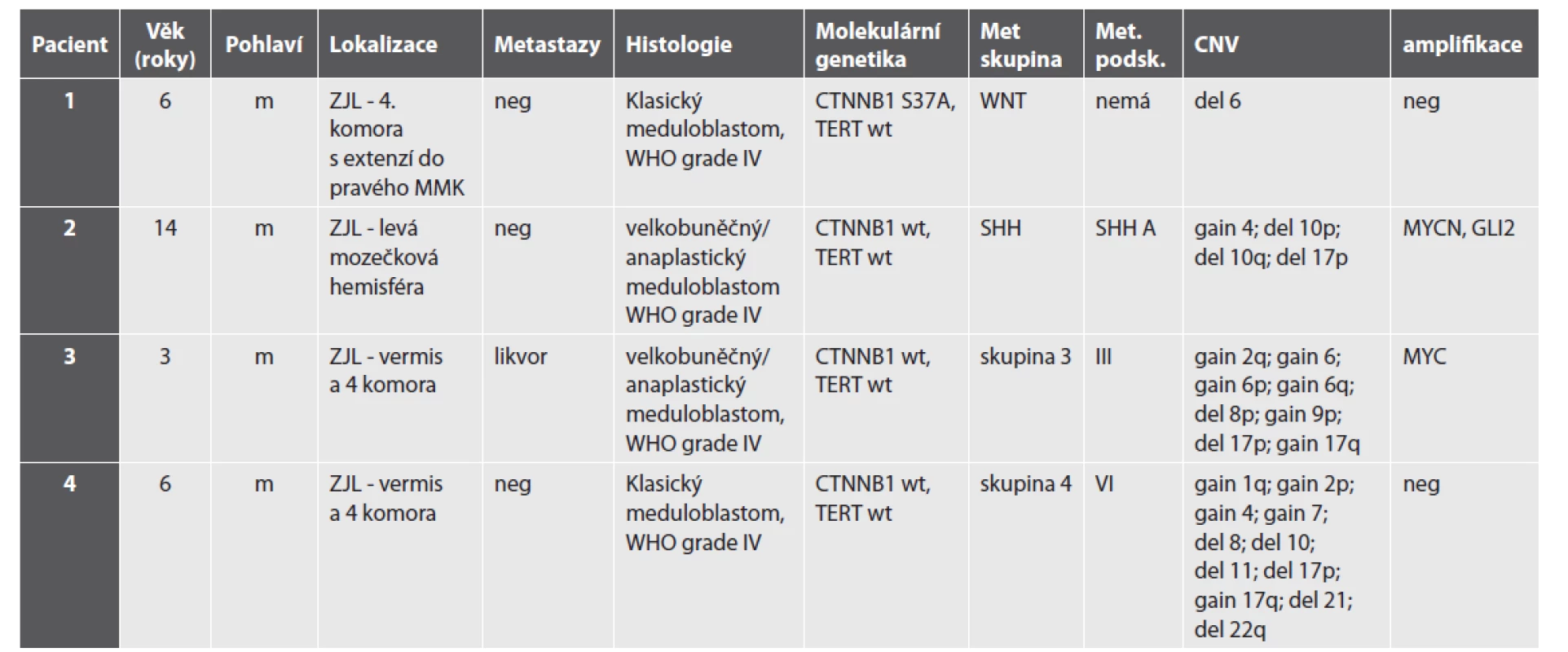
Použité zkratky: ZJL – zadní jáma lební, m – mužské, Met skupina – metylační skupina, Met. podsk. – metylační podskupina, CNV – chromozomální změny, neg. – negativní Image 3. Zobrazení MRI charakteristik jednotlivých podskupin meduloblastomu. A – Wnt-aktivovaný meduloblastom s extenzí do mostomozečkového koutu vpravo a do mozečkového pedunklu. B – SHH – aktivovaný meduloblastom lokalizovaný v levé mozečkové hemisféře. Takové laterální uložení je charakteristické pro SHH meduloblastom. C – Skupina 3 – tumor uložený ve vermis a 4. mozkové komoře s výrazným homogenním sycením po podání kontrastní látky. D – Skupina 4 – uložený ve vermis a 4. mozkové komoře s absencí sycení po podání kontrastní látky, což je charakteristika typická pro meduloblastom Skupiny 4. 
Image 4. Histologický nález pacientů s meduloblastomem. Pacient 1- Klasický meduloblastom tvořený hustě nakupenými atypickými protáhlými buňkami s oválnými jádry. Pacient 2 - Velkobuněčný/anaplastický meduloblastom tvoří kromě menších atypických buněk i četné obrovské buňky s výrazně nepravidelnými jádry. Pacient 3- Velkobuněčný/anaplastický meduloblastom je složený jednak z oblastí vzhledu klasického meduloblastomu, ale převažují úseky tvořené většími polygonálními buňkami s anaplastickými rysy a s velkými jádry místy s patrnými jadérky. Pacient 4- Klasický meduloblastom tvořený atypickými protáhlými buňkami, které místy naznačeně tvoří rytmické struktury. 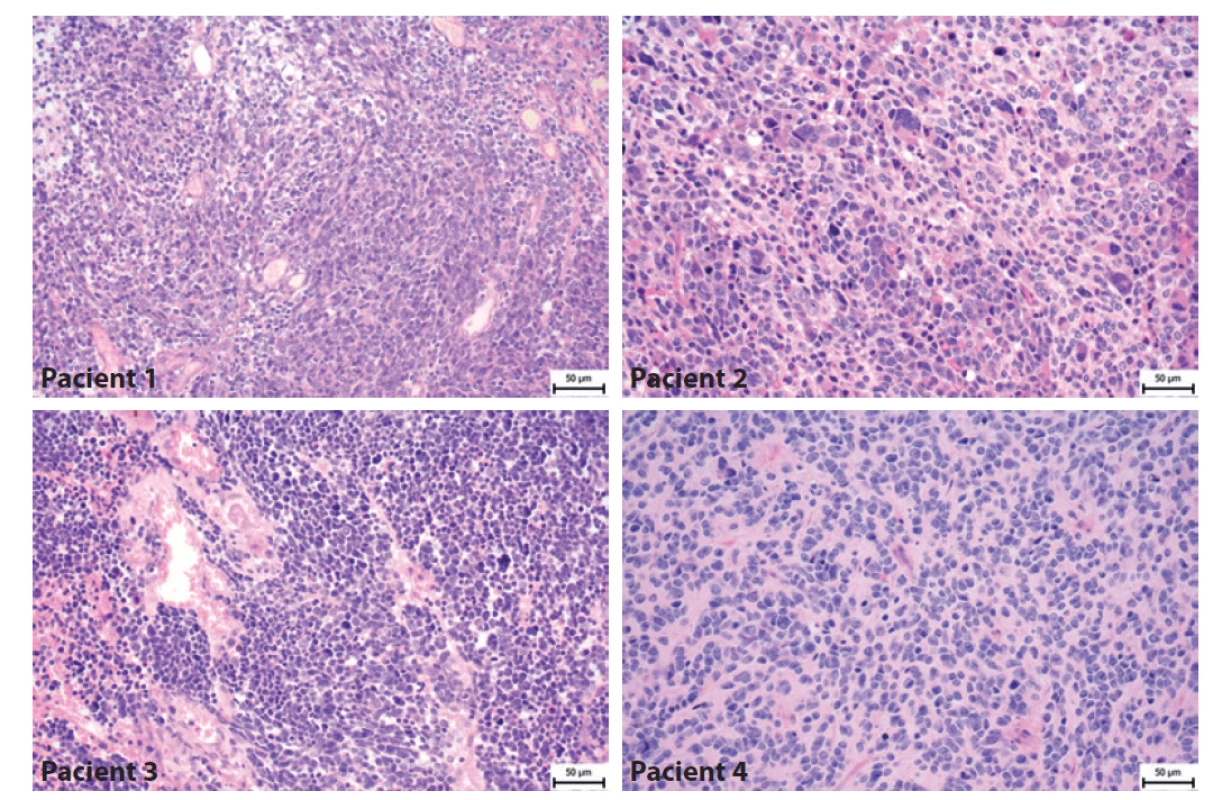
Pacient 1 - WNT-aktivovaný meduloblastom
U pacienta s několik dní trvající anamnézou zvracení a zhoršené stability chůze byl ve věku šesti let diagnostikován meduloblastom skupiny WNT. Tumor byl makroskopicky radikálně odstraněn. Pooperačně se u pacienta rozvinul těžký neurologický deficit, hodnocený jako kombinace cerebelárního akinetického mutismu a neurologicky podmíněné poruchy hybnosti levostranných končetin, až charakteru plegie. Pro těžký neurologický stav bylo zahájení radioterapie odloženo a pacient zahájil systémovou léčbu dvěma cykly chemoterapie. Po stabilizaci neurologického stavu absolvoval protonovou radioterapii v dávce 23,4Gy na oblast kraniospinální osy s boostem 30,6 Gy na lůžko tumoru. Nyní probíhá udržovací chemoterapie podle protokolu PNET5 MB. Neurologický stav pacienta se v průběhu léčby s přispěním intenzivní rehabilitace výrazně zlepšil. Prognóza pacienta je velmi příznivá s pravděpodobností celkového 5 letého přežití přes 90 % (60).
Pacient 2-SHH-aktivovaný meduloblastom
Čtrnáctiletý pacient s významnou rodinou onkologickou anamnézou a dva měsíce trvajícími bolestmi hlavy byl přijat k léčbě s diagnózou meduloblastomu v levé mozečkové hemisféře. Tumor byl makroskopicky radikálně odstraněn a molekulárně - patologické vyšetření stanovilo meduloblastom skupiny SHH s mutací genu TP53. Genetickým vyšetřením byl u pacienta potvrzen Li-Fraumeni syndrom, proto byla jako léčba 1. linie zvolen přístup založený na podávání chemoterapie, včetně intraventrikulární aplikace metotrexátu, s cílem vyhnout se radioterapii. Dva měsíce po ukončené chemoterapii při udržovací léčbě vinblastinem se objevila lokální recidiva tumoru, která byla makroskopicky radikálně odstraněna. Následně byla zahájena protonová radioterapie na kraniospinální osu (36Gy) s boostem na lůžko tumoru a levou mozečkovou hemisféru (30Gy). Poté pacient zahájil udržovací chemoterapii dle standardního ramene protokolu PNET 5 MB, která aktuálně probíhá. Pacient je na podkladě nádorové predispozice a prodělané radioterapie v riziku rozvoje sekundární malignity.
Pacient 3 –meduloblastom skupina 3
Pacientovi byl ve třech letech diagnostikován meduloblastom vyplňující IV. mozkovou komoru s mikroskopickou generalizací do likvoru. V předchorobí je u pacienta popisováno asi měsíc trvající opakované zvracení, bolesti hlavy, postupně se přidává únava. Iniciálně byla provedena kompletní resekce tumoru zadní jámy lební, histologicky byl tumor klasifikován jako klasický meduloblastom (s ložisky anaplastické/velkobuněčné varianty) dle WHO gr.IV, dle metylační array byl zařazen do skupiny 3 s prokázanou amplifikací MYC onkogenu. Pooperačně se u chlapce rozvinul neurologický deficit v podobě cerebelárního akinetického mutismus a centrální pravostranná hemiparéza. Vzhledem k věku dítěte a známé generalizaci onemocnění, byla nejprve zahájena intenzivní chemoterapie (dva cykly carboplatiny a vepesidu, dále chemoterapie dle protokolu ACNS0334: Indukční rameno B s metotrexátem) vč. intratékálního podávání Topotecanu, s cílem kontroly základního onemocnění a snahou o odklad radioterapie centrální nervové soustavy (CNS) do pozdějšího věku. Tímto přístupem se nepodařilo dosáhnout sanace likvoru, a proto byla do terapie časně zařazena toho času jediná možná léčba s kurativním potenciálem - radioterapie CNS v rozsahu kraniospinální osy 36Gy s boostem na lůžko tumoru v celkové dávce 54Gy, kterou pacient absolvoval v protonovém centru. Časně po ukončení radioterapie se objevilo difúzní metastatické postižení mening, bez lokální recidivy. V necelých 4 letech pacient zemřel, 8 měsíců od stanovení diagnózy, což podtrhuje agresivní průběh biologicky nepříznivého meduloblastomu skupiny 3 s amplifikací onkogenu MYC.
Pacient 4 –meduloblastom skupina 4
Pacientovi s téměř půl roční anamnézou zvracení, bolestí hlavy a patologickým stereotypem chůze byl ve věku šesti let diagnostikován objemný meduloblastom obklopujícím a prakticky uzavírající IV. mozkovou komoru. Makroskopicky radikálně odstraněný tumoru zadní jámy lební odpovídá meduloblastomu, dle metylační array zařazen do skupiny 4. Po operaci se u pacienta rozvinul těžký syndromu zadní jámy s celeberáním mutismem, psychomotorickým neklidem, nystagmem, hypotonií a ataxií. Pro závažný neurologický stav byl před zahájením radioterapie vložen 1 cyklus chemoterapie. Po stabilizaci neurologického stavu pacient zahájil protonovou radioterapii na kraniospinální osu 23,4Gy a lůžko tumoru. Nyní radioterapie u pacienta probíhá a dále je plánovaná udržovací terapie. Prognóza celkového přežití se pro skupinu 4 pohybuje kolem 75%. Tento pacient demonstruje nedávné zjištění, které naznačuje největší riziko rozvoje syndromu zadní jámy lební právě u dětí s meduloblastomem skupiny 4 (61).
DISKUZE
Využití metylačního profilu je další z molekulárně biologických technik, které se staly součástí diagnostického spektra vyšetření u nádorů CNS. Metylační profil jednotlivých typů nádorů by mohl být zařazen do doporučeného diagnostického schématu i v rámci WHO klasifikace, a to zejména u skupin a podskupin nádorů CNS, kde nebude možné jinými technikami diagnózu stanovit. Do takovéto skupiny patří embryonální nádory, včetně meduloblastomu a ependymomy zadní jámy, které nemají specifické genetické alterace, které by umožnily jejich jednoznačné zařazení do skupiny nebo podskupiny daného tumoru. Metylační SNP lze také využít jako doplňkovou techniku v rámci vyšetření tam, kde známe specifickou genetickou alteraci, ale rádi bychom znali celý genetický profil chromozomálních změn k prokázání prognostických faktorů a potvrdili zařazení na základě metylačního profilu. Příkladem může být supratentoriální ependymom, který je spojen s ZFTA-RELA a YAP-MAMLD1 fúzí, často na podkladě chromotripsis 11p (30,44). I přesto zbývá přibližně 10 % supratentoriálních ependymomů, které je obtížné zařadit (62). Jedinečnou vlastností metylační SNP je, že na základě podobnosti metylačního profilu a chromozomálních změn můžeme nalézt nové podskupiny nádorů CNS, u kterých následně hledáme další specifické morfologické nebo genetické změny (3,29). U morfologicky podobných skupin tumorů (např. Tanycytické ependymomy), které nemají definované specifické genetické změny (opakující se chromozomální nebo mutační změna nebo fúzní gen) charakterizující danou skupinu, můžeme k potvrzení histologické diagnózy využít stanovení metylačního profilu (62). Využití metylační SNP u většiny embryonálních a high-grade tumorů je již dobře zařazeno do vyšetřovacího schématu (3,17,22,24,29,34,39,45). Jako méně významné se však jeví jeho použití u dětských low-grade gliomů. Ačkoliv některé publikace stratifikovaly dětské low-grade gliomy na základě metylačního profilu (41,63–66), při porovnáni s histologickým obrazem, lokalizací tumoru a genetickými změnami není toto zařazení jednoznačné. Ne vždy se tedy daří nádory na základě metylačního profilu klasifikovat. Zde může hrát významnou roli přítomnost nenádorových buněk, a to jak normální mozkové tkáně, tak i infiltrujících lymfocytů, které naruší metylační profil nádorových buněk. Problémem je, že se množství těchto nenádorových buněk může lišit vzorek od vzorku a není možné zajistit jejich homogenitu. Nicméně i u této skupiny nádorů Fukuoka a kol. prokázali možné využití metylační SNP, a to při stanovení diagnózy Diffuse leptomeningeal glioneuronal tumour (DLGNT) na podkladě metylačního profilu a v kombinaci s histologickým obrazem, lokalizací tumoru, genetických změn a infiltrace leukocyty, stanovené na základě metylační SNP (67), kde jsou rozděleny dětské low-grade tumory do 5 diagnosticko-prognostických skupin (36). Je tak zřejmé, že kromě diagnostického, je metylační SNP také prognostickým nástrojem, který je, nebo bude v budoucnu využíván k výběru protinádorové terapie. Ani při použití všech nyní dostupných diagnostických prostředků není možné diagnostikovat některé vzácné nádory CNS. Proto autoři Heidelberského klasifikátoru předpokládají, že se tato platforma stane celosvětovou databází takto vyšetřených vzorků a umožní klasifikaci dalších vzácných podjednotek nádorů CNS i tam, kde to v současné době není možné (3,4).
Předpokládáme, že se metylační SNP zařadí do široké palety diagnostických postupů využívaných v diagnostice a prognostice nádorů CNS, nebo k výběru cílené terapie (53), protože často jen kombinace většiny nebo dokonce všech těchto postupů, stanoví správnou diagnózu a umožní nadále zlepšovat péči o pacienty s nádory v oblasti CNS.
PODĚKOVÁNÍ
Práce byla podpořena MZ ČR – RVO, FN v Motole 00064203 a z projektu 1000 statečných
PROHLÁŠENÍ
Autor práce prohlašuje, že v souvislosti s tématem, vznikem a publikací tohoto článku není ve střetu zájmů a vznik ani publikace článku nebyly podpořeny žádnou farmaceutickou firmou. Toto prohlášení se týká i všech spoluautorů.
Adresa pro korespondenci:
MUDr. Aleš Vícha, PhD.
Klinika dětské hematologie a onkologie 2.LF UK a FN Motol
V úvalu 84, Praha 5, 15008
Tel: 224436470, Fax: 224436417
e-mail: ales.vicha@fnmotol.cz
Sources
1. Pickles JC, Fairchild AR, Stone TJ, et al. DNA methylation-based profiling for paediatric CNS tumour diagnosis and treatment: a population-based study. Lancet Child Adolesc Heal 2020; 4 : 121–30. doi:10.1016/S2352 - 4642(19)30342-6.
2. Anvar Z, Acurzio B, Roma J, Cerrato F, Verde G. Origins of DNA methylation defects in Wilms tumors. Cancer Letters 2019; 457 : 119–128.
3. Capper D, Jones DTW, Sill M, et al. DNA methylation-based classification of central nervous system tumours. Nature 2018; 555 : 469–474.
4. Koelsche C, Schrimpf D, Stichel D, et al. Sarcoma classification by DNA methylation profiling. Nat Commun 2021; 12. doi:10.1038/ s41467-020-20603-4.
5. Nobre L, Zapotocky M, Khan S, et al. Pattern of Relapse and Treatment Response in WNT-Activated Medulloblastoma. Cell Reports Med 2020; 1(3): 100038.
6. Sievers P, Sill M, Blume C, et al. Clear cell meningiomas are defined by a highly distinct DNA methylation profile and mutations in SMARCE1. Acta Neuropathol 2021; 141 : 281 – 290.
7. Sievers P, Appay R, Schrimpf D, et al. Rosette - forming glioneuronal tumors share a distinct DNA methylation profile and mutations in FGFR1, with recurrent co-mutation of PIK3CA and NF1. Acta Neuropathol 2019; 138 : 497–504.
8. Sumerauer D, Krskova L, Vicha A, et al. Rare IDH1 variants are common in pediatric hemispheric diffuse astrocytomas and frequently associated with Li-Fraumeni syndrome. Acta Neuropathol 2020; 139 : 795–7.
9. Van Paemel R, De Koker A, Vandeputte C, et al. Minimally invasive classification of paediatric solid tumours using reduced representation bisulphite sequencing of cell-free DNA: a proof-of-principle study. Epigenetics 2020; 16 : 1–13.
10. Witt H, Gramatzki D, Hentschel B, et al. DNA methylation-based classification of ependymomas in adulthood: Implications for diagnosis and treatment. Neuro Oncol 2018; 20 : 1616–1624.
11. Mancarella D, Plass C. Epigenetic signatures in cancer: proper controls, current challenges and the potential for clinical translation. Genome Medicine 2021; 3(1): 23.
12. Machnik M, Oleksiewicz U. Dynamic Signatures of the Epigenome: Friend or Foe? Cells 2020; 9(3): 653.
13. Corley J, Cox SR, Harris SE, et al. Epigenetic signatures of smoking associate with cognitive function, brain structure, and mental and physical health outcomes in the Lothian Birth Cohort 1936. Transl Psychiatry 2019; 9(1): 248.
14. Hillary RF, Stevenson AJ, Cox SR, et al. An epigenetic predictor of death captures multi-modal measures of brain health. Mol Psychiatry 2019, In Print.
15. Fernandez AF, Assenov Y, Martin-Subero JI, et al. A DNA methylation fingerprint of 1628 human samples. Genome Res 2012; 22 : 407–419.
16. Ehrlich M. DNA hypomethylation in cancer cells. Epigenomics 2009; 1 : 239–259.
17. Hovestadt V, Remke M, Kool M, et al. Robust molecular subgrouping and copy-number profiling of medulloblastoma from small amounts of archival tumour material using high-density DNA methylation arrays. Acta Neuropathologica 2013; 125 : 913–916.
18. Korshunov A, Chavez L, Northcott PA, et al. DNA-methylation profiling discloses significant advantages over NanoString method for molecular classification of medulloblastoma. Acta Neuropathologica 2017; 134 : 965–967.
19. Sahm F, Schrimpf D, Stichel D, et al. DNA methylation-based classification and grading system for meningioma: a multicentre, retrospective analysis. Lancet Oncol 2017; 18 : 682–694.
20. Sexton-Oates A, Dodgshun A, Hovestadt V, et al. Methylation profiling of paediatric pilocytic astrocytoma reveals variants specifically associated with tumour location and predictive of recurrence. Mol Oncol 2018; 12 : 1219–1232.
21. Hovestadt V, Jones DTW, Picelli S, et al. Decoding the regulatory landscape of medulloblastoma using DNA methylation sequencing. Nature 2014; 510 : 537–541.
22. Koelsche C, Hartmann W, Schrimpf D, et al. Array-based DNA-methylation profiling in sarcomas with small blue round cell histology provides valuable diagnostic information. Mod Pathol 2018; 31 : 1246–1256.
23. Sievers P, Sill M, Schrimpf D, et al. A subset of pediatric-type thalamic gliomas share a distinct DNA methylation profile, H3K27me3 loss and frequent alteration of EGFR. Neuro Oncol 2021; 23 : 34–43.
24. Binder H, Willscher E, Loeffler-Wirth H, et al. DNA methylation, transcriptome and genetic copy number signatures of diffuse cerebral WHO grade II/III gliomas resolve cancer heterogeneity and development. Acta Neuropathol Commun 2019; 7 : 59.
25. Braczynski AK, Capper D, Jones DTW, et al. High density DNA methylation array is a reliable alternative for PCR-based analysis of the MGMT promoter methylation status in glioblastoma. Pathol Res Pract 2020; 216(1): 152728.
26. Koelsche C, Kriegsmann M, Kommoss FKF, et al. DNA methylation profiling distinguishes Ewing-like sarcoma with EWSR1–NFATc2 fusion from Ewing sarcoma. J Cancer Res Clin Oncol 2019; 145(5): 1273-1281.
27. Capper D, Engel NW, Stichel D, et al. DNA methylation-based reclassification of olfactory neuroblastoma. Acta Neuropathol 2018; 136 : 255–271.
28. Capper D, Engel NW, Stichel D, et al. Correction to: DNA methylation-based reclassification of olfactory neuroblastoma (Acta Neuropathologica, (2018), 136, 2, (255-271), 10.1007/ s00401-018-1854-7). Acta Neuropathologica 2018; 136 : 505.
29. Capper D, Stichel D, Sahm F, et al. Practical implementation of DNA methylation and copy-number-based CNS tumor diagnostics: the Heidelberg experience. Acta Neuropathol 2018; 136 : 181–210. 3
0. Pagès M, Pajtler KW, Puget S, et al. Diagnostics of pediatric supratentorial RELA ependymomas: integration of information from histopathology, genetics, DNA methylation and imaging. Brain Pathol 2019; 29 : 325–335.
31. Moran S, Martínez-Cardús A, Sayols S, et al. Epigenetic profiling to classify cancer of unknown primary: a multicentre, retrospective analysis. Lancet Oncol 2016; 17 : 1386–1395.
32. Pickles JC, Fairchild AR, Stone TJ, et al. DNA methylation-based profiling for paediatric CNS tumour diagnosis and treatment: a population - based study. Lancet Child Adolesc Heal 2020; 4 : 121–130.
33. Stichel D, Schrimpf D, Sievers P, et al. Accurate calling of KIAA1549-BRAF fusions from DNA of human brain tumours using methylation array-based copy number and gene panel sequencing data. Neuropathol Appl Neurobiol 2021; 47 : 406–414.
34. Korshunov A, Sahm F, Zheludkova O, et al. DNA methylation profiling is a method of choice for molecular verification of pediatric WNT-activated medulloblastomas. Neuro Oncol 2019; 21 : 214–221.
35. Ellison DW, Aldape KD, Capper D, et al. cIMPACT - NOW update 7: advancing the molecular classification of ependymal tumors. Brain Pathology 2020; 30 : 863–866.
36. Fukuoka K, Mamatjan Y, Tatevossian R, et al. Clinical impact of combined epigenetic and molecular analysis of pediatric low-grade gliomas. Neuro Oncol 2020; 22 : 1474–1483.
37. Halliday GC, Junckerstorff RC, Bentel JM, et al. The case for DNA methylation based molecular profiling to improve diagnostic accuracy for central nervous system embryonal tumors (not otherwise specified) in adults. J Clin Neurosci 2018; 47 : 163–167.
38. Hou Y, Pinheiro J, Sahm F, et al. Papillary glioneuronal tumor (PGNT) exhibits a characteristic methylation profile and fusions involving PRKCA. Acta Neuropathol 2019; 137 : 837–846.
39. Jaunmuktane Z, Capper D, Jones DTW, et al. Methylation array profiling of adult brain tumours: diagnostic outcomes in a large, single centre. Acta Neuropathol Commun 2019; 7 : 24.
40. Koch A, Joosten SC, Feng Z, et al. Analysis of DNA methylation in cancer: Location revisited. Nature Reviews Clinical Oncology 2018; 15 : 459–466.
41. Lambert SR, Witt H, Hovestadt V, et al. Differential expression and methylation of brain developmental genes define location-specific subsets of pilocytic astrocytoma. Acta Neuropathol 2013; 126 : 291–301.
42. Petruzzellis G, Alessi I, Colafati GS, et al. Role of DNA methylation profile in diagnosing astroblastoma: A case report and literature review. Front Genet 2019; 10 : 391.
43. Sievers P, Stichel D, Hielscher T, et al. Chordoid meningiomas can be sub-stratified into prognostically distinct DNA methylation classes and are enriched for heterozygous deletions of chromosomal arm 2p. Acta Neuropathologica 2018; 136 : 975–978.
44. Pajtler KW, Witt H, Sill M, et al. Molecular Classification of Ependymal Tumors across All CNS Compartments, Histopathological Grades, and Age Groups. Cancer Cell 2015; 27 : 728–743.
45. Sharma T, Schwalbe EC, Williamson D, et al. Second-generation molecular subgrouping of medulloblastoma: an international meta - analysis of Group 3 and Group 4 subtypes. Acta Neuropathol 2019; 138 : 309–326.
46. Wesseling P, Capper D. WHO 2016 Classification of gliomas. Neuropathology and Applied Neurobiology 2018; 44 : 139–150.
47. Orr BA. Pathology, diagnostics, and classification of medulloblastoma. Brain Pathology 2020; 30 : 664–678.
48. Ramaswamy V, Remke M, Bouffet E, et al. Risk stratification of childhood medulloblastoma in the molecular era: the current consensus. Acta Neuropathol 2016; 131 : 821–831.
49. Thompson MC, Fuller C, Hogg TL, et al. Genomics identifies medulloblastoma subgroups that are enriched for specific genetic alterations. J Clin Oncol 2006; 24 : 1924–1931.
50. Kool M, Koster J, Bunt J, et al. Integrated genomics identifies five medulloblastoma subtypes with distinct genetic profiles, pathway signatures and clinicopathological features. PLoS One 2008; 3(8): e3088.
51. Kool M, Korshunov A, Remke M, et al. Molecular subgroups of medulloblastoma: An international meta-analysis of transcriptome, genetic aberrations, and clinical data of WNT, SHH, Group 3, and Group 4 medulloblastomas. Acta Neuropathol 2012; 123 : 473–484.
52. Cho YJ, Tsherniak A, Tamayo P, et al. Integrative genomic analysis of medulloblastoma identifies a molecular subgroup that drives poor clinical outcome. J Clin Oncol 2011; 29 : 1424–1430.
53. Zou H, Poore B, Broniscer A, Pollack IF, Hu B. Molecular Heterogeneity and Cellular Diversity: Implications for Precision Treatment in Medulloblastoma. Cancers (Basel) 2020; 12(3): 643.
54. Taylor MD, Northcott PA, Korshunov A, et al. Molecular subgroups of medulloblastoma: The current consensus. Acta Neuropathol 2012; 123 : 465–472.
55. Hovestadt V, Ayrault O, Swartling FJ, Robinson GW, Pfister SM, Northcott PA. Medulloblastomics revisited: biological and clinical insights from thousands of patients. Nature Reviews Cancer 2020; 20 : 42–56.
56. Northcott PA, Jones DTW, Kool M, et al. Medulloblastomics: The end of the beginning. Nature Reviews Cancer 2012; 12 : 818–834.
57. Schwalbe EC, Hicks D, Rafiee G, et al. Minimal methylation classifier (MIMIC): A novel method for derivation and rapid diagnostic detection of disease-associated DNA methylation signatures. Sci Rep. 2017; 7(1): 13421.
58. Schwalbe EC, Williamson D, Lindsey JC, et al. DNA methylation profiling of medulloblastoma allows robust subclassification and improved outcome prediction using formalin - fixed biopsies. Acta Neuropathol 2013; 125 : 359–371.
59. Cavalli FMG, Remke M, Rampasek L, et al. Intertumoral Heterogeneity within Medulloblastoma Subgroups. Cancer Cell 2017; 31 : 737-754.e6.
60. Nobre L, Zapotocky M, Khan S, et al. Pattern of Relapse and Treatment Response in WNT-Activated Medulloblastoma. Cell Reports Med 2020; 1(3):100038.
61. Jabarkheel R, Amayiri N, Yecies D, et al. Molecular correlates of cerebellar mutism syndrome in medulloblastoma. Neuro Oncol 2020; 22 : 290–297.
62. Zschernack V, Jünger ST, Mynarek M, et al. Supratentorial ependymoma in childhood: more than just RELA or YAP. Acta Neuropathol 2021; 141 : 455–466.
63. Fukuoka K, Mamatjan Y, Tatevossian R, et al. Clinical impact of combined epigenetic and molecular analysis of pediatric low-grade gliomas. Neuro Oncol 2020; 22 : 1474–1483.
64. Jeyapalan JN, Doctor GT, Jones TA, et al. DNA methylation analysis of paediatric lowgrade astrocytomas identifies a tumour-specific hypomethylation signature in pilocytic astrocytomas. Acta Neuropathol Commun 2016; 4 : 54.
65. Qaddoumi I, Orisme W, Wen J, et al. Genetic alterations in uncommon low-grade neuroepithelial tumors: BRAF, FGFR1, and MYB mutations occur at high frequency and align with morphology. Acta Neuropathol 2016; 131 : 833–845.
66. Stone TJ, Keeley A, Virasami A, et al. Comprehensive molecular characterisation of epilepsy - associated glioneuronal tumours. Acta Neuropathol 2018; 135 : 115–129.
67. Aran D, Sirota M, Butte AJ. Systematic pan-cancer analysis of tumour purity. Nat Commun 2015; 6 : 8971.
Labels
Anatomical pathology Forensic medical examiner Toxicology
Article was published inCzecho-Slovak Pathology

2021 Issue 3-
All articles in this issue
- Next-Generation Pathology
- Cesta k cíli vede přes spolupráci
- 'PATOLOGIE MĚKKÝCH TKÁNÍ
- 'PATOLOGIE ORL OBLASTI
- 'UROPATOLOGIE
- 'GYNEKOPATOLOGIE
- 'NEFROPATOLOGIE
- 'HEPATOPATOLOGIE
- 'KARDIOPATOLOGIE
- 'HEMATOPATOLOGIE
- 'PATOLOGIE MĚKKÝCH TKÁNÍ
- 'PATOLOGIE GIT
- 'CYTODIAGNOSTIKA
- 'LABORATORNÍ METODY
- Sekvenování nové generace – nástroj vědy či rutinní patologie?
- 'PULMOPATOLOGIE
- Sekvenování nové generace a molekulární onkologický multidisciplinární tým z pohledu onkologa
- Možnosti molekulárního testování somatických aberací v nádorové tkáni metodou NGS v rutinní praxi - aktuální situace v České republice
- Sekvenování nové generace a jeho aplikace v diagnostice neuromuskulárních onemocnění
- Využití metylačního profilu v diagnostice a prognostice nádorových onemocnění CNS
- Přehled problematiky hodnocení tumor infiltrujících lymfocytů u karcinomu prsu v rutinní diagnostické praxi
- Nediagnostikovaná kryptokoková meningoencefalitída s rozsiahlymi hypoxickoischemickými zmenami mozgu charakteru tretieho stupňa difúzneho axonálneho poškodenia
- Doporučení pro testování PD-L1: metodika testování a reportování výsledků Verze_1 (17. 5. 2021)
- Molekulární testování u karcinomu endometria (společné doporučení ČOS, onkogynekologické sekce ČGPS, SROBF a SČP ČLS JEP)
- Obrovskobuněčná myokarditida u mladé ženy odhalená při pitvě: kazuistika
- Czecho-Slovak Pathology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Doporučení pro testování PD-L1: metodika testování a reportování výsledků Verze_1 (17. 5. 2021)
- Sekvenování nové generace – nástroj vědy či rutinní patologie?
- Molekulární testování u karcinomu endometria (společné doporučení ČOS, onkogynekologické sekce ČGPS, SROBF a SČP ČLS JEP)
- Využití metylačního profilu v diagnostice a prognostice nádorových onemocnění CNS
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

