-
Medical journals
- Career
Sekvenování nové generace
: Eva Froňková
: Laboratoř molekulární genetiky CLIP, Klinika dětské hematologie a onkologie 2. LF UK a FN Motol, Praha
: Čes.-slov. Patol., 49, 2013, No. 3, p. 129-132
: Reviews Article
Sekvenování nové generace, nazývané také masivně paralelní sekvenování (MPS), je v současnosti nejrychleji se rozvíjející metodou molekulární genetiky, která přinese zlom v oblasti personalizované medicíny. V tomto přehledu stručně popisujeme hlavní typy MPS, kterými jsou celogenomová a exomová sekvenace, sekvenace transkriptomu a amplikonové sekvenování. Dále je uveden souhrn výhod, nevýhod a možných aplikací technologií nabízených v současnosti v České republice.
Klíčová slova:
masivně paralelní sekvenování – sekvenování nové generace – exom –transkriptom – amplikonové sekvenováníSekvenování nové generace, nazývané také masivně paralelní sekvenování (MPS), je nejmodernější a nejrychleji se rozvíjející metodou molekulární genetiky, která způsobí zásadní zlom ve výzkumu patologických stavů a především v oblasti tzv. personalizované medicíny. Pomocí stále výkonnějších přístrojů je MPS schopno v jediném vyšetření generovat až stovky milionů různých nukleotidových sekvencí (v angličtině tzv. „reads“). Náklady na MPS velmi rychle klesají; těžko by někdo ještě před několika lety čekal, že v roce 2012 bude možno přečíst celý genom jedince během několika dnů za cenu menší než 10 000 USD. Aplikace metod MPS bude znamenat vedle nových diagnostických možností zároveň i jistou změnu v nárocích na interpretaci výsledků vyšetření. V laboratorní práci bude třeba stále více využívat dovednosti z oblasti bioinformatiky s nutností alespoň základní orientace v principech generování a analýzy dat, softwarových aplikacích a systému internetových databází. V následujícím přehledu chceme shrnout současné možnosti MPS s vědomím, že nabídka a dostupnost metod na trhu se neustále mění a informace rychle ztrácejí svoji aktuálnost. Rychlý vývoj zároveň znamená neustálenost českých pojmenování, proto vždy uvádíme příslušné anglické termíny.
APLIKACE
Vysoký výkon současných sekvenátorů lze použít v rozsáhlém spektru aplikací od pokrytí rozsáhlé oblasti, např. celého genomu jedince („ultraširoká sekvenace”), po sekvenaci pouze jediné oblasti s vysokým počtem přečtení v případě tzv. amplikonového sekvenování („ultrahluboká sekvenace”) (tab. 1).
1. Přehled různých aplikací masivně paralelního sekvenování. 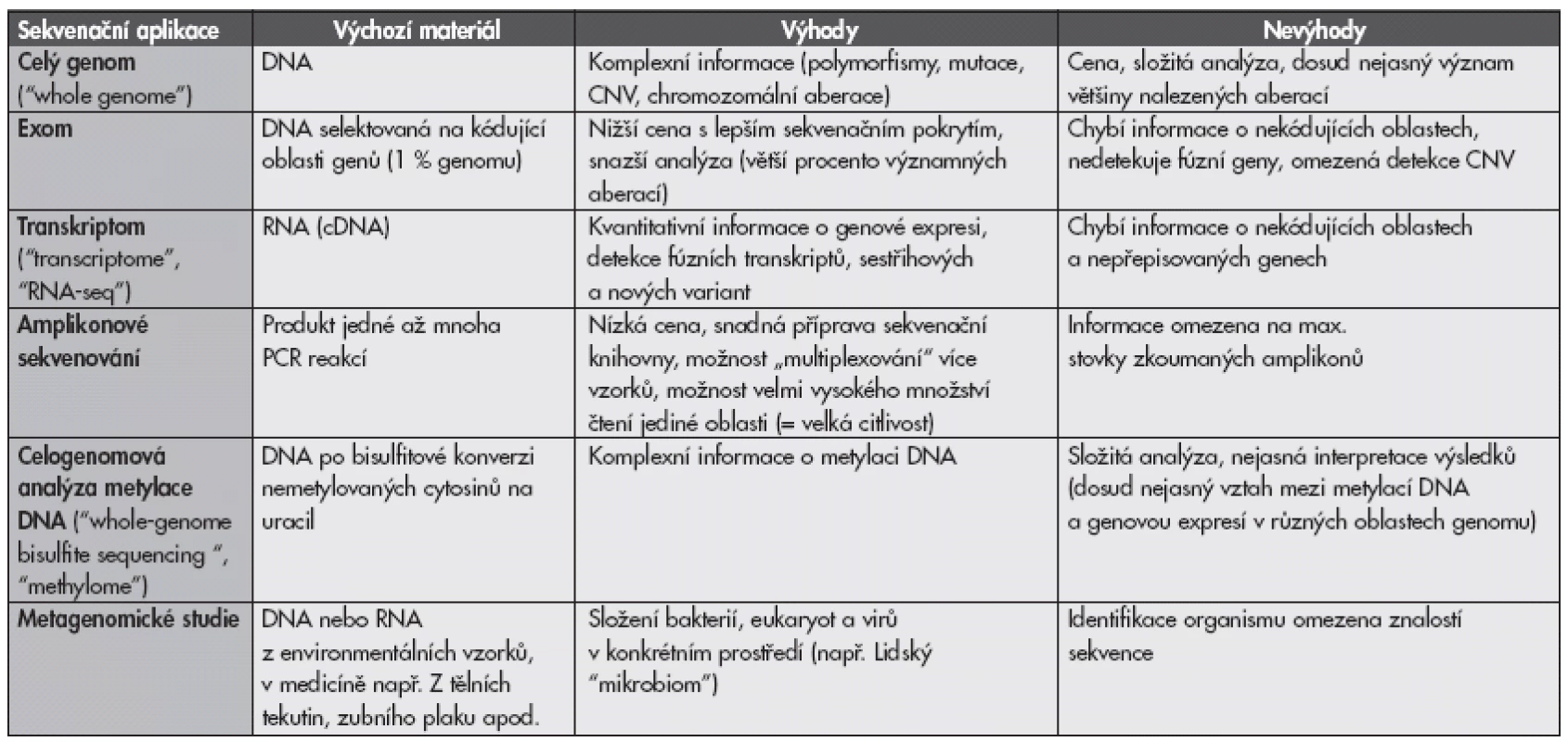
CNV, copy number variation Celogenomová sekvenace („whole-genome sequencing“, obr. 1A) poskytuje nejkomplexnější obraz o genomu – kromě genů i o variacích v nekódujících oblastech, potenciálních chromozomálních translokacích, delecích a amplifikacích (tzv. „copy number variants“, CNV). Cílem je pokrýt každý nukleotid v genomu obvykle 30 – 40x, část přečtených sekvencí je ale ztracena kvůli chybám sekvenace nebo špatnému mapování k referenčnímu genomu. Nevýhodou zůstává cena a obrovská komplexita získaných dat. U solidních nádorů je sekvenování problematičtější než např. u vzorků krve z důvodu nerovnoměrné infiltrace vzorku nádorovými buňkami a časté diverzity s přítomností více klonů s různými genomy. Toto lze částečně překonat zvýšením počtu čtení a úpravou algoritmů pro analýzu dat (1). Přítomnost nekrotických buněk nebo fixace materiálu rovněž způsobuje horší kvalitu izolované DNA. Proto je výhodné počítat s případnou genetickou analýzou a zajistit izolaci materiálu z čerstvého vzorku. V jednom lidském genomu jsou odhadem 3 – 4 miliony zděděných variací a stovky CNV (2). To znamená, že u nádorového genomu bude většina objevených variací zděděnými polymorfismy a ne získanými mutacemi. Kvůli tomu je důležité u vzorků nádorů zároveň sekvenovat normální tkáň pacienta (např. remisní vzorek krve u leukémií). Po „odfiltrování“ zděděných variant zbývají řádově tisíce alterací, jejichž množství se velmi liší mezi jednotlivými typy nádorů. Pro určení, které mutace jsou klinicky důležité a které jsou pouze „vedlejším produktem“ nádorové transformace (tzv. „driver“ a „passenger mutations“), lze využít porovnání většího souboru vzorků od různých pacientů nebo databáze, které predikují efekt záměny aminokyseliny na strukturu a funkci proteinu (např. SIFT, PolyPhen).
1. Zjednodušené schéma tvorby sekvenační knihovny u různých typů masivně paralelního sekvenování. A. celogenomové sekvenace: po naštěpení jsou sekvenovány všechny fragmenty DNA o vhodné délce; B. exomová sekvenace: jsou selektovány pouze fragmenty obsahující sekvence kódujících oblastí; C. transkriptomová sekvenace: sekvenována RNA po přepisu do cDNA; D. amplikonová sekvenace: sekvenovány s velkou hloubkou čtení produkty jedné nebo více PCR reakcí cílených na oblast zájmu (např. mutace u nádoru). Použité primery mohou již obsahovat sekvence adaptorů specifických pro daný přístroj (šedá barva). 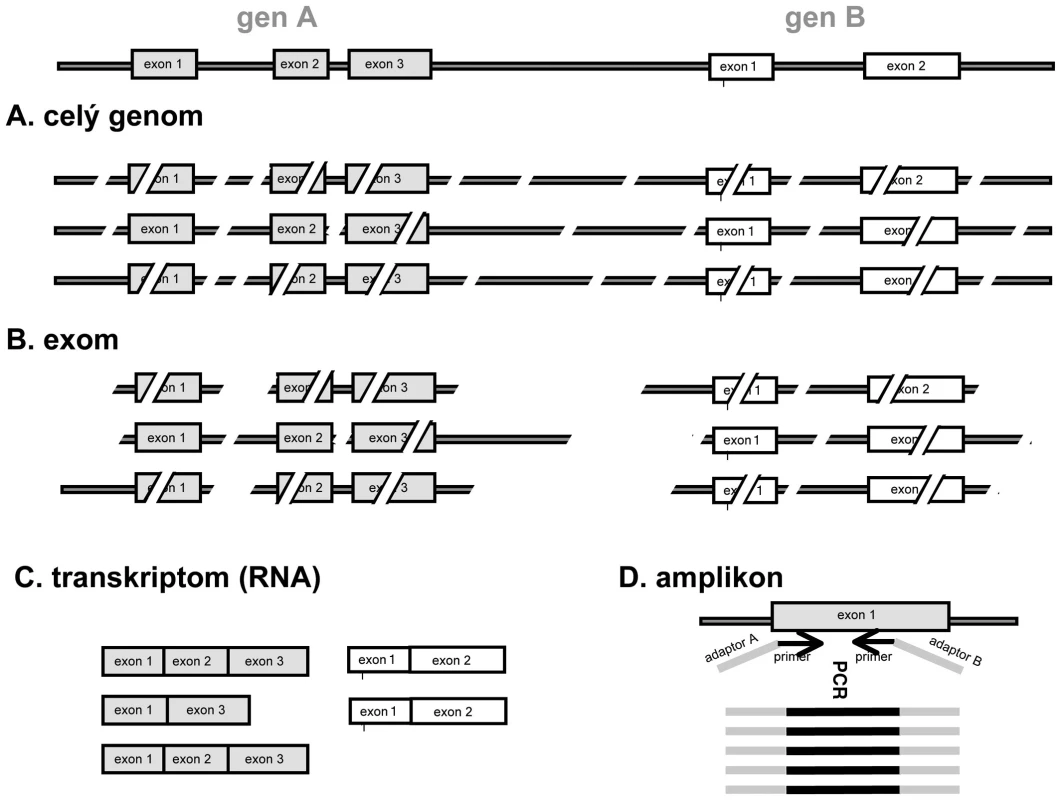
Zjednodušenou variantou je tzv. „exomové sekvenování“, obr. 1B. Jako vstupní materiál slouží také DNA. Díky různým způsobům obohacení požadovaných úseků mohou být sekvenovány pouze exony genů kódující proteiny, které tvoří cca 1 % genomu (3). Tím je analýza jednodušší a metoda cenově přijatelnější při vyšším množství přečtení jednotlivých úseků. Zároveň jsou většinou sekvenovány přechodové sekvence exon-intron k určení mutací ovlivňujících genový splicing. Nevýhodou je nemožnost detekovat strukturální aberace (mutace, chromozomální translokace) vyskytující se mimo kódující oblasti a CNV většího rozsahu, i když byl popsán bioinformatický algoritmus k vytvoření tzv. „pseudo-CNV“ profilu u nádorů na základě rozdílu průměrného sekvenačního pokrytí nádorového a normálního vzorku (4).
Transkriptomové sekvenování („transcriptome sequencing“, „RNA-seq“, obr. 1C) pokrývá kódující i nekódující RNA. Jednou z jeho výhod je kvantitativní charakter získané informace, neboť množství transkriptu se přibližně odráží v zastoupení přečtených úseků. Předpokládá se, že metoda časem vytlačí expresní profilování, oproti kterému není nutná předchozí znalost vyšetřovaných genů. K tomu však bude třeba dostatečné množství „čtení“ (obvykle v řádu desítek milionů), protože některé abundantně přepisované geny mohou způsobit malé sekvenační pokrytí ostatních (5). Metoda má spolu s exomovým sekvenováním nevýhodu v absenci informace o nekódujících oblastech, dovede ale navíc detekovat transkripty fúzních genů a jednotlivé sestřihové varianty. Oproti exomu neumí sekvenovat varianty, které nejsou exprimovány.
Pro provedení sekvenační reakce je nejprve nutno vytvořit tzv. sekvenační knihovnu („sequencing library“). DNA je štěpena na náhodné fragmenty o délce řádově stovek bazí. Pro fragmentaci lze použít fyzikální metody (např. sonikace, nebulizace) nebo enzymatické štěpení. K fragmentům o vhodné délce je třeba připojit z obou stran tzv. sekvenační adaptory, což jsou oligonukleotidové sekvence specifické pro konkrétní přístroj, umožňující „ukotvení“ jediné molekuly např. na mikrokuličce nebo na sklíčku a následnou klonální amplifikaci. Tzv. amplikonové sekvenování využívá pro přípravu sekvenační knihovny produkty klasické PCR pro jeden nebo více genů. Výhodné je použití tzv. fúzních primerů, kdy primery používané pro amplifikaci zároveň obsahují sekvence adaptorů. Výsledný PCR produkt pak již funguje přímo jako sekvenační knihovna (obr. 1D), takže odpadá krok zahrnující ligaci adaptorů. Výhodou amplikonového sekvenování je kromě jednoduché a levné přípravy možnost detekce mutací s velkou hloubkou. Toho lze využít např. při stanovení klonální diverzity nádorů nebo při sledování minimální zbytkové choroby (6). Firmy vyvíjejí a nabízejí systémy na sekvenování desítek až stovek vybraných genů v jediném sekvenačním běhu na bázi amplikonového sekvenování (např. Ion AmpliSeqTM Cancer Panel od Life Sciences nebo TruSeq Amplicon-Cancer Panel od firmy Illumina). Lze předpokládat, že v rámci diagnostiky nádorů budou v příštích letech podobným způsobem vyšetřovány panely genů obsahující prognosticky relevantní onkogeny, ale i např. polymorfismy ve farmakodynamicky důležitých genech. Pro maximální využití kapacity přístrojů lze sekvenovat více vzorků zároveň pomocí vložení krátkých nukleotidových sekvencí (tzv. „barcodes“ nebo „MIDs“) mezi sekvenované fragmenty a sekvenační adaptory. Při bioinformatické analýze je pak na základě těchto sekvencí tříděna informace pro jednotlivé pacienty/vzorky, což vede k výrazné finanční úspoře.
Dalšími aplikacemi MPS jsou epigenomické studie jako je celogenomová analýza metylomu (metylace DNA)(7) nebo lokalizace modifikovaných histonů. Výzkum mikroorganismů zaznamenal díky MPS prudký rozvoj v oblasti tzv. metagenomických studií (tj. studií zkoumající druhové složení v konkrétním prostředí, v medicíně např. složení střevní mikroflóry). V konkrétní situaci je s výhodou použít kombinaci přístupů (např. celogenomová a transkriptomová sekvenace plus analýza metylomu u nádorů).
NEJČASTĚJI POUŽÍVANÉ PŘÍSTROJE A PRINCIPY METODY
Nabídka technologií se rychle vyvíjí a informace o konkrétním přístroji bývá často zkreslena konkurenčním bojem firem ve snaze o ovládnutí trhu. Zkušenosti uživatelů i publikované studie se shodují, že nejdůležitější je vybírat přístroj již s ohledem na plánované aplikace, neboť každý má slabé a silné stránky vzhledem k různým možnostem použití (8–11). Obecně existují přístroje s vysokým výkonem a dražším provozem pro velké projekty a zároveň tzv. „stolní“ varianty přístrojů určené pro běžný laboratorní provoz (tab. 2). V tomto přehledu uvádíme pouze přístroje v současnosti dostupné v České republice. Před pořízením přístroje je nutné pečlivě zvážit množství a druh plánovaných sekvenací, financování obsluhujícího personálu, údržby a chodu přístroje. V současnosti se stále rozrůstá nabídka firemní sekvenace formou služeb, kdy laboratoř zasílá pouze genetický materiál (DNA, PCR produkt apod.) a firmou jsou zpětně dodána sekvenační data většinou již s primární bioinformatickou analýzou. Především u projektů s menším počtem zkoumaných vzorků na sekvenátorech s vysokou kapacitou (genom, exom) je často tato možnost finančně výhodnější než pořízení, údržba a provoz vlastního stroje.
2. Porovnání sekvenačních technologií dostupných v ČR v roce 2012. Údaje byly převzaty z dostupných firemních specifikací a/nebo z referencí (8,9). 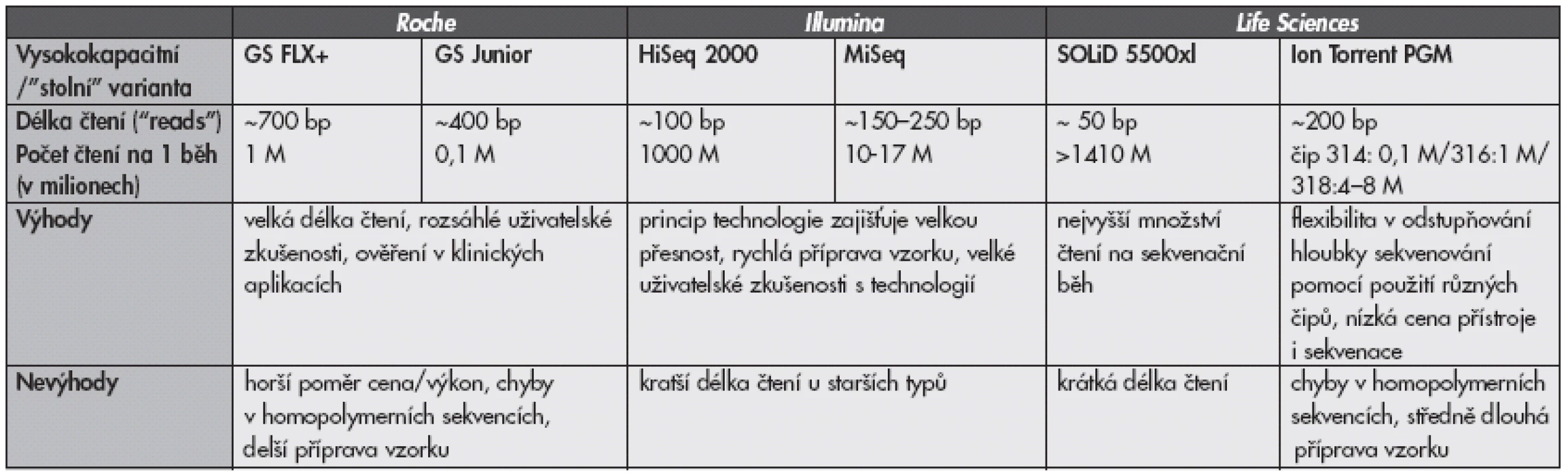
bp, base-pairs Nejdéle fungujícím systémem na trhu, dostupným od roku 2005, je technologie 454 firmy Roche (v současnosti přístroj GS FLX+ a jeho stolní varianta GS Junior). Sekvenační reakce v přístroji je zde založena na pyrosekvenaci s předchozí amplifikací jednotlivých fragmentů na kuličkách v olejové emulzi (tzv. „emulsion PCR“). Existuje celá řada studií popisující využití těchto přístrojů v medicíně jako je např. ultrahluboká detekce mutací, minimální reziduální nemoci, detekce patogenů apod. (6). Výhodou tohoto systému je vedle širokého ověření uživateli dosud nepřekonaná délka přečtených sekvencí (běžně 400 bp, nově až 1000 bp), nevýhodou je poměrně zdlouhavá příprava vzorků a ve srovnání s nejmodernějšími přístroji nižší počet čtení na jeden experiment spolu s horším poměrem cena/výkon.
V posledních letech zaujímá především v oblasti sekvenátorů s vysokým výkonem hlavní postavení na trhu firma Illumina s principem Solexa (starší verze přístrojů Genome Analyzer a novější HiSeq), v posledním roce se začíná masivně rozšiřovat „stolní“ varianta, přístroj MiSeq. Sekvenační reakce je založena na klasické sekvenaci syntézou s kompeticí všech čtyřech fluorescenčně značených modifikovaných nukleotidů, což přináší větší přesnost ve srovnání s přístroji založenými na postupném přidávání jednotlivých nukleotidů a snímáním změny signálu, které generují chyby v delších úsecích s identickými nukleotidy (homopolymerech). Další výhodou tohoto systému je poměrně rychlá příprava vzorků. Nevýhodou jsou poměrně krátké přečtené úseky, u starších typů do 100 bp; u přístroje MiSeq už jsou prodlouženy na 150–250 bp.
Firma Life Sciences distribuuje od roku 2008 přístroj SOLiD založený na sekvenaci ligací, který produkuje dosud nepřekonané množství stovek milionů krátkých sekvencí (<50 bp). Jako stolní variantu uvedla firma nedávno na trh přístroj založený na principu pHmetrie - Ion Torrent PGM. Princip metody je podobný systému 454 (navázání fragmentu nukleové kyseliny na mikrokuličku, oddělená amplifikace tohoto fragmentu cestou emulzní PCR, sekvenace na jednotlivých kuličkách), pouze je jako signál namísto chemiluminescence snímána změna pH. Přístroj má výhodu v poměrně nízké pořizovací ceně i ceně sekvenace, větší délce čtení (200bp) a možným odstupňováním požadovaného výkonu ve formě použití různě výkonných sekvenačních čipů. Příprava vzorku je delší než u přístrojů firmy Illumina, ale díky automatizaci emulzní PCR kratší než u přístrojů firmy Roche. Společnou nevýhodou přístrojů založených na postupném přidávání jednotlivých nukleotidů zůstává chybovost v homopolymerních sekvencích, v současnosti se však intenzívně pracuje na softwarovém řešení těchto chyb.
V současnosti probíhá vývoj nových sekvenátorů na nejrůznějších principech, které slibují další výhody, např. v podobě čtení jednotlivých molekul včetně modifikací nukleotidových bazí, čtení velmi dlouhých úseků apod. Je těžké určit, které z těchto přístrojů se prosadí v příštích letech na trhu. Někdy se přístroje rozlišují na tzv. sekvenátory druhé a třetí generace, kdy přístroje druhé generace ještě vyžadují PCR amplifikaci sekvenovaného produktu, zatímco přístroje třetí generace sekvenují pomocí nejrůznějších principů již jen jedinou molekulu.
BUDOUCNOST
V ideálním případě bude v horizontu cca 10 let molekulárně-genetická diagnostika nádorových onemocnění probíhat cestou celogenomové sekvenace vzorku tumoru zároveň se sekvenací normální tkáně pacienta. Spolu s narůstajícím množstvím studií a přenášením jejich výsledků do databází bude větší možnost předpovídat klinický význam objevených variací, zpřesnit rizikovou stratifikaci, odhadnout riziko dané choroby pro jedince apod. Díky tomu bude možno v horizontu týdnů stanovit u nádoru prognosticky relevantní změny, aberace vhodné k léčebným zásahům pomocí cílené léčby, farmakogenomicky důležité polymorfismy apod. a zároveň bude k dispozici informace o genetické predispozici pro případné poradenství v rámci rodiny. Tato možnost ale předpokládá zlepšení finanční dostupnosti celé procedury a její hladké zavedení do běžné klinické praxe. Reálnější je možnost, že se MPS uplatní v laboratorní praxi patologa především při vyšetřování mutací v prognosticky významných genech, nejspíše v podobě multiplexního sekvenování desítek až stovek relevantních genů na bázi amplikonové sekvenace. Tento přístup je již v současnosti cenově srovnatelný s klasickou sekvenací a přitom je rychlejší a citlivější. V horizontu cca 5 let bude zřejmě ze stejných důvodů pomocí MPS prováděno měření minimální zbytkové choroby pomocí genových mutací nebo u malignit z lymfoidní řady pomocí klonálně-specifických přestaveb genů pro imunoglobuliny a T-buněčné receptory.
Rychlý rozvoj nových metod vyžaduje a bude vyžadovat neustálé doplňování informací a velmi kritický přístup při porovnávání přínosu jednotlivých přístupů v konkrétní situaci. Na jednu stranu bude třeba včas nahradit zastaralé metody, ale zároveň bude nutné v zavádění nových technologií zachovávat „zdravý rozum“, aby v některých případech nebyly zbytečně opuštěny současné osvědčené a cenově efektivní postupy.
PODĚKOVÁNÍ
Podpořeno projektem (Ministerstva zdravotnictví) koncepčního rozvoje výzkumné organizace 00064203 (FN MOTOL).
Adresa pro korespondenci:
MUDr. Eva Froňková, Ph.D.
Laboratoř molekulární genetiky CLIP
Klinika dětské hematologie a onkologie 2. LF UK a FN Motol
V Úvalu 84, 150 06 Praha 5
tel.: 22443 6487, fax: 224436521
eva.fronkova@lfmotol.cuni.cz
Sources
1. Meyerson M, Gabriel S, Getz G. Advances in understanding cancer genomes through second-generation sequencing. Nat Rev Genet 2011; 11(10): 685–696.
2. Welch JS, Link DC. Genomics of AML: clinical applications of next-generation sequencing. Hematology Am Soc Hematol Educ Program 2011; 30–35.
3. Teer JK, Mullikin JC. Exome sequencing: the sweet spot before whole genomes. Hum Mol Genet 2010; 19(R2): R145–151.
4. Gartner J, Davis S, Wei X, et al. Comparative exome sequencing of metastatic lesions provides insights into the mutational progression of melanoma. BMC Genomics 2012; 13 : 505.
5. Malone JH, Oliver B: Microarrays, deep sequencing and the true measure of the transcriptome. BMC Biol 2011; 9 : 34.
6. Logan AC, Gao H, Wang C, Sahaf B, et al. High-throughput VDJ sequencing for quantification of minimal residual disease in chronic lymphocytic leukemia and immune reconstitution assessment. Proc Natl Acad Sci USA 2011; 108(52): 21194 – 21199.
7. Lister R, Pelizzola M, Dowen RH, et al. Human DNA methylomes at base resolution show widespread epigenomic differences. Nature 2009; 462(7271): 315–322.
8. Glenn TC. Field guide to next-generation DNA sequencers. Mol Ecol Resour 2011; 11 : 759–769.
9. Loman NJ, Misra RV, Dallman TJ, et al. Performance comparison of benchtop high-throughput sequencing platforms. Nat Biotechnol 2012; 30(5): 434–439.
10. Liu L, Li Y, Li S, et al. Comparison of next-generation sequencing systems. J Biomed Biotechnol 2012; 251364.
11. Quail MA, Smith M, Coupland P, et al. A tale of three next generation sequencing platforms: comparison of Ion Torrent, Pacific Biosciences and Illumina MiSeq sequencers. BMC Genomics 2012; 13 : 341.
Labels
Anatomical pathology Forensic medical examiner Toxicology
Article was published inCzecho-Slovak Pathology

2013 Issue 3-
All articles in this issue
- Polymerase chain reaction: basic principles and applications in molecular pathology
- Sequencing – classical method
- Next-generation sequencing
- False aneurysm of the wall of a venous graft in a patient with an implanted MGuard type of coronary stent: case report and description of the microscopic changes
- Giant cell interstitial pneumonia without exposure to hard metals
- Endometriosis in a mesothelial cyst of tunica vaginalis of the testis. Report of a case
- Czecho-Slovak Pathology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Polymerase chain reaction: basic principles and applications in molecular pathology
- Sequencing – classical method
- Next-generation sequencing
- Endometriosis in a mesothelial cyst of tunica vaginalis of the testis. Report of a case
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

