-
Medical journals
- Career
Vzorec exprese homeodoménových genů nedefinuje známé podskupiny dětských akutních lymfoblastických leukémií
Authors: J. Stárková 1; B. Zámostná 2; E. Mejstříková 1; J. Trka 1
Authors‘ workplace: Klinika dětské hematologie a onkologie, 2. lékařská fakulta, Karlova univerzita, Praha přednosta prof. MUDr. J. Starý, DrSc. 1; Katedra genetiky a mikrobiologie, Přírodovědecká fakulta, Karlova univerzita, Praha přednostka prof. RNDr. Z. Palková, CSc. 2
Published in: Čes-slov Pediat 2010; 65 (9): 497-502.
Category: Original Papers
Overview
Účel studie:
Základní imunofenotypové a genotypové podskupiny dětských akutních lymfoblastických leukémií (ALL) mají charakteristický expresní profil RNA. Homeodoménové (HOX) geny hrají důležitou roli v normální hematopoeze a jejich role se předpokládá i v patogenezi akutních leukémií. Zkoumali jsme, zda expresní vzorec 21 HOX genů ze skupiny HOXA, HOXB a CDX definuje vybrané imunofenotypové a genotypové skupiny dětských ALL. Srovnáním s expresí HOX genů ve fyziologických prekurzorech lymfoidních buněk jsme sledovali, zda exprese v maligních buňkách odráží jejich relativní zralost.Metody:
Analyzovali jsme expresi HOX genů s použitím kvantitativní RT-PCR ve skupině dětských ALL a normálních prekurzorů lymfoidních buněk. K analýze dat jsme použili metodu hierarchického klastrování.Výsledky:
Přestože exprese jednotlivých HOX genů se významně lišila mezi jednotlivými podskupinami dětských ALL (T-ALL, B prekurzorová ALL s fúzními geny TEL/AML1, BCR/ABL, MLL/AF4 a hyperdiploidií), celkový expresní vzorec nedefinoval spolehlivě jednotlivé podskupiny. Srovnání exprese jednotlivých HOX genů ve vývojových stadiích fyziologických lymfoidních buněk prokázalo, že jejich maligní protějšky exprimují HOX geny nezávisle na své relativní zralosti.Závěry:
Leukemické buňky aberantně exprimují vybrané HOX geny. Stanovení expresního vzorce HOX genů však není vhodným prostředkem ke klasifikaci dětských leukémií. Vysoce exprimované HOX geny v jednotlivých skupinách mohou potenciálně sloužit jako cíle biologické léčby.Klíčová slova:
dětská akutní lymfoblastická leukémie, homeodoménové genyÚvod
Akutní lymfoblastická leukémie (ALL) je nejčastější dětskou malignitou. Pojem ALL v sobě zahrnuje celou řadu imunofenotypově a genotypově definovaných podskupin, které se odlišují linií původu, relativní zralostí postižené buňky, mechanismem maligní transformace a z toho vycházejícími biologickými vlastnostmi leukemických blastů. Biologické vlastnosti leukemických blastů ovlivňují klinické chování onemocnění, zejména odpověď na léčbu kombinovanou chemoterapií. Z tohoto důvodu vyžadují moderní léčebné protokoly (včetně v Čechách používaných protokolů německé skupiny Berlín – Frankurt – Münster: BFM) podrobnou charakterizaci leukemických buněk při diagnóze a následné sledování dynamiky leukemického klonu v časném stadiu léčby. Iniciální diagnostika je tak kromě klasické morfologie postavena zejména na určení linie a relativní zralosti leukemických buněk průtokovou cytometrií. Tato metoda je doplněna klasickou i fluorescenční cytogenetikou a metodami molekulární genetiky, založenými na technologii polymerázové řetězové reakce (PCR). Tyto přístupy detekují v leukemických buňkách prognosticky významné aberace, u ALL zejména početní změny chromozomů a translokace. Fúzní geny, vycházející z translokací BCR/ABL, TEL/AML1 a MLL/AF4 patří mezi důležité prognostické znaky u dětských ALL [1].
V posledním desetiletí se objevují pokusy využít ke klasifikaci dětských ALL i moderní postupy molekulární genetiky, tzv. čipové technologie. Prvním pokusem použít metodu expresního profilování – GEP, tedy gene expression profiling – byla studie Yeoha et al. v roce 2002 [2]. Přístup využívající metody kvantifikace transkriptů celého genomu leukemické buňky a následné statistické analýzy získaných dat (založené zejména na metodě hierarchického klastrování) by měl umožnit klasifikaci leukémie, určení podtypu (včetně identifikace nových, dosud neznámých podskupin) a také předpovědět chování leukemického klonu v odpovědi na léčbu. Další studie, využívající GEP, tento předpoklad obecně potvrdily, třebaže se jim vzhledem k nárokům na kvalitu zkoumaného materiálu a ceně vyšetření dosud nepodařilo vstoupit do praktické diagnostiky [3]. Vzhledem k tomu, že i v těchto studiích jsou klastry ve skutečnosti určovány expresním profilem jen řádově stovek nebo desítek genů, alternativou k celkovému GEP je tak vytipování menšího souboru genů. Ten pak může být vyšetřován jak čipovou technologií, tak klasickou reverzně transkriptázovou PCR (RT-PCR) [4].
Homeodoménové (HOX) geny kódují sadu vysoce konzervativních transkripčních faktorů, obsahujících DNA vazebný motiv (homeodoména) strukturovaný jako dvojitá smyčka složená z 60 aminokyselin. Jsou rozloženy na celkem 4 chromozomech (7, 17, 12 a 2) a rozdělují se do 4 skupin, klastrů A, B, C a D [5, 6]. Exprese těchto genů se mění v průběhu normální hematopoézy a zasahuje tak do vývoje krevních elementů. Studie zabývající se rolí jednotlivých HOX genů přinesly zajímavé informace o jejich vlivu na diferenciaci myeloidních i lymfoidních prekurzorů [7, 8].
Skupina Drabkina a spolupracovníků v sérii prací popsala, že expresní vzorec HOX genů definuje genotypové podskupiny pacientů s akutní myeloidní leukémií a má také prognostický význam. Vzhledem k omezenému počtu zkoumaných genů použili Drabkin et al. nikoli čipový přístup ke stanovení expresního vzorce, ale metodu založenou na kvantitativní reverzně transkriptázové PCR [9]. Tento postup jim umožnil individuální optimalizaci detekce transkriptů jednotlivých HOX genů a tím i přesnější kvantitifikaci jejich celkové exprese.
V naší studii jsme použili modifikovaný přístup Drabkina et al. – tedy stanovení expresního vzorce HOX genů z klastrů A a B a neklastrových HOX genů CDX – na iniciální vzorky dětských pacientů s ALL. Analyzovali jsme kvantitativně hladinu transkriptů 21 HOX genu (HOXA1, A2, A3, A4, A5, A6, A7, A9, A10, A11, A13, B2, B3, B4, B5, B6, B7, B8, B9, CDX1 a CDX2) ve vzorcích kostní dřeně 61 pacientů a v izolovaných podskupinách fyziologických lymfoidních progenitorů. Pomocí metody hierarchického klastrování jsme zjišťovali, zda expresní vzorec HOX genů koreluje s imunofenotypem a/nebo genotypem dětské ALL. Dále jsme měli v úmyslu určit, zda expresní vzorec HOX genů v leukemických buňkách odpovídá principiálně jejich relativní zralosti (v porovnání s fyziologickými protějšky), či zda se jedná o kompletně aberatní expresi vyvolanou procesem maligní transformace.
Materiál a metody
Izolace a příprava vzorků
Zralé B a T buňky byly izolovány z periferní krve zdravých dárců. Další B buněčná stadia byla získána z kostní dřeně zdravých jedinců (dětí rok po ukončení terapie nebo pacientů, u kterých nebyly hematologické malignity potvrzeny). Kortikální T buňky byly izolovány z thymů dětí, které prošly kardiochirurgickým zákrokem. Jednotlivé populace byly sortovány podle imunofenotypických znaků: CD19+, CD34+: B-progenitory (BLP; n = 3), CD19+, 34-, CD10+: B-prekurzory (pre-B; n = 3), CD19+, CD34-, CD10-, CD20+: zralé B buňky (zralé B; n = 3), CD19-, CD34+, CD33+: nezralé myeloidní progenitory (IMP; n = 4) a CD3+, CD45+: zralé T buňky (zralé T; n = 3).
Pro studium exprese HOX genů v leukemických buňkách byly testovány vzorky 61 dětských ALL pacientů léčených podle léčebných protokolů ALL BFM (Berlín – Frankfurt – Münster) 95, ALL BFM-IC 2002, POG 9407 a Interfant 99. Analyzovali jsme cDNA pacientů rozdělených podle fenotypu [T-ALL (n = 10), BCP-ALL (n = 51)], prognózy [PGR – prednisone good responders (n = 9), PPR – prednisone poor responders (n = 9)] a genotypu [BCR/ABL (n = 6), MLL/AF4 (n = 9), TEL/AML1 (n = 9), hyperdiploidní (n = 8); bez fúzních genů (n = 18)]. Věk pacientů byl mezi 1 měsícem a 13 lety. Práce na pacientských vzorcích byla schválena etickou komisí FN Motol, UK 2. LF. RNA byla izolována v pacientských vzorcích z 10×106 buněk, u fyziologických protějšků z množství získaného sortováním na průtokovém sorteru FACS Aria (BD, San Jose), pomocí modifikované metody popsané Chomczynskim a Sacchi nebo RNeasy Plus mini a RNeasy micro kitů (Qiagen GmbH, Hilden, Germany). Poté následovalo odstranění gDNA pomocí DNase I (Qiagen GmbH, Hilden, Germany). RNA pak byla přepsána do cDNA použitím iScript™cDNA Synthesis Kit (Bio-rad, Hercules, CA, USA).
Kvantitativní RT-PCR
Kvantitativní real-time PCR byla prováděná na přístroji iCycler iQ system (Bio-rad). Sekvence primerů byly publikovány již dříve s výjimkou HOXA6 (5´-CGAGCAGCAGTACAAACCCGACAGCA-3´; 5´-CGCCCATGGCTCCCATACACAGCAC-3´) a ABL1 (5´-TTCAGCGGCCAGTAGCATCTGACTT-3´; 5´-TGTGATTATAGCCTAAGACCCGGAGCTTTT-3´). Primery jsme navrhovali pomocí softwarů Primer Express 2.0 (Applied Biosystems, Foster City, CA) a Vector NTI (Invitrogen Corporation, Carlsbad, CA, USA). PCR reakce byla prováděna s 2,5 pmol primeru a 1 μl cDNA do finálního objemu 25 μl použitím iQ™SYBR Green Supermixu (Bio-rad, Hercules, CA, USA). Použili jsme ABL1 jako kontrolní gen, k jehož expresi jsme výsledky kvantifikace HOX genové exprese normalizovali. Všechny reakce byly prováděny v duplikátu.
Statistická analýza
Hierarchická klastrovací analýza (HCL) obecně řadí podobné objekty k sobě. Principem této statistické metody je opakovaný výpočet vzdálenosti mezi testovanými objekty, v našem případě se jednalo o euklidovskou vzdálenost. Pro HCL jsme použili MeV software (Boston, MA, USA).
Test Kruskalův-Wallise je neparametrický statistický test, který se používá pro testování shody populací ve třech a více skupinách. V našem případě sloužil k určení genů, jejichž exprese se nejsignifikantněji liší mezi skupinami. Statistické hodnoty jsme korigovali Bonferroniho koeficientem, abychom odstranili případnou falešnou pozitivitu vzniklou mnohočetným srovnáváním v rámci jednoho souboru.
Výsledky
Pomocí kvantitativní RT-PCR jsme vyšetřili expresi RNA 21 HOX genů u 61 pacientů s ALL rozdělených podle fenotypu na skupinu BCP-ALL a T-ALL pacientů, podle genotypu na TEL/AML1, BCR/ABL, MLL/AF4, hyperdiploidní leukémie a podskupinu s normálním karyotypem. Dalším kritériem byla odpověď na prednison v iniciální fázi léčby, na základě které jsme rozdělili pacienty s dobrou a špatnou odpovědí na prednison. Toto kritérium je jedním z hlavních stratifikačních ukazatelů v léčebném protokolu BFM používaném při léčbě ALL. Dále jsme vyšetřili fyziologické protějšky B a T lymfoidní řady izolované z kostní dřeně, thymu a periferní krve zdravých jedinců. Pomocí FAC sortování jsme izolovali šest vzorků: kortikální T-buňky, zralé T-buňky, nezralé myeloidní progenitory, B-prekurzory, pre-B a zralé B buňky.
Pomocí HCL s využitím euklidovské vzdálenosti jsme nejprve studovali, zda se pacientské vzorky seřadí do genotypových, fenotypových a/nebo prognostických skupin na základě expresního vzorce HOX genů (obr. 1).
Image 1. Hierarchická klastrovací analýza leukemických buněk pacientů s ALL a jejich fyziologických protějšků na základě exprese HOX genů. Sledováním exprese všech studovaných HOX genů (a) a HOX genů signifikantně rozdílných na základě analýzy Kruskalovým-Wallisovým testem (b) jsme provedli HCL u vzorků dětských ALL pacientů rozdělených podle fenotypu, genotypu a prognózy. Tyto vzorky jsme dále srovnávali s normálními fyziologickými protějšky B a T buněk. HCL podle míry shody exprese jednotlivých genů shlukuje vyšetřované vzorky do skupin (klastrů). 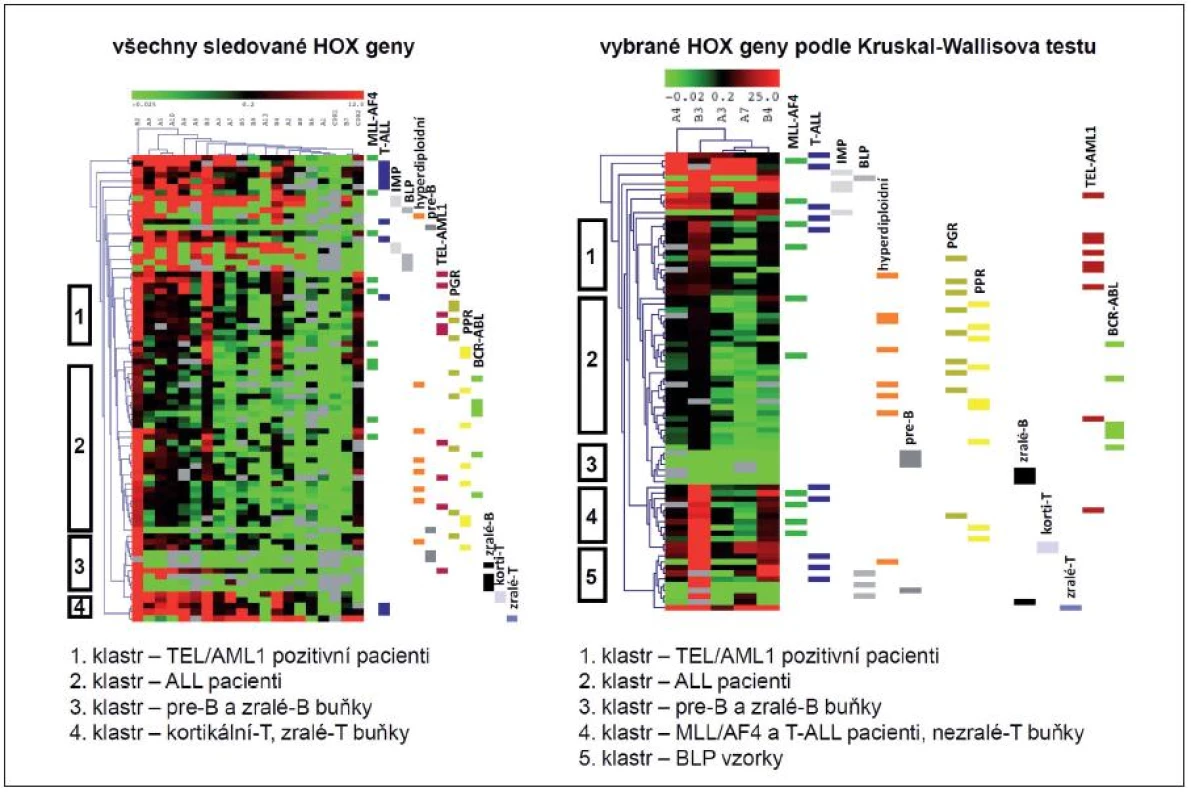
Z analýzy vyplývá, že HOX genová exprese u ALL nerozlišuje pacienty na základě těchto charakteristik. Pacientské vzorky vytvořily jeden společný klastr sestávající hlavně z pacientů hyperdiploidních, BCR/ABL pozitivních a pacientů s normálním karyotypem. Pacienti MLL/AF4 pozitivní a T-ALL pacienti se řadili vedle sebe anebo samostatně. Oddělila se pouze skupina pacientů s TEL/AML1 pozitivní leukémií. Celkově vychází, že pacienti s T-ALL a leukémií nesoucí MLL/AF4 mají nejvyšší hladinu exprese HOX genů v porovnání s pacienty s BCR/ABL pozitivní leukémií a s pacienty s normálním karyotypem, u kterých byla hladina exprese HOX genů nejnižší.
Exprese HOX genů v sortovaných populacích normálních B a T buněk se významně lišila. Nejvyšší exprese HOX genů byla detekována ve vzorcích IMP, z čehož HOXA1-A6, B5, B7-B9 byly výhradně exprimovány pouze v této sortované populaci. Dále geny HOXA7-10, A13, B2-4 byly transkribovány v obou populacích IMP a BLP. Pre-B a zralé B buňky neexprimovaly žádné HOX geny kromě HOXB2. Navíc HOXB6 a CDX2 nebyly transkribovány v žádném námi sledovaném stadiu vývoje B buněk. Klastrovací analýza zařadila normální buňky odděleně od vzorků pacientů – IMP a B-progenitory do jedné skupiny a pre-B a zralé B buňky do dalšího samostatného klastru.
Následně jsme detekovali HOX geny, které jsou mezi určitými skupinami exprimovány odlišně. Patří mezi ně zejména HOXA3, A4, A7, A9, A10, B3 a B4. Pomocí statistického testu jsme tento rozdíl potvrdili u genu pro HOXA3 (Kruskal-Wallis; p = 0,000098), A4 (p = 0,000619), A7 (p = 0,000045), B3 (p = 0,00015) a B4 (p = 0,001925). HCL tvořená pouze HOX geny signifikantními podle Kruskalova-Wallisova testu vytvořila ještě zřetelnější rozdělení. Rozdělení pacientských vzorků bylo stejné jako v předchozí analýze. Vzorky IMP a BLP sortovaných populací netvořily žádný klastr, pre-B a zralé B buňky klastrovaly společně. Kortikální T buňky exprimovaly všechny HOXA geny, zralé T buňky pouze HOXA1, A2, A3, A4 a A7, přičemž exprimovaly všechny HOXB geny kromě HOXB6. Pomocí HCL byly zařazeny samostatně.
Leukemické buňky BCP-ALL zapadají svým relativním vývojovým stadiem mezi BLP a pre-B buňky. Nejméně zralé jsou MLL/AF4 pozitivní leukemické buňky, nejvíce BCR/ABL pozitivní leukemické buňky. V leukemických buňkách pacientů s ALL byla detekována exprese genů HOXA2-A10, B2-B6 a CDX2. Exprese HOX genů v leukemických buňkách nekopíruje exprese vývojového stadia, jemuž odpovídá relativní zralost buňky. Z toho vyplývá, že u leukemických buněk dochází k re-expresi HOX genů, která je spojená s procesem maligní transformace.
Diskuse
Prvním důležitým krokem studie byla optimalizace technického postupu. Ta zahrnovala jednak optimalizaci všech PCR v našich podmínkách (včetně nového návrhu systému pro kvantifikaci genu HOXA6, jehož detekce originálním systémem nebyla zcela specifická), nalezení a otestování kontrolního genu pro normalizaci stanovených expresí na hladinu celkové RNA přítomné v buňce. Po vyladění všech technologických postupů jsme získali expresní vzorce zkoumaných 21 HOX genů pro všechny studované podskupiny dětských ALL: leukémie z T řady, leukémie z prekurzorů B řady bez známých genetických aberací, B prekurzorové leukémie nesoucí fúzní gen BCR/ABL, TEL/AML1 a MLL/AF4 a B prekurzorové leukémie s hyperdiploidním počtem chromozomů.
Současně jsme analyzovali expresi HOX genů v normálních protějšcích lymfoidních leukemických buněk. Samozřejmě je velmi obtížné definovat, co přesně takovým protějškem je, neboť tomu brání aberantní a asynchronní exprese některých antigenů. Technicky je potom překážkou omezený přístup k fyziologickému materiálu, resp. jeho množství, které významně limituje postupy FAC sortování. Omezili jsme se proto na podskupiny zralých a kortikálních T buněk jako protějšků leukémií z T řady; v B řadě jsme sortovali nezralé progenitory, dva vývojové stupně B prekurzorových buněk a konečně zralé B buňky. Všechny leukemické blasty, vycházející z podskupin B leukémií zařazených do studie, patří svou relativní zralostí právě mezi ony dva vývojové stupně fyziologických B prekurzorů. Pokud by platilo, že exprese jednotlivých HOX genů v leukemických buňkách odpovídá jejich relativní zralosti, musely by být exprimovány také alespoň v jednom z námi definovaných fyziologických B progenitorů. Tato hypotéza se však nepotvrdila, minimálně ve dvou případech jsme nalezli v leukemických buňkách expresi genů, jejichž transkripty nebyly vůbec detekovány v žádném studovaném vývojovém stadiu B řady. Je tedy zřejmé, že expresní vzorec HOX genů je alespoň u B prekurzorových leukémií určován aberantní expresí spuštěnou v průběhu maligní transformace a/nebo se na ní přímo podílející.
Hlavní otázkou naší studie však bylo, zda expresní profil 21 genů z HOX klastrů A, B a neklastrových genů CDX může zařadit leukemické blasty do imunofenotypových a/nebo genotypových podskupin dětských ALL. Třebaže exprese některých HOX genů byla charakteristicky zvýšená u určitých podskupin (HOXA3, A4, A7, B3 a B4), celkový expresní vzorec nedokázal na rozdíl od AML rozřadit v hierarchické analýze pacienty s ALL do definovaných imunofenotypových a genotypových skupin s výjimkou oddělení nekompletní skupiny pacientů s TEL/AML1 pozitivní ALL. Toto selhání by bylo možno u části námi použitých skupin připsat na vrub nedostatečného počtu vzorků v jednotlivých skupinách nebo náhodného výběru do skupin s dobrou a špatnou odpovědí na iniciální léčbu prednisonem. Pohled na čísla vyšetřených pacientů ve studii Drabkina et al. [9, 10] však napovídá, že toto vysvětlení není pravděpodobné. Navíc, u vybraných genotypových skupin (jakými jsou bezpochyby dobře definované a homogenní skupiny pacientů s fúzním genem BCR/ABL, MLL/AF4 nebo TEL/AML1) tento argument neplatí zcela jistě. Je tedy zřejmé, že na rozdíl od AML expresní vzorec HOX genů u dětských pacientů s ALL nemůže sloužit k jejich klasifikaci.
Nízká výpovědní hodnota expresního vzorce jako celku je však jen jedním aspektem práce. Nalezení specifické aberantní exprese jednotlivých HOX genů u určitých subtypů dětských ALL přináší další možnosti využití. Vzhledem ke známé i předpokládané roli těchto genů v normální hematopoéze i leukemogenezi mohly by jejich transkripty potenciálně sloužit jako cíle pro biologickou léčbu. Stejně tak nalezená aberantní exprese HOX genů může přispět k objasnění jejich role v leukemogenezi.
Podpořeno grantem IGA NR/9526-3.
Mgr. Júlia Starková, Ph.D.
CLIP
Klinika dětské hematologie a onkologie
2. lékařská fakulta, Karlova univerzita
V Úvalu 84
150 06 Praha 5
e-mail: julia.starkova@lfmotol.cuni.cz
Sources
1. Pui CH, Robison LL, Look AT. Acute lymphoblastic leukaemia. Lancet 2008; 371 : 1030–1043.
2. Yeoh EJ, Ross ME, Shurtleff SA, et al. Classification, subtype discovery, and prediction of outcome in pediatric acute lymphoblastic leukemia by gene expression profiling. Cancer Cell. 2002; 1 : 133–143.
3. Cario G, Stanulla M, Fine BM, et al. Distinct gene expression profiles determine molecular treatment response in childhood acute lymphoblastic leukemia. Blood 2005; 105 : 821–826.
4. Holleman A, den Boer ML, de Menezes RX, et al. The expression of 70 apoptosis genes in relation to lineage, genetic subtype, cellular drug resistance, and outcome in childhood acute lymphoblastic leukemia. Blood 2006; 107 : 769–776.
5. Abramovich C, Humphries RK. Hox regulation of normal and leukemic hematopoietic stem cells. Curr. Opin. Hematol. 2005; 12 : 210–216.
6. Grier DG, Thompson A, Kwasniewska A, et al. The pathophysiology of hox genes and their role in cancer. J. Pathol. 2005; 205 : 154–171.
7. Calvo KR, Sykes DB, Pasillas M, et al. Hoxa9 immortalizes a granulocyte-macrophage colony-stimulating factor-dependent promyelocyte capable of biphenotypic differentiation to neutrophils or macrophages, independent of enforced meis expression. Mol. Cell Biol. 2000; 20 : 3274–3285.
8. Thorsteinsdottir U, Sauvageau G, Hough MR, et al. Overexpression of hoxa10 in murine hematopoietic cells perturbs both myeloid and lymphoid differentiation and leads to acute myeloid leukemia. Mol. Cell Biol. 1997; 17 : 495–505.
9. Drabkin HA, Parsy C, Ferguson K, et al. Quantitative hox expression in chromosomally defined subsets of acute myelogenous leukemia. Leukemia 2002; 16 : 186–195.
10. Andreeff M, Ruvolo V, Gadgil S, et al. Hox expression patterns identify a common signature for favorable AML. Leukemia 2008; 22 : 2041–2047.
Labels
Neonatology Paediatrics General practitioner for children and adolescents
Article was published inCzech-Slovak Pediatrics

2010 Issue 9-
All articles in this issue
- Vzorec exprese homeodoménových genů nedefinuje známé podskupiny dětských akutních lymfoblastických leukémií
- Vplyv iniciálneho ventilačného manažmentu na incidenciu bronchopulmonálnej dysplázie a ďalšie ochorenia u novorodencov narodených v 24.–27. týždni tehotenstva na Novorodeneckej klinike FNsP Nové Zámky
- Hydrocefalus u novorodencov – význam sonografického vyšetrenia mozgu
- Kongenitálny hyperinzulinizmus – najčastejšia príčina perzistujúcich hypoglykémií u novorodencov a dojčiat
- 19. kongres Európskej spoločnosti pre pediatrický klinický výskum
- Abstrakta
- Czech-Slovak Pediatrics
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Kongenitálny hyperinzulinizmus – najčastejšia príčina perzistujúcich hypoglykémií u novorodencov a dojčiat
- Hydrocefalus u novorodencov – význam sonografického vyšetrenia mozgu
- Vplyv iniciálneho ventilačného manažmentu na incidenciu bronchopulmonálnej dysplázie a ďalšie ochorenia u novorodencov narodených v 24.–27. týždni tehotenstva na Novorodeneckej klinike FNsP Nové Zámky
- Vzorec exprese homeodoménových genů nedefinuje známé podskupiny dětských akutních lymfoblastických leukémií
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

