-
Medical journals
- Career
Detekce EGFR mutací v cirkulující nádorové DNA (ctDNA) v plazmě – mezilaboratorní porovnání referenčních laboratoří v České republice
Authors: Čapková Linda 1; Kalinová Markéta 1; Tichá Ivana 2; Parobková Eva 3; Matějčková Milada 3; Vošmiková Hana 4; Horký Ondřej 5; Bartáková Karolína 5; Drábek Jiří 6; Bajerová Monika 7; Dundr Pavel 2
Authors‘ workplace: Ústav patologie a molekulární medicíny, 2. LF UK a FN Motol, Praha 1; Ústav patologie, 1. LF UK a VFN v Praze 2; Oddělení patologie a molekulární medicíny, Thomayerova nemocnice, Praha 3; Fingerlandův ústav patologie, LF UK a FN Hradec Králové 4; Oddělení onkologické patologie, Masarykův onkologický ústav, Brno 5; Ústav molekulární a translační medicíny, LF UP v Olomouci 6; Centrum molekulární biologie a genové terapie, Interní hematologická a onkologická klinika LF MU a FN Brno 7
Published in: Klin Onkol 2018; 31(5): 353-360
Category: Original Articles
doi: https://doi.org/10.14735/amko2018353Overview
Úvod:
Mutační analýza genu pro receptor epidermálního růstového faktoru (epidermal growth factor receptor – EGFR) z nádorové tkáně je v současnosti standardním testem u pacientů s nemalobuněčným karcinomem plic. Molekulární testování cirkulující nádorové DNA (circulating tumor DNA – ctDNA) z plazmy se ukazuje být spolehlivou alternativou detekce mutací v případech, kdy není možné získat bioptický či cytologický vzorek z nádoru či je třeba monitorovat léčebnou odpověď pomocí opakovaných vyšetření. Navíc dle recentních prací ctDNA lépe odráží heterogenitu nádorového procesu než odběr z izolované nádorové léze či metastázy. Za účelem zhodnocení analytické kvality testování mutací v genu EGFR z ctDNA izolované z plazmy bylo zorganizováno mezilaboratorní porovnání v rámci referenčních laboratoří prediktivní diagnostiky v České republice.
Materiál a metody:
Celkem 7 laboratořím bylo zasláno 13 komerčně dostupných referenčních vzorků 2 ml plazmy obsahující nejčastější senzitivní mutace genu EGFR (delece v exonu 19, L858R) a rezistentní mutaci T790M s frekvencí mutací 5; 0,5 a 0,05 %. Jeden vyšetřovaný vzorek obsahoval nemutovanou DNA. Vzorky byly analyzovány metodami standardně využívanými v diagnostické praxi. V 6 ze 7 laboratoří byl použit cobas® EGFR Mutation Test v2, v 1 laboratoři Super-ARMS® EGFR Mutation Detection Kit.
Výsledky:
Určeno bylo 91 genotypů s celkovou chybovostí 24,2 % (22/91). Na 0,5% a vyšší úrovni mutační frekvence činila celková chybovost 3,2 % (2/63), na 5% úrovni 0 % (0/35). Nebyly detekovány žádné falešně pozitivní výsledky. Platforma cobas® dosáhla konzistentně úspěšné detekce na hladině 0,05 % pro deleci v exonu 19. Pro mutace L858R a T790M byl práh detekce nad hodnotou 0,5 %.
Závěr:
Naše výsledky poukazují na limitovanou citlivost vyšetření mutací EGFR z plazmy, a to zejména mutace T790M. Obzvláště pro vyšetřování ctDNA z plazmy, jejíž frakce může být velice nízká (až 0,01 %), je nutné klást důraz na používání vysoce senzitivních molekulárních metod. Výsledky tohoto porovnání kvality potvrzují nutnost pokusu o rebiopsii u pacientů s negativním výsledkem, neboť oproti vyšetření solidního vzorku tkáně stoupá podíl falešně negativních případů.
Klíčová slova
cirkulující DNA – tekutá biopsie – receptor epidermálního růstového faktoru – nemalobuněčný karcinom plic – externí posouzení kvality
Úvod
Karcinom plic (lung cancer – LC) zůstává globálně hlavní příčinou mortality na nádorová onemocnění. Většina pacientů je stále diagnostikována v pokročilém stadiu s nepříznivou prognózou [1]. Objevení řídících mutací v genu kódujícím receptor epidermálního růstového faktoru (epidermal growth factor receptor – EGFR) jako primární onkogenní změny u části adenokarcinomů plic vedlo k vývoji specifických tyrozinkinázových inhibitorů (TKI) tohoto receptoru [2]. Mutace genu EGFR se vyskytují zejména v jeho intracelulární doméně, v exonu 18–21. Celosvětově jsou přítomny u 10–30 % pacientů s nemalobuněčným karcinomem plic (non-small cell lung cancer – NSCLC) [3,4], v europoidní (kavkazské) populaci jsou identifikovány přibližně u 10 % nemocných [5]. Delece v exonu 19 (del 19) a bodové mutace L858R (c.2573T>G, c.2573_2574TG>GT; p.Leu858Arg) v exonu 21 genu EGFR představují 85–90 % všech popsaných mutací genu EGFR a souvisejí se zvýšenou senzitivitou pacientů k léčbě EGFR-TKI [2]. Oproti tomu bodová substituce c.2369C>T, vedoucí k záměně aminokyseliny threonin za methionin v kodonu 790 (p.Thr790Met, T790M), a inzerce v exonu 20 jsou asociovány s rezistencí na EGFR-TKI [6,7]. U pacientů s pokročilým NSCLC a prokázanou senzitivní mutací EGFR, kteří byli léčeni EGFR-TKI (gefitinibem, erlotinibem či afatinibem) zaznamenalo několik velkých klinických studií signifikantní prodloužení intervalu do progrese onemocnění. Došlo též k prodloužení celkové délky přežití (na více než 24 měsíců) a zlepšení kvality života ve srovnání s pacienty léčenými konvenční chemoterapií [8–12]. Pacienti s pokročilým NSCLC neskvamózní histologie by proto měli být vyšetřeni na přítomnost mutací genu EGFR před zahájením léčby 1. linie. V průběhu 10–14 měsíců léčby EGFR-TKI se u naprosté většiny pacientů vyvine v důsledku nádorové heterogenity klonální evoluce a selekce rezistence vůči této terapii. Ta je až v 60 % případů způsobena bodovou mutací T790M v exonu 20 [3,13,14]. V nedávné době byly vyvinuty ireverzibilní TKI 3. generace (osimertinib, rociletinib) namířené též proti EGFR mutaci T790M a překonávající tuto rezistenci [15]. Randomizované studie prokázaly významný rozdíl v přežití pacientů s mutací T790M léčených osimertinibem oproti chemoterapii [16], a proto posílil význam opakování biopsie při progresi onemocnění za účelem detekce této genetické změny.
Primárním zdrojem materiálu pro molekulární analýzu u pacientů s pokročilým LC jsou tradičně vzorky tkáně získané bioptickým odběrem, příp. vzorky cytologické. Nicméně u velké části pacientů s metastatickým nádorovým onemocněním není možné tento materiál pro nepřístupnost léze získat vůbec či s ohledem na jejich klinický stav provádět vyšetření opakovaně a monitorovat tak dynamický vývoj mutačního stavu nádoru v průběhu terapie [17]. Rovněž v rámci vhodně navržených rozsáhlých klinických studií (IPASS, INTEREST, FLEX) bylo pouze u 20–40 % pacientů možné získat k molekulárnímu vyšetření dostatek materiálu ze solidní léze [8,10,18,19]. Dalším limitujícím faktorem odběru z jednoho ložiska (primárního či metastatického) je fakt, že nemusí odrážet biologickou heterogenitu nádoru a jeho metastáz [20,21].
Neinvazivním zdrojem materiálu pro detekci EGFR mutací se v současnosti ukazuje být tzv. tekutá biopsie, resp. jedna z jejích forem, stanovení volné nádorové DNA cirkulující v plazmě (circulating tumor DNA – ctDNA) [22,23]. Přestože mechanizmus uvolňování ctDNA do krve není přesně známý, zřejmě se jedná o proces úzce spjatý s apoptózou či nekrózou nádorových buněk a zčásti může jít i o aktivní sekreci [23–25]. Výhodou ctDNA je, že dle experimentálních výsledků obsahuje reprezentativní složky celého nádorového genomu [26]. Rovněž četné klinické studie prokázaly, že její detekce v periferní krvi u pacientů s LC ve vysokém procentu případů koreluje s nálezem mutace v nádorové tkáni, a tudíž může být použita k určení mutačního stavu EGFR a predikci účinnosti léčby EGFR-TKI [22,27–33]. Též aktualizované doporučené postupy profesních společností College of American Pathologists, International Association for the Study of Lung Cancer a Association for Molecular Pathology uvádějí vyšetření ctDNA ze vzorku plazmy jako alternativní test mutačního statusu v případě, že vzorek solidní tkáně není dostačující anebo není k dispozici [34]. Přes všechny výhody testování plazmy je nutno podotknout, že plazmatická DNA je vysoce fragmentovaná (nejčastěji o velikosti okolo 180 bp), což znesnadňuje účinnou amplifikaci cílové oblasti, a může být přítomna ve velmi nízké koncentraci. Frakce ctDNA z celkové volné DNA v plazmě (circulating free DNA – cfDNA) kolísá mezi 0,01 a 10 % [35,36]. Proto je v posledním desetiletí věnována velká pozornost vývoji vysoce citlivých, specifických a v neposlední řadě cenově dostupných molekulárních metod k detekci ctDNA [23,29,31,37–43].
Pro diagnostickou praxi byly regulačními orgány Evropské unie i Spojených států amerických (European Medicine Agency, U.S. Food and Drug Administration) v nedávné době schváleny první testy k provádění testování EGFR z plazmy u pacientů s LC jako prediktoru léčebné odpovědi na terapii gefitinibem v případech, kde není možné získat diagnostický vzorek tkáně [25]. Mezi tyto testy patří zejména cobas® EGFR Mutation Test v2 [44] založený na alelicky specifické polymerázové řetězové reakci (polymerase chain reaction – PCR) a Therascreen® EGFR RGQ Plasma PCR kit [45]. Navíc se ukázala metoda detekce ctDNA vhodná k monitorování vývoje rezistence na léčbu EGFR-TKI a relapsu onemocnění [29,46,47].
Vzhledem ke skutečnosti, že v Evropě nebylo do počátku roku 2018 širšímu počtu laboratoří dostupné externí hodnocení kvality testování mutačního stavu EGFR ctDNA v plazmě u pacientů s NCSLC, bylo zorganizováno mezilaboratorní porovnání na úrovni ČR. Cílem této studie bylo zhodnotit účinnost metod používaných v referenčních laboratořích prediktivní diagnostiky v ČR k detekci EGFR mutací v krevní plazmě.
Materiál a metody
Vzorky
V rámci studie byly každou ze 7 zúčastněných referenčních laboratoří (tab. 1) vyšetřovány 2 totožné sady 13 standardů obsahujících 2 ml plazmy značené „EGFR cfDNA Ring Trial Pack 1 Plasma DNA“ a „EGFR cfDNA Ring Trial Pack 2 Plasma DNA“ (Horizon Discovery Group plc, Velká Británie). Testovány byly 4 sady různých mutací genu EGFR s frekvencí mutované alely 5; 0,5 a 0,05 % a jeden vzorek bez mutované DNA (wild type) (tab. 2). Vzorky plazmy byly až do zpracování uchovávány při –80 °C.
Table 1. Seznam zúčastněných referenčních laboratoří prediktivní diagnostiky. 
Table 2. Vyšetřované standardy (Horizon Discovery Group plc – standard samples). 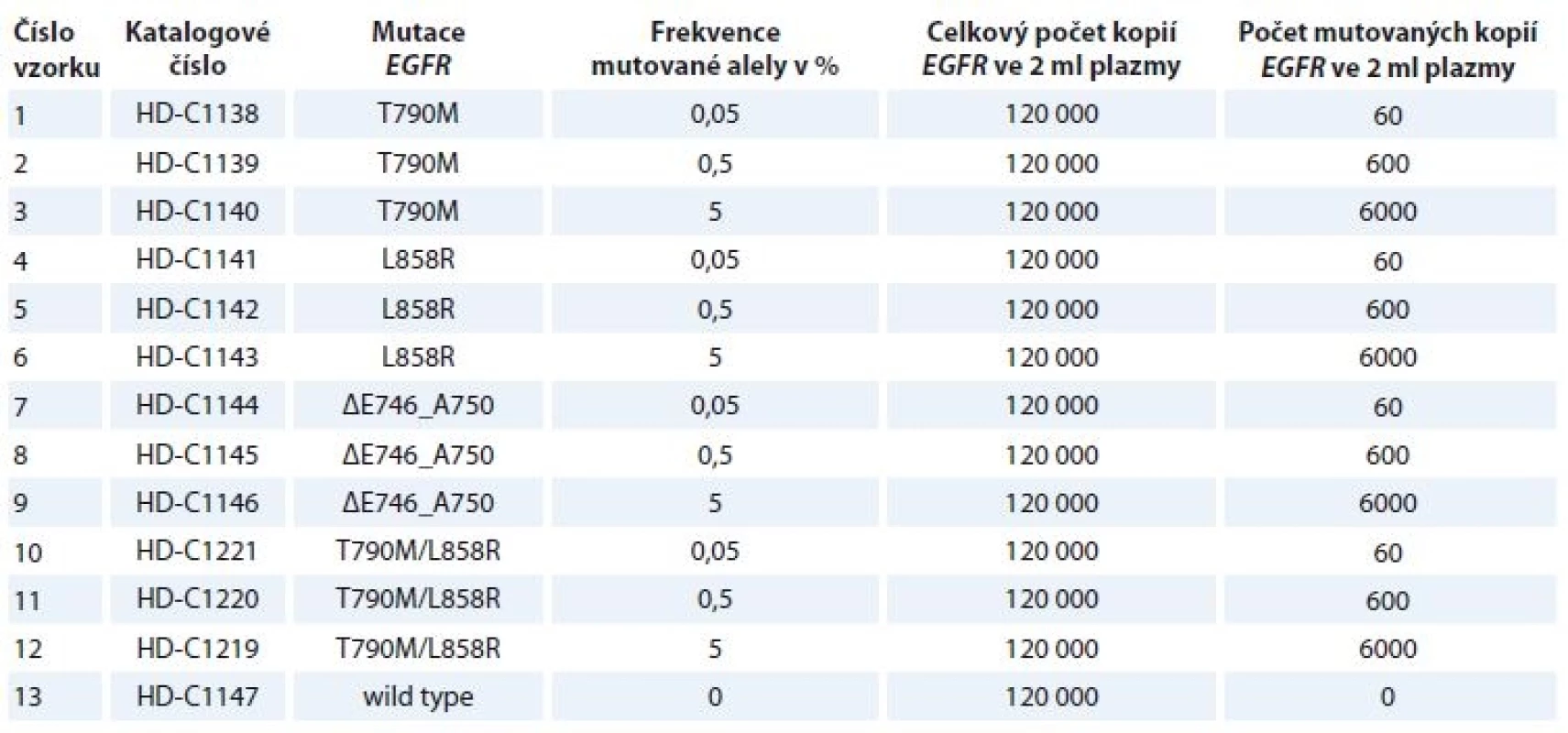
Pozn.: Standard ΔE746_A750 je registrovaný v databázi COSMIC jako nejčastější z delecí v exonu 19 genu EGFR [57]. Extrakce DNA
K izolaci cfDNA z plazmy byl použit v 6 případech certifikovaný (in vitro diagnostics – CE IVD) izolační kit cobas® cfDNA Sample Preparation Kit (Roche Molecular Systems Inc., Švýcarsko), v jednom případě pak CE IVD kit QIAamp Circulating Nucleic Acid Kit (Qiagen, Německo). Postupováno bylo dle instrukcí výrobce. Izolovaná cfDNA byla eluována do 100 ml DNA elučního pufru, z nichž bylo po finální centrifugaci k dalšímu použití odebráno 80 ml eluátu. Koncentrace DNA byla kvantifikována pomocí spektrofotometru NanoDrop® ND-1000 (Thermo Fisher Scientific, USA) či VivaSpec® (Sartorius Stedim Biotech, Německo) nebo semikvantitativně elektroforeticky. Extrahovaná cfDNA byla uchována při –20 °C, příp. byla analyzována bezprostředně po izolaci.
Mutační analýza pomocí PCR (COBAS®, Super-ARMS®)
Mutační stav genu EGFR v cfDNA izolované z plazmy byl stanoven pomocí PCR v reálném čase. V 6 laboratořích byl použit kit cobas® EGFR Mutation Test v2 (Roche Molecular Systems Inc., Švýcarsko) na přístroji cobas® 4800 (Roche Molecular Systems Inc., Švýcarsko). Tato sada detekuje mutace G719A/C/S v exonu 18, 29 různých delecí v exonu 19, mutace S768I, T790M a 5 odlišných inzercí v exonu 20 a mutace L858R a L861Q v exonu 21. Pro každou reakci bylo použito 25 ml neředěného eluátu cfDNA. V jedné z referenčních laboratoří byly vzorky vyšetřovány kitem Super-ARMS® EGFR Mutation Detection Kit (AmoyDx, ČLR) využívajícím ARMS-PCR (amplification refractory mutation system – ARMS) na přístroji Stratagene Mx3000P® (Agilent Technologies, USA). Vyšetřované mutace jsou obdobné jako u předchozího výrobce, navíc sada umožňuje detekci jedné další inzerce v exonu 20. Reakční směsi a jejich analýza byly provedeny podle instrukcí výrobců.
Výsledky
Výsledky genotypizace v jednotlivých referenčních laboratořích jsou shrnuty v tab. 3.
Table 3. Výsledky genotypizace vyšetřovaných standardů plazmy v jednotlivých referenčních laboratořích (I–VII). 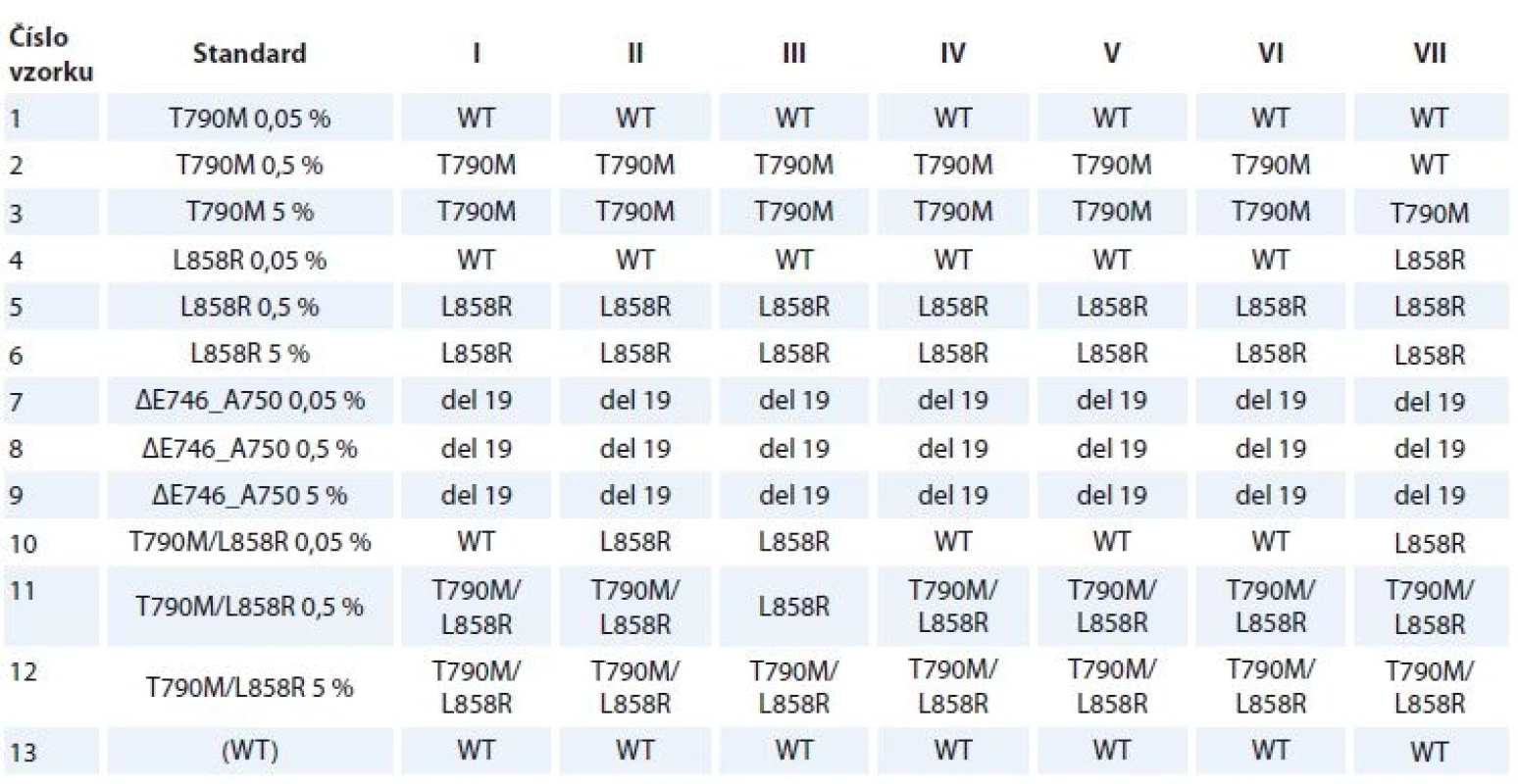
del 19 – delece v exonu 19 genu EGFR, WT – wild type, nemutovaná DNA Mutace T790M v exonu 20 ve standardu obsahujícím tuto mutaci s frekvencí 0,05 % nebyla detekována ani jedním kitem a žádným z účastníků studie. Metodou cobas® byla ve vzorcích s frekvencí 0,5 a 5 % detekována všemi zúčastněnými laboratořemi, metodou Super-ARMS® pak pouze ve vzorku s mutační frekvencí 5 %. Mutace L858R v exonu 21 nebyla metodou cobas® detekována ve vzorku s frekvencí 0,05 % žádným účastníkem, naopak vzorky s frekvencí mutace 0,5 a 5 % byly správně identifikovány všemi zúčastněnými laboratořemi. Metodou Super-ARMS® byla anotovaná varianta L858R detekována ve všech vzorcích, tj. s frekvencí 0,05; 0,5 a 5 %. Delece v exonu 19 (c.2235_2249del15, ΔE746_A750) byla oběma metodami a všemi účastníky stanovena ve všech vzorcích s touto anotovanou genetickou změnou, tj. s mutační frekvencí 0,05; 0,5 a 5 %. Kombinaci mutací T790M a L858R detekovali všichni účastníci správně ve vzorku s 5% frekvencí obou mutací. Správně zaznamenalo obě varianty ve vzorku s 0,5% frekvencí obou mutací 6 ze 7 laboratoří, přičemž v 1 laboratoři byla detekována pouze varianta L858R. Ve vzorku s frekvencí 0,05 % nebyly varianty detekovány na 4 pracovištích. Ve 2 laboratořích byla detekována pouze varianta L858R, žádný z účastníků nezachytil v tomto vzorku anotovanou variantu T790M. Vzorky s nemutovanou (wild type) DNA byly shodně všemi zúčastněnými hodnoceny jako negativní. Žádné falešně pozitivní výsledky nebyly zaznamenány.
Průměrná chybovost byla vypočítána jako součet všech falešně pozitivních a falešně negativních výsledků dělený počtem všech vyšetření. Celkově bylo stanoveno 91 genotypů s průměrnou chybovostí 24,2 % (22/91). Průměrná chybovost činila v případě vyšetření senzitivních mutací 14,3 % (6/42), pro mutaci T790M 38,1 % (16/42) a pro wild type DNA 0 % (0/7). Vezmeme-li v úvahu pouze výsledky s frekvencí mutací 0,5 % a vyšší a vyšetření wild type DNA, činila celková chybovost 3,2 % (2/63). Pro výstupy na úrovni 5% frekvence mutací a wild type DNA dosáhla chybovost 0 % (0/35). Ve všech případech se jednalo o falešně negativní výsledky.
Při porovnání obou použitých metod byla chybovost obdobná (24,4 % (19/78) u testu cobas®, resp. 23,1 % (3/13) u metody Super-ARMS®).
Diskuze
S rozvojem terapeutických možností v oblasti péče o pacienty s pokročilým NSCLC je kladen důraz na precizní molekulární diagnostiku potenciálních léčebných cílů u co největšího procenta nemocných. Jedná se zejména o primární detekci mutací genu EGFR a možnost monitorování léčby EGFR-TKI s případným vznikem rezistence. Rezistentní mutace T790M v exonu 20 genu EGFR se vyskytuje až u 60 % pacientů jako sekundární rezistence v důsledku léčby EGFR-TKI. Její detekce představuje novou výzvu, neboť hraje klíčovou roli v indikaci EGFR-TKI 3. generace (osimertinibu) [14,48]. Molekulární vyšetření z opakované biopsie v době progrese onemocnění tak ovlivňuje další terapeutická rozhodnutí. Vzhledem k jednoduchosti a nízkému riziku odběru periferní krve pro následnou separaci plazmy v porovnání s invazivním bioptickým či cytologickým odběrem a u pacientů, kde není možné získat materiál z primárního nádoru, se detekce EGFR mutací z ctDNA izolované z plazmy (tzv. tekutá biopsie) stává metodou první volby. Zatímco malý bioptický vzorek nemusí být reprezentativní pro celkovou nádorovou masu [49], vzorek tekuté biopsie je rychle dostupný, postihuje intra-a intertumorální heterogenitu, a může tak představovat výhodné řešení pro monitorování molekulárních změn v nádoru, léčebné odpovědi či sekundárně vzniklé rezistence na cílenou terapii [35].
Prediktivní vyšetření ctDNA nicméně skýtá i řadu nevýhod. Frakce ctDNA kolísá mezi 0,01 až více než 10 % cfDNA v závislosti na typu nádoru, stadiu nádorového onemocnění, celkovém množství nádorových buněk a jejich schopnosti invadovat do cévního řečiště [35,36]. K obtížné detekci navíc přispívá vysoká fragmentace ctDNA. K vyšetření EGFR ctDNA z plazmy byla v posledních několika letech vyvinuta celá řada metod založených primárně na PCR [37]. Pouze u části z nich jsou však k dispozici spolehlivá data z prospektivních validačních studií s porovnáním výsledků s referenční nádorovou tkání. Aby mohlo dojít k rozšíření detekčních platforem do praxe, musejí tyto být navíc komfortní, efektivní a současně ekonomicky únosné. Jedná se jak o přístupy digitální PCR (droplet digital PCR; beads, emulsions, amplification and magnetics – BEAMing dPCR; chip-based dPCR), tak platformy nedigitální (cobas®, therascreen®). Samozřejmě nelze opomenout možnosti sekvenování nové generace (new generation sequencing – NGS). Metody digitální PCR v kapičkách reakční směsi dosahují sice velmi nízkého limitu detekce (limit of detection – LoD), a to až 0,01 %, nicméně jsou poměrně finančně i časově náročné [38]. Senzitivita a specificita detekce ctDNA jednotlivých metod se při porovnávání vůči nádorové tkáni dle použité technologie do jisté míry liší, avšak konkordance výsledků je vysoká (okolo 90 %) [39,49]. Pro detekci delecí v exonu 19 a mutace L858R se u různých platforem pohybuje senzitivita mezi 67–93 % a specificita mezi 87–100 %. Detekce rezistentní mutace T790M má senzitivitu a specificitu o něco nižší (61–81, resp. 58–67 %) [29,31,36,40,41,43,50].
K zavedení vyšetřování ctDNA do klinické praxe je nutná spolehlivost a reprodukovatelnost výsledků molekulárního vyšetření, garantovaná úspěšnými výsledky externí a interní kontroly kvality. Evropští poskytovatelé kontrol kvality pod záštitou organizace International Quality Network for Pathology v roce 2016 provedli průzkum používaných metodik k testování ctDNA z plazmy u genů EGFR a RAS na 167 pracovištích v různých zemích [51]. Na jeho základě v létě 2017 zorganizovali za účasti 32 vybraných laboratoří z celého světa společné pilotní kolo externího posouzení kvality (external quality assessment – EQA) pro izolaci a analýzu EGFR ctDNA z plazmy [52]. V letošním roce je organizováno pilotní schéma EQA pro široký okruh zájemců [53]. Vzhledem k dosavadní nedostupnosti EQA vyšetřování EGFR ctDNA v plazmě bylo zorganizováno mezilaboratorní porovnání na úrovni ČR. V rámci naší studie jsme v 7 referenčních laboratořích ČR za použití komerčně dostupného standardizovaného materiálu (s 5; 0,5 a 0,05% frekvencí mutací) validovali metodiku testování mutačního stavu EGFR u pacientů s NSCLC.
Ke genotypizaci byly použity 2 různé metodiky, přičemž jejich celková chybovost byla obdobná (24,4 % (19/78) u metody cobas®, resp. 23,1 % (3/13) u metody Super-ARMS®). Platforma Super-ARMS® však byla zastoupena pouze jednou laboratoří a co se týče porovnání obou metodik, nelze z této práce vyvozovat bližší závěry. Obě metody použité ve studii jsou schopné poměrně spolehlivě správně detekovat mutaci s vyšší než 0,5% frekvencí jak senzitivních mutací (del 19, L858R), tak rezistentní mutace T790M. U vyšetření s frekvencí mutací 0,5 % a vyšší a vyšetření wild type DNA činila celková chybovost 3,2 %. Při posouzení vzorků s frekvencí mutací 5 % byly všechny výsledky správné (chybovost 0 %, 0/35). Nejnižší citlivost obou metod pro detekci varianty T790M se projevuje při typizaci vzorků s velmi nízkým podílem (0,05 %) mutované ctDNA (graf 1). Metoda cobas® dosáhla v nejnovějších publikovaných studiích pro detekci T790M v plazmě senzitivity 60–73 % a specificity 67–81 % při LoD 0,1 % [29,50]. V naší srovnávací studii bylo pro detekci T790M dosaženo LoD 0,5 % v 100 % (6/6) laboratoří používajících tuto metodu. Pro samotné senzitivní mutace dosáhlo 0,5% LoD 83,3 % (5/6) laboratoří, pro del 19 byl v 100 % (6/6) pracovišť LoD min. 0,05 %. Ukázalo se, že analytická senzitivita metodiky cobas® v rámci naší studie je konzistentní se senzitivitou udávanou výrobcem (0,75 % pro del 19 a 1 % pro mutace L858R a T790M) [54]. Pro kit Super-ARMS® EGFR Mutation Detection Kit je analytická senzitivita udaná výrobcem 0,2 % pro deleci E746_A750 v exonu 19, pro mutaci L858R a rovněž pro mutaci T790M. Této citlivosti nebylo ve studii dosaženo v jednom případě, a to pro mutaci T790M. Mutace L858R a del 19 byly zachyceny ve všech případech, tj. i ve vzorcích s frekvencí mutantní alely 0,05 %.
Graph 1. Přehled výsledků genotypizace v závislosti na typu a frekvenci detekované mutace ve vzorku plazmy. 
del 19 – delece v exonu 19 genu EGFR Poměrně vysoké procento chybovosti této studie oproti jiným běžně prováděným molekulárním testům je možné vysvětlit použitím metod s vyšším limitem detekce, než byla nejnižší vyšetřovaná alelická frekvence 0,05 %. Obzvláště pro vyšetřování ctDNA z plazmy s velice nízkou frakcí (až 0,01 %) je k získání spolehlivých výsledků testování nutné použití vysoce senzitivních molekulárních metod. Optimální senzitivita pro detekci ctDNA v současné době však není zřejmá. Pro použití v klinické praxi je rovněž třeba vyřešit standardizaci preanalytické fáze, zejména odběr vzorku periferní krve (vč. typu odběrové zkumavky) a jeho zpracování, především dodržení časových limitů mezi odběrem, oddělením plazmy a izolací nukleových kyselin, jejíž řešení však přesahuje rámec této publikace.
Z praktického hlediska z proběhlých studií i naší práce vyplývá, že standardně používané metodiky detekce EGFR ctDNA jsou schopné detekovat nejčastější senzitivní mutace (zejména del 19) s vysokou spolehlivostí, a být tak podkladem pro léčbu EGFR-TKI. V případě detekce sekundárně vzniklé rezistentní mutace T790M v ctDNA je výsledek možné brát jako východisko pro indikaci léčby TKI 3. generace. Obdobná léčebná odpověď u pacientů s touto mutací v plazmě jako v primárním nádoru byla prokázána v rámci několika klinických studií [29,50]. Díky tekuté biopsii by se tak s ohledem na frekvenci mutace T790M a na senzitivitu vyšetření ctDNA mohlo vyhnout bioptickému či cytologickému odběru více než 50 % pacientů s rezistencí na léčbu TKI [55]. Detekce ctDNA může být negativně ovlivněna nízkou nádorovou náloží, blokádou cytotoxickými léky či preanalytickými a analytickými faktory, především výběrem metody kvantifikace a analýzy ctDNA [56]. V případě negativního výsledku je proto všeobecně doporučována maximální snaha o získání materiálu z nádorového ložiska (schéma 1).
Schéma 1. Algoritmus vyšetřování sekundární rezistentní mutace T790M genu EGFR z plazmy. Genotypizace mutace T790M z plazmy při vzniku rezistence na léčbu EGFR-TKI u EGFR pozitivních pacientů je možná před bioptickým odběrem. K vyšetření T790M z nádorové tkáně získané rebiopsií je možné přistoupit až při negativním molekulárním výsledku z plazmy. Modifi kováno dle Oxnard et al [55]. ![Schéma 1. Algoritmus vyšetřování sekundární rezistentní mutace T790M genu <i>EGFR</i> z plazmy.
Genotypizace mutace T790M z plazmy při vzniku rezistence na léčbu <i>EGFR</i>-TKI u <i>EGFR</i> pozitivních pacientů je možná před bioptickým odběrem.
K vyšetření T790M z nádorové tkáně získané rebiopsií je možné přistoupit až při negativním molekulárním výsledku z plazmy. Modifi kováno
dle Oxnard et al [55].](https://pl-master.mdcdn.cz/media/image_pdf/3d25f09a5936feabd38d62a4cf1dc907.jpeg?version=1539607390)
TKI – inhibitor tyrozinkinázového receptoru Závěr
Tekutá biopsie se stává realitou každodenní klinické praxe. Testování mutačního stavu EGFR z ctDNA z plazmy má potenciál získat si své trvalé místo v rutinní diagnostice a monitorování léčby pacientů s NSCLC, zejména vezmeme-li v potaz, že extrakci cfDNA a stanovení mutačního stavu je možné provést v průběhu jednoho pracovního dne. Díky vývoji nových vysoce senzitivních metod a jejich zavedení do praxe představuje detekce ctDNA z plazmy vhodnou metodu v případech nedostatečného množství tkáně k molekulárnímu testování nebo u pacientů, kde není možné bioptický odběr provést či vyšetření provádět opakovaně. Musíme si nicméně uvědomit, že tekutá biopsie není pro detekci EGFR statusu konkurenční metodou k odběru bioptickému či cytologickému, ale jeho cennou alternativou. Eseje užívané k typizaci EGFR ctDNA z plazmy prokázaly ve většině publikovaných studií relativně vysokou specificitu a pozitivní prediktivní hodnotu, avšak limitovanou senzitivitu, zejména pro mutaci T790M. Rovněž naše výsledky vzhledem k nižší citlivosti detekce rezistentní mutace T790M potvrzují nutnost pokusu o rebiopsii u pacientů s negativním výsledkem, neboť zde oproti vyšetření solidního vzorku tkáně stoupá podíl falešně negativních případů. Jedině národní či mezinárodní schémata kontroly kvality jsou schopná vést k posouzení, udržení, popřípadě zlepšení úrovně molekulárního vyšetřování EGFR z ctDNA z plazmy, aby se mohlo stát robustním opěrným bodem pro terapeutické rozhodování.
Poděkování
Děkujeme pracovníkům všech zúčastněných labora-toří a prof. MUDr. A. Ryškovi, Ph.D., za iniciaci této studie.
Práce byla realizována za podpory firmy Astra - Zeneca a podpořena projektem (Ministerstva zdravotnictví) koncepčního rozvoje výzkumné organizace 00064203 (FN MOTOL).
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do biomedicínských časopisů.
MUDr. Linda Čapková, Ph.D.
Ústav patologie a molekulární medicíny 2. LF UK a FN
Motol V Úvalu 84 150 06 Praha 5
e-mail: linda.capkova@fnmotol.cz
Obdrženo: 4. 6. 2018
Přijato: 1. 8. 2018
Sources
1. Miller KD, Siegel RL, Lin CC et al. Cancer treatment and survivorship statistics, 2016. CA Cancer J Clin 2016; 66 (4): 271–289. doi: 10.3322/caac.21349.
2. Herbst RS, Heymach JV, Lippman SM. Lung cancer. N Engl J Med 2008; 359 (13): 1367–1380. doi: 10.1056/NEJMra0802714.
3. Rosell R, Moran T, Queralt C et al. Screening for epidermal growth factor receptor mutations in lung cancer. N Engl J Med 2009; 361 (10): 958–967. doi: 10.1056/NEJMoa0904554.
4. Fiala O, Pešek M, Fínek J et al. EGFR mutations in patients with advanced NSCLC. Klin Onkol 2012; 25 (4): 267–273. doi: 10.14735/amko2012267.
5. Reck M, Popat S, Reinmuth N et al. Metastatic non-small-cell lung cancer (NSCLC): ESMO clinical practice guidelines for diagnosis, treatment and follow-up. Ann Oncol 2014; 25 (Suppl 3): iii27 – iii 39. doi: 10.1093/annonc/mdu199.
6. Sharma SV, Bell DW, Settleman J et al. Epidermal growth factor receptor mutations in lung cancer. Nat Rev Cancer 2007; 7 (3): 169–181. doi: 10.1038/nrc2088.
7. Heneberg P. Indication of EGFR kinase inhibitors should be refined. Klin Onkol 2011; 24 (2): 87–93. doi: 10.14735/amko201187.
8. Kim ES, Hirsh V, Mok T et al. Gefitinib versus docetaxel in previously treated non-small-cell lung cancer (INTEREST): a randomised phase III trial. Lancet 2008; 372 (9652): 1809–1818. doi: 10.1016/S0140-6736 (08) 61758-4.
9. Inoue A, Sugawara S, Yamazaki K et al. Randomized phase II trial comparing amrubicin with topotecan in patients with previously treated small-cell lung cancer: north Japan lung cancer study group trial 0402. J Clin Oncol 2008; 26 (33): 5401–5406. doi: 10.1200/JCO.2008.18.1974.
10. Mok TS, Wu YL, Thongprasert S et al. Gefitinib or carboplatin-paclitaxel in pulmonary adenocarcinoma. N Engl J Med 2009; 361 (10): 947–957. doi: 10.1056/NEJMoa0810699.
11. Rosell R, Carcereny E, Gervais R et al. Erlotinib versus standard chemotherapy as first-line treatment for European patients with advanced EGFR mutation-positive non-small-cell lung cancer (EURTAC): a multicentre, open-label, randomised phase 3 trial. Lancet Oncol 2012; 13 (3): 239–246. doi: 10.1016/S1470-2045 (11) 70393-X.
12. Sequist LV, Yang JC, Yamamoto N et al. Phase III study of afatinib or cisplatin plus pemetrexed in patients with metastatic lung adenocarcinoma with EGFR mutations. J Clin Oncol 2013; 31 (27): 3327–3334. doi: 10.1200/JCO.2012.44.2806.
13. Sequist LV, Waltman BA, Dias-Santagata D et al. Genotypic and histological evolution of lung cancers acquiring resistance to EGFR inhibitors. Sci Transl Med 2011; 3 (75): 75ra26. doi: 10.1126/scitranslmed.3002003.
14. Yu HA, Arcila ME, Rekhtman N et al. Analysis of tumor specimens at the time of acquired resistance to EGFR-TKI therapy in 155 patients with EGFR-mutant lung cancers. Clin Cancer Res 2013; 19 (8): 2240–2247. doi: 10.1158/1078-0432.CCR-12-2246.
15. Cross DA, Ashton SE, Ghiorghiu S et al. AZD9291, an irreversible EGFR TKI, overcomes T790M-mediated resistance to EGFR inhibitors in lung cancer. Cancer Discov 2014; 4 (9): 1046–1061. doi: 10.1158/2159-8290.CD-14-0337.
16. Mok TS, Wu YL, Ahn MJ et al. Osimertinib or platinum-pemetrexed in EGFR T790M-positive lung cancer. N Engl J Med 2017; 376 (7): 629–640. doi: 10.1056/NEJMoa1612674.
17. Yoon HJ, Lee HY, Lee KS et al. Repeat biopsy for mutational analysis of non-small cell lung cancers resistant to previous chemotherapy: adequacy and complications. Radiology 2012; 265 (3): 939–948. doi: 10.1148/radiol.12112613.
18. Yang JC, Wu YL, Chan V et al. Epidermal growth factor receptor mutation analysis in previously unanalyzed histology samples and cytology samples from the phase III Iressa Pan-ASia Study (IPASS). Lung Cancer 2014; 83 (2): 174–181. doi: 10.1016/j.lungcan.2013.11.021.
19. Pirker R, Pereira JR, von Pawel J et al. EGFR expression as a predictor of survival for first-line chemotherapy plus cetuximab in patients with advanced non-small-cell lung cancer: analysis of data from the phase 3 FLEX study. Lancet Oncol 2012; 13 (1): 33–42. doi: 10.1016/S1470-2045 (11) 70318-7.
20. de Bruin EC, McGranahan N, Mitter R et al. Spatial and temporal diversity in genomic instability processes defines lung cancer evolution. Science 2014; 346 (6206): 251–256. doi: 10.1126/science.1253462.
21. Fisher R, Pusztai L, Swanton C. Cancer heterogeneity: implications for targeted therapeutics. Br J Cancer 2013; 108 (3): 479–485. doi: 10.1038/bjc.2012.581.
22. Yung TK, Chan KC, Mok TS et al. Single-molecule detection of epidermal growth factor receptor mutations in plasma by microfluidics digital PCR in non-small cell lung cancer patients. Clin Cancer Res 2009; 15 (6): 2076–2084. doi: 10.1158/1078-0432.CCR-08-2622.
23. Brevet M, Johnson ML, Azzoli CG et al. Detection of EGFR mutations in plasma DNA from lung cancer patients by mass spectrometry genotyping is predictive of tumor EGFR status and response to EGFR inhibitors. Lung Cancer 2011; 73 (1): 96–102. doi: 10.1016/j.lungcan.2010.10.014.
24. Jahr S, Hentze H, Englisch S et al. DNA fragments in the blood plasma of cancer patients: quantitations and evidence for their origin from apoptotic and necrotic cells. Cancer Res 2001; 61 (4): 1659–1665.
25. Thierry AR, El Messaoudi S, Gahan PB et al. Origins, structures, and functions of circulating DNA in oncology. Cancer Metastasis Rev 2016; 35 (3): 347–376. doi: 10.1007/s10555-016-9629-x.
26. Chan KC. Scanning for cancer genomic changes in plasma: toward an era of personalized blood-based tumor markers. Clin Chem 2013; 59 (11): 1553–1555. doi: 10.1373/clinchem.2013.207381.
27. Murtaza M, Dawson SJ, Tsui DW et al. Non-invasive analysis of acquired resistance to cancer therapy by sequencing of plasma DNA. Nature 2013; 497 (7447): 108–112. doi: 10.1038/nature12065.
28. Nakamura T, Sueoka-Aragane N, Iwanaga K et al. A noninvasive system for monitoring resistance to epidermal growth factor receptor tyrosine kinase inhibitors with plasma DNA. J Thorac Oncol 2011; 6 (10): 1639–1648. doi: 10.1097/JTO.0b013e31822956e8.
29. Thress KS, Brant R, Carr TH et al. EGFR mutation detection in ctDNA from NSCLC patient plasma: A cross-platform comparison of leading technologies to support the clinical development of AZD9291. Lung Cancer 2015; 90 (3): 509–515. doi: 10.1016/j.lungcan.2015.10.004.
30. Sakai K, Horiike A, Irwin DL et al. Detection of epidermal growth factor receptor T790M mutation in plasma DNA from patients refractory to epidermal growth factor receptor tyrosine kinase inhibitor. Cancer Sci 2013; 104 (9): 1198–1204. doi: 10.1111/cas.12211.
31. Oxnard GR, Paweletz CP, Kuang Y et al. Noninvasive detection of response and resistance in EGFR-mutant lung cancer using quantitative next-generation genotyping of cell-free plasma DNA. Clin Cancer Res 2014; 20 (6): 1698–1705. doi: 10.1158/1078-0432.CCR-13-2482.
32. Douillard JY, Ostoros G, Cobo M et al. Gefitinib treatment in EGFR mutated caucasian NSCLC: circulating-free tumor DNA as a surrogate for determination of EGFR status. J Thorac Oncol 2014; 9 (9): 1345–1353. doi: 10.1097/JTO.0000000000000263.
33. Reck M, Hagiwara K, Han B et al. ctDNA determination of EGFR mutation status in European and Japanese patients with advanced NSCLC: the ASSESS study. J Thorac Oncol 2016; 11 (10): 1682–1689. doi: 10.1016/j.jtho.2016.05.036.
34. Lindeman NI, Cagle PT, Aisner DL et al. Updated molecular testing guideline for the selection of lung cancer patients for treatment with targeted tyrosine kinase inhibitors: guideline from the College of American Pathologists, the International Association for the Study of Lung Cancer, and the Association for Molecular Pathology. J Mol Diagn 2018; 20 (2): 129–159. doi: 10.1016/j.jmoldx.2017.11.004.
35. Diehl F, Schmidt K, Choti MA et al. Circulating mutant DNA to assess tumor dynamics. Nat Med 2008; 14 (9): 985–990. doi: 10.1038/nm.1789.
36. Kukita Y, Uchida J, Oba S et al. Quantitative identification of mutant alleles derived from lung cancer in plasma cell-free DNA via anomaly detection using deep sequencing data. PLoS One 2013; 8 (11): e81468. doi: 10.1371/journal.pone.0081468.
37. Sacher AG, Komatsubara KM, Oxnard GR. Application of plasma genotyping technologies in non-small cell lung cancer: a practical review. J Thorac Oncol 2017; 12 (9): 1344–1356. doi: 10.1016/j.jtho.2017.05.022.
38. Suzawa K, Yamamoto H, Ohashi K et al. Optimal method for quantitative detection of plasma EGFR T790M mutation using droplet digital PCR system. Oncol Rep 2017; 37 (5): 3100–3106. doi: 10.3892/or.2017. 5567.
39. Bartels S, Persing S, Hasemeier B et al. Molecular analysis of circulating cell-free DNA from lung cancer patients in routine laboratory practice: a cross-platform comparison of three different molecular methods for mutation detection. J Mol Diagn 2017; 19 (5): 722–732. doi: 10.1016/j.jmoldx.2017.05.008.
40. Kasahara N, Kenmotsu H, Serizawa M et al. Plasma epidermal growth factor receptor mutation testing with a chip-based digital PCR system in patients with advanced non-small cell lung cancer. Lung Cancer 2017; 106 : 138–144. doi: 10.1016/j.lungcan.2017.02.001.
41. Reckamp KL, Melnikova VO, Karlovich C et al. A highly sensitive and quantitative test platform for detection of NSCLC EGFR mutations in urine and plasma. J Thorac Oncol 2016; 11 (10): 1690–1700. doi: 10.1016/j.jtho.2016.05.035.
42. Yoshida H, Kim YH, Ozasa H et al. EGFR T790M detection in circulating tumor DNA from non-small cell lung cancer patients using the PNA-LNA clamp method. Anticancer Res 2017; 37 (5): 2721–2725. doi: 10.21873/anticanres.11623.
43. Jenkins S, Yang JC, Ramalingam SS et al. Plasma ctDNA analysis for detection of the EGFR T790M mutation in patients with advanced non-small cell lung cancer. J Thorac Oncol 2017; 12 (7): 1061–1070. doi: 10.1016/j.jtho.2017.04.003.
44. Summary of safety and effectiveness data (SSED). FDA: U.S. Food and Drug Administration. [online]. Available from: https: //www.accessdata.fda.gov/cdrh_docs/pdf15/p150044b.pdf.
45. QIAGEN announces first-ever regulatory registration of a lung cancer companion diagnostic based on liquid biopsies. PR Newswire. [online]. Available from: https: //www.prnewswire.com/news-releases/qiagen-announces-first-ever-regulatory-registration-of-a-lung-cancer-companion-diagnostic-based-on-liquid-biopsies-288226911.html.
46. Mok T, Wu YL, Lee JS et al. Detection and dynamic changes of EGFR mutations from circulating tumor DNA as a predictor of survival outcomes in NSCLC patients treated with first-line intercalated erlotinib and chemothera-py. Clin Cancer Res 2015; 21 (14): 3196–3203. doi: 10.1158/1078-0432.CCR-14-2594.
47. List of cleared or approved companion diagnostic devices (in vitro and imaging tools). FDA: U.S. Food and Drug Administration. [online]. Available from: https: //www.fda.gov/medicaldevices/productsandmedicalprocedures/invitrodiagnostics/ucm301431.htm.
48. McCoach CE, Jimeno A. Osimertinib, a third-generation tyrosine kinase inhibitor targeting non-small cell lung cancer with EGFR T790M mutations. Drugs Today (Barc) 2016; 52 (10): 561–568. doi: 10.1358/dot. 2016.52.10. 2541343.
49. Weber B, Meldgaard P, Hager H et al. Detection of EGFR mutations in plasma and biopsies from non-small cell lung cancer patients by allele-specific PCR assays. BMC Cancer 2014; 14 : 294. doi: 10.1186/1471-2407-14-294.
50. Karlovich C, Goldman JW, Sun JM et al. Assessment of EGFR mutation status in matched plasma and tumor tissue of NSCLC patients from a phase i study of rociletinib (CO-1686). Clin Cancer Res 2016; 22 (10): 2386–2395. doi: 10.1158/1078-0432.CCR-15-1260.
51. Deans ZC, Williams H, Dequeker EMC et al. Review of the implementation of plasma ctDNA testing on behalf of IQN Path ASBL: a perspective from an EQA providers‘ survey. Virchows Arch 2017; 471 (6): 809–813. doi: 10.1007/s00428-017-2222-z.
52. AIOM.it. Associazione Italiana di oncologia medica. [online]. Available from: www.aiom.it/aiom-servizi/eventi_aiom_servizi/6th_meeting_on-external-quality-assessment-in-molecular-pathology/1,4046,1.
53. Molecular testing of cfDNA in plasma for EGFR gene mutations (pilot). European Molecular Quality Network. [online]. Available from: https: //www.emqn.org/schemes/molecular-testing-EGFR-gene-mutations-ctdna-pilot/.
54. Cobas® EGFR mutation test v2. FDA: U.S. Food and Drug Administration. [online]. Available from: www.accessdata.fda.gov/cdrh_docs/pdf12/P120019s007c.pdf.
55. Oxnard GR, Thress KS, Alden RS et al. Association between plasma genotyping and outcomes of treatment with osimertinib (AZD9291) in advanced non-small-cell lung cancer. J Clin Oncol 2016; 34 (28): 3375–3382. doi: 10.1200/JCO.2016.66.7162.
56. Heitzer E, Ulz P, Geigl JB. Circulating tumor DNA as a liquid biopsy for cancer. Clin Chem 2015; 61 (1): 112–123. doi: 10.1373/clinchem.2014.222679.
57. Catalogue of somatic mutations in cancer. [online]. Available from: http: //cancer.sanger.ac.uk/cancergenome/projects/cosmic/.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2018 Issue 5-
All articles in this issue
- Gravidita a ovariální stimulace u pacientek s karcinomem prsu
- Význam mutace BRAFV600E v tyreoidální onkologii a možnosti léčebného ovlivnění – aktuální poznatky a výsledky studií
- Psychoneuroimunológia nádorových chorôb – súčasné východiská a perspektívy
- Detekce EGFR mutací v cirkulující nádorové DNA (ctDNA) v plazmě – mezilaboratorní porovnání referenčních laboratoří v České republice
- Zhodnocení výskytu a charakteru neurologických/neuropsychiatrických komplikací v souboru pacientů s maligním melanomem léčených adjuvantně vysokodávkovaným interferonem
- Pituitárna metastáza u pacienta s pľúcnym adenokarcinómom prezentujúca sa poruchou vedomia
- Solidní pseudopapilární tumor pankreatu – vzácné onemocnění 20leté ženy
- Afatinib v léčbě pokročilého nemalobuněčného karcinomu plic se vzácnou mutací EGFR (v exonu 18-T719X) – kazuistika
- Metaanalýza polymorfizmů v BRCA1 a náchylnost k nádorům prsu
- Metastáza nádoru do nádoru – raritný prípad svetlobunkového renálneho karcinómu obsahujúceho metastázu adenokarcinómu neznámeho pôvodu
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Solidní pseudopapilární tumor pankreatu – vzácné onemocnění 20leté ženy
- Psychoneuroimunológia nádorových chorôb – súčasné východiská a perspektívy
- Význam mutace BRAFV600E v tyreoidální onkologii a možnosti léčebného ovlivnění – aktuální poznatky a výsledky studií
- Gravidita a ovariální stimulace u pacientek s karcinomem prsu
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

