-
Články
- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Detailní molekulární charakterizace izolátů Neisseria meningitidis metodou sekvenace celého genomu (WGS), Česká republika, 2010–2019
Detailed molecular characterization of Neisseria meningitidis isolates by whole genome sequencing (WGS), Czech Republic, 2010–2019
Aim: Presentation of the first results of the analysis of Neisseria meningitidis isolates from invasive meningococcal disease and from clinically and/or epidemiologically linked cases from 2010–2019. Whole genome sequencing (WGS) was used for the study.
Material and methods: The study set included 59 isolates of N. meningitidis from 2010–2019. Serogrouping was done by conventional serological methods and confirmed by RT-PCR. WGS was used for detailed molecular characterization, covering not only basic genes but also ribosomal and capsular genes, antibiotic resistance gene penA, and outer membrane protein gene porA.
Results: WGS analysis of N. meningitidis isolates resulted in a detailed molecular characterization. In a large part of the genes analysed, new mutated allelic variants were found. They were submitted to the PubMLST database and subsequently annotated by the curator. All 59 study isolates were assigned to BAST types, characterized by a unique combination of allelic variants of N. meningitidis B vaccine (MenB vaccine) antigen genes. Overall, 32 different BAST types were identified, and 10 isolates either carried an unknown combination of BAST loci or a new allelic variant in some of the BAST loci. Furthermore, the MenDeVAR index, which provides information on the functional effect of MenB vaccines on a given isolate, was determined.
Conclusions: The results obtained add to the body of knowledge of the transmission of invasive and non-invasive strains of N. meningitidis in the population. The WGS analysis provided detailed data on the coverage of these strains by new MenB vaccines.
Keywords:
Neisseria meningitidis – whole genome sequencing (WGS)
Autoři: M. Honskus
; Z. Okonji
; M. Musílek
; P. Křížová
Působiště autorů: Národní referenční laboratoř pro meningokokové nákazy, Centrum epidemiologie a mikrobiologie, Státní zdravotní ústav, Praha
Vyšlo v časopise: Epidemiol. Mikrobiol. Imunol. 70, 2021, č. 3, s. 168-177
Kategorie: Původní práce
Souhrn
Cíl: Prezentace prvních výsledků analýzy souboru izolátů Neisseria meningitidis z invazivního meningokokového onemocnění a izolátů spjatých s nimi klinicky a/nebo epidemiologicky z období 2010–2019. Ke studiu byla použita metoda sekvenace celého genomu (WGS).
Materiál a metody: Studovaný soubor tvořilo celkem 59 izolátů N. meningitidis z období 2010–2019. Séroskupiny byly určeny klasickými sérologickými metodami a ověřeny metodou RT-PCR. Metodou WGS byla provedena detailní molekulární charakterizace, která kromě genů základní molekulární charakterizace zahrnuje i analýzu ribozomálních a kapsulárních genů, genu antibiotické rezistence penA a genu porA, který kóduje protein zevní buněčné membrány.
Výsledky: Studium izolátů N. meningitidis metodou WGS poskytlo detailní molekulární charakterizaci. U velké části studovaných genů byly zjištěny nové, mutované alelové varianty, které byly registrovány v databázi PubMLST a následně anotovány kurátorem. U všech 59 studovaných izolátů byl určen BAST typ, což je kombinace alelových variant genů antigenů vakcín proti onemocněním vyvolaným N. meningitidis B (MenB vakcín). Celkem bylo zjištěno 32 různých BAST typů, z toho 10 izolátů obsahovalo buď neznámou kombinaci BAST lokusů, nebo neslo novou alelovou variantu v některém z nich. Dále byl u studovaných izolátů určen index MenDeVAR, který poskytuje informace o funkčním účinku MenB vakcín na daný izolát.
Závěry: Získané výsledky prohlubují znalosti o přenosu invazivních a neinvazivních kmenů N. meningitidis v populaci. Metodou WGS byla získána detailní data o pokrytí těchto kmenů novými MenB vakcínami.
Klíčová slova:
Neisseria meningitidis – sekvenace celého genomu (WGS)
ÚVOD
Invazivní meningokokové onemocnění (IMO) patří mezi infekční onemocnění, které i přes pokrok moderní terapie má stále vysokou smrtnost a způsobuje závažné celoživotní následky až u 20 % přeživších. Nicméně až u 10 % osob ve zdravé populaci je v horních cestách dýchacích meningokok přítomen, aniž by působil jakékoli klinické projevy – hovoříme o zdravých nosičích Neisseria meningitidis. Faktory, které rozhodují o tom, jak se setkání člověka s meningokokem vyvine, jsou na straně hostitele i bakterie.
N. meningitidis je výlučně lidský patogen, který se přenáší vzdušnou cestou. Patří mezi jedny z nejvariabilnějších bakterií a jeho chování v organismu je odrazem jeho genetické informace [1]. Genom bakterie N. meningitidis je vysoce plastický a často zde dochází k různým spontánním mutacím, jako jsou delece, inzerce nebo substituce [2, 3]. Kromě toho byla u meningokoků prokázána i možnost horizontálního přenosu genů.
V současné době jsou k dispozici dvě vakcíny proti onemocněním vyvolaným N. meningitidis B (MenB vakcíny): 4komponentní vakcína (Bexsero) a 2složková vakcína (Trumenba). Vakcína Bexsero obsahuje peptidové produkty tří genů: fHbp, nhba, nadA a část proteinu zevní buněčné membrány porA. Vakcína Trumenba obsahuje peptidové produkty dvou různých variant genu fHbp [4].
Nejmodernější metodou, která poskytuje široké možnosti ke studiu vlastností N. meningitidis, je sekvenování celého genomu (WGS). V novém projektu studujeme metodou WGS izoláty od pacientů s IMO a od zdravých nosičů N. meningitidis s cílem zlepšit poznatky o faktorech virulence N. meningitidis, prohloubit znalosti o úloze nosičství N. meningitidis při šíření IMO, porozumět přenosu invazivních a neinvazivních kmenů a posoudit možnost pokrytí těchto kmenů novými MenB vakcínami.
Prezentujeme zde první výsledky studia metodou WGS u souboru 59 izolátů N. meningitidis z IMO a izolátů spjatých s nimi klinicky a/nebo epidemiologicky z období 2010–2019. Obsahem je detailní molekulární charakterizace, která kromě genů základní molekulární charakterizace zahrnuje i analýzu ribozomálních a kapsulárních genů, genu antibiotické rezistence penA a genu porA, který kóduje protein zevní buněčné membrány. Získaná WGS data jsou uložena v mezinárodní databázi PubMLST [5] a jsou veřejně dostupná.
MATERIÁL A METODY
Izoláty N. meningitidis
Pro studium metodou WGS byly vybrány párové izoláty od pacientů s IMO a jejich zdravých kontaktů a párové izoláty z různého klinického materiálu od pacientů s IMO. Studovaný soubor tvořilo celkem 59 izolátů N. meningitidis z období 2010–2019 (33 izolátů z IMO, 26 izolátů od jejich zdravých kontaktů). Výběr izolátů byl proveden na základě epidemiologických a klinických informací o bakteriálních kulturách v databázi NRL, která obsahuje i data získaná klasickou sekvenací [6, 7, 8].
Klasická charakterizace N. meningitidis
Příslušné bakteriální kultury N. meningitidis, které jsou uchovávány ve sbírce NRL pro meningokokové nákazy, byly vyočkovány na čokoládový Mueller-Hinton agar a kultivovány 18–24 hodin ve 37 oC v 5% CO2 atmosféře. Séroskupiny byly určeny klasickými sérologickými metodami a ověřeny molekulární metodou RT-PCR [9].
Celogenomová sekvenace N. meningitidis
K izolaci deoxyribonukleové kyseliny (DNA) byla použita souprava QIAamp DNA Mini Kit (QIAGEN) a postup izolace probíhal podle pokynů výrobce [10]. DNA byla následně odeslána k celogenomové sekvenaci na externí pracoviště EMBL (European Molecular Biology Laboratory, Heidelberg, Německo). Sekvenace proběhla na platformě Illumina MiSeq s využitím referenčního genomu N. meningitidis kmen MC58. K sestavení výsledných genomů z primárních celogenomových raw dat byl na našem pracovišti použit software Velvet de novo Assembler (assembly de novo) [11]. Genomy izolátů byly poté registrovány v databázi PubMLST, která využívá platformu BIGSdb (Bacterial Isolate Genome Sequence Database) [5, 12].
VÝSLEDKY A DISKUSE
Studie prezentuje první výsledky analýz molekulárních charakteristik N. meningitidis metodou WGS. V další publikaci bude provedeno srovnání rozdílů mezi izoláty od pacientů s IMO a od jejich zdravých kontaktů.
1. Určení séroskupin a genoskupin
Klasickými metodami byla u jednotlivých izolátů zjištěna příslušnost k séroskupinám: A (1 izolát), B (23 izolátů), C (14 izolátů), W (8 izolátů) a Y (4 izoláty). U 9 izolátů nebylo možné séroskupinu určit (N. meningitidis NG, non-groupable).
Za produkci kapsulárního polysacharidu a jeho transport na buněčný povrch odpovídají geny kapsulárních regionů A, B a C. Region A obsahuje geny, které kódují enzymy pro vlastní biosyntézu kapsulárního polysacharidu a geny regionů B a C jsou nezbytné pro aktivní translokaci syntetizovaného polysacharidu na buněčný povrch. V případě genoskupiny A je kapsulární region A tvořen čtyřmi specifickými geny (csaA, csaB, csaC a csaD), které se u jiných genoskupin nevyskytují. U genoskupin B, C, Y a W je region A tvořen konzervovanými geny cssA, cssB, cssC, ctrG a genem pro séroskupinově specifickou kapsulární polymerázu (csb, csc, csy, csw). U genoskupin C, Y a W se v kapsulárním regionu A navíc vyskytuje gen pro O-acetyltransferázu – cssE (genoskupina C) nebo cssF (genoskupiny Y a W) [13].
Zatímco metodou sklíčkové aglutinace jsou séroskupiny určeny u 91 % izolátů a metodou RT-PCR u 99 %, metoda WGS poskytuje nejpřesnější genetické podklady pro správné určení séroskupiny N. meningitidis [14]. Metodou WGS byla provedena analýza genů kapsulárního regionu A, tj. zařazení izolátu do genoskupiny, která by měla korespondovat s určenou séroskupinou. Analýzu genů kapsulárního regionu A shrnuje tabulka 1.
Tab. 1. Analýza genů kapsulárního regionu A u 59 izolátů N. meningitidis, 2010–2019, Česká republika
Table 1. Analysis of capsular region A genes in 59 N. meningitidis isolates, 2010–2019, Czech Republic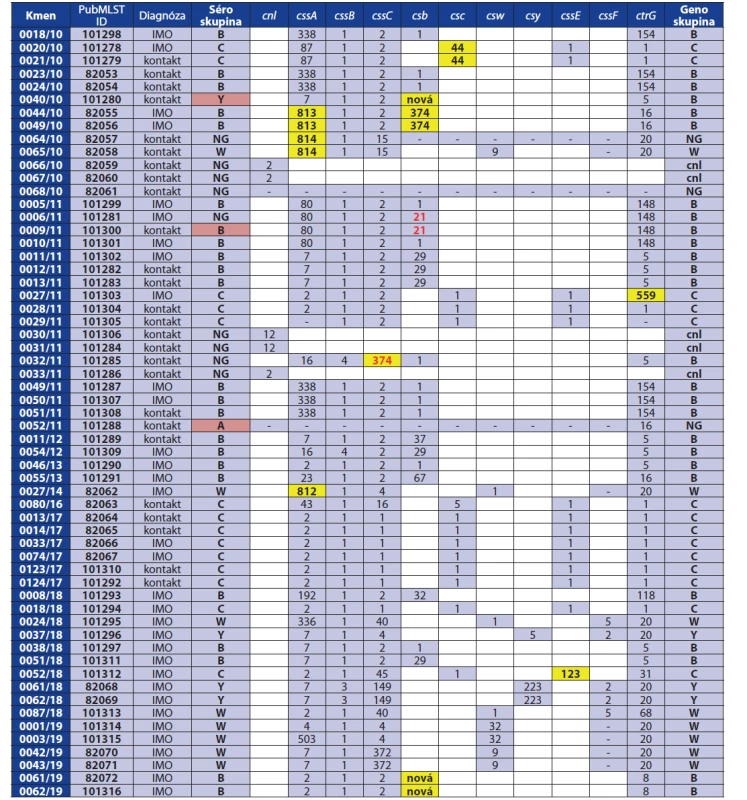
IMO = invazivní meningokokové onemocnění
NG (non-groupable) = izolát, u kterého nebylo možné určit séroskupinu
červené písmo = alela, která je v PubMLST databázi definována jako inaktivní
žluté pole = nově popsaná alelová varianta/nová alela, která je ve fázi registrace v databázi PubMLST
červené pole = určená séroskupina neodpovídá genetické výbavě izolátu
bílé pole = gen, který se v kapsulárním regionu A příslušné genoskupiny nevyskytuje
IMD = invasive meningococcal disease
NG (non-groupable) = serogroup could not be determined
Writing in red = allele defined as inactive in the PubMLST database
Yellow box = newly described allelic variant /new allele at the stage of registration in the PubMLST database
Red box = serogroup inconsistent with the genetic makeup of the isolate
White box = gene not found in capsular region A of the respective genogroupU izolátu 0052/11 séroskupiny A analýza neodhalila žádnou alelovou variantu genu příslušné genoskupiny A, ale prokázala, že izolát nesl popsanou aktivní alelu 16 genu ctrG, který ve svém genetickém profilu sdílí čtyři různé genoskupiny (B, C, W a Y). Žádné další popsané alely, nově mutované alely ani fragmenty genových sekvencí, které by umožnily blíže zařadit tento izolát, nebyly nalezeny.
Z 23 izolátů séroskupiny B byla u 19 zjištěna v kapsulárním regionu A kompletní sada genů genoskupiny B (cssA, cssB, cssC, csb, ctrG) s již popsanými alelami. V 18 případech se jednalo o alely aktivní. Jeden izolát (0009/11) obsahoval u genu kapsulární polymerázy (csb) alelovou variantu 21, která je inaktivována fázovou variací a tento izolát by tedy neměl být schopen produkovat kapsulární polysacharid. Zbylé 4 izoláty nesly rovněž kapsulární geny odpovídající séroskupině B, nicméně část z nich byla v mutovaných, nově popsaných nebo dosud nepopsaných alelových variantách (geny csb a cssA). Vzhledem k výskytu nových alel nelze zatím u těchto izolátů zhodnotit schopnost produkovat kapsulární polysacharid.
Ze 14 izolátů séroskupiny C devět obsahovalo kompletní sadu kapsulárních genů genoskupiny C (cssA, cssB, cssC, csc, cssE, ctrG) s popsanými a aktivními alelami. Dva izoláty (0020/10 a 0021/10), které byly epidemiologicky příbuzné, nesly identicky mutovanou alelu 44 genu kapsulární polymerázy séroskupiny C (csc), která se od nejbližší známé alely (9) lišila jednou bodovou mutací a jednonukleotidovou delecí. Izolát 0027/11 obsahoval mutovanou alelu genu ctrG - 559 (dvě bodové mutace alely 453) a izolát 0052/18 nesl mutovanou alelu genu cssE - 123 (jedna bodová mutace alely 1). U izolátu 0029/11 byly nalezeny pouze alely genů cssB, cssC, csc a cssE.
Všech 8 izolátů séroskupiny W neslo v kapsulárním regionu A odpovídající geny (cssA, cssB, cssC, csw, cssF, ctrG). Ve dvou případech se jednalo o kompletní sadu popsaných alel a dva izoláty (0065/10 a 0027/14) obsahovaly nově popsané alely genu cssA (812 a 814). Šest izolátů postrádalo kompletní alelu genu cssF, což odpovídá údajům v databázi PubMLST o výskytu aktivní alely tohoto genu u izolátů genoskupiny W.
Ze 4 izolátů séroskupiny Y byla u 3 prokázána kompletní sestava kapsulárních genů genoskupiny Y (cssA, cssB, cssC, csy, cssF, ctrG) v popsaných a aktivních alelových variantách. Izolát 0040/10, zařazený k séroskupině Y, nesl známé alely pouze u genů cssA, cssB, cssC a ctrG, které se vyskytují u genoskupin B, C, W a Y. Detailnější analýzou se nám následně podařilo prokázat přítomnost značně mutované sekvence kapsulární polymerázy séroskupiny B (csb), která se liší od nejbližší popsané alely [37] především jednonukleotidovou delecí na pozici 95, která způsobuje posun čtecího rámce, a činí tak alelu nefunkční.
U 9 izolátů nezařazených ke konkrétní séroskupině (NG) jsme se setkali s různými příčinami této skutečnosti. Pět izolátů neslo alelu lokusu cnl (capsule null). Tento lokus vylučuje přítomnost jiných kapsulárních genů a způsobuje tak, že bakterie není schopna produkovat žádný kapsulární polysacharid na svůj povrch. Izolát 0006/11 obsahoval kompletní sadu kapsulárních genů genoskupiny B, nicméně alela 21 genu kapsulární polymerázy csb je popsána jako inaktivována fázovou variací a tedy nefunkční. U izolátu 0032/11 byla popsána nová alelová varianta genu cssC – 374, která mutací získala interní stop kodon. Izolát 0064/10 obsahoval nekompletní sadu kapsulárních genů (cssA, cssB, cssC, ctrG) a u izolátu 0068/10 nebyly detekovány v kapsulárním regionu A žádné geny.
2. Molekulární charakterizace
Molekulární charakterizace zahrnovala v první řadě MLST geny (abcZ, adk, aroE, fumC, gdh, pdhC a pgm) a lokusy finetypingu (2 variabilní regiony genu porA a 1 variabilní region genu fetA), které se využívají v aktuální nomenklatuře k molekulárnímu popisu kmene [15]. Dále byly analyzovány ribozomální geny, gen penA (antibiotická rezistence) a geny antigenů MenB vakcín (fHbp, nhba, nadA). Na základě alelových variant těchto genů jsme u každého izolátu určili finetyping, sekvenační typ (ST) a příslušnost ke klonálnímu komplexu (cc), ribozomální sekvenační typ (rST) a BAST typ, což je kombinace alelových variant genů antigenů MenB vakcín [4, 6, 15, 16]. Molekulární charakterizaci všech izolátů prezentují tabulky 2 a 3.
Tab. 2. Molekulární charakterizace 59 izolátů N. meningitidis, 2010–2019, Česká republika
Table 2. Molecular characterization of 59 N. meningitidis isolates, 2010–2019, Czech Republic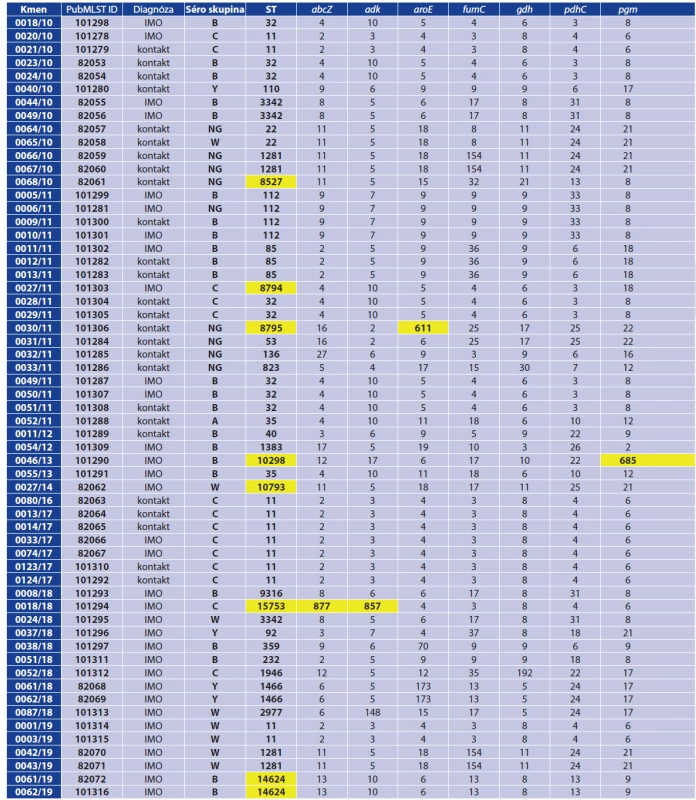
IMO = invazivní meningokokové onemocnění, NG (non-groupable) = izolát, u kterého nebylo možné určit séroskupinu, ST = sekvenační typ, cc = klonální komplex, UA (unassigned) = v databázi PubMLST nepřiřazen ST do klonálního komplexu, rST = ribozomální sekvenační typ, VR, VR1, VR2 = variabilní regiony genů porA a fetA, červené písmo = alela, která je v PubMLST databázi definována jako inaktivní, žluté pole = nově popsaná alelová varianta/nová alela, která je ve fázi registrace v databázi PubMLST / nově popsaný sekvenační typ/nově popsaný ribozomální sekvenační typ 
IMD = invasive meningococcal disease, NG (non-groupable) = serogroup could not be determined, ST = sequence type, cc = clonal complex, UA (unassigned) = ST not assigned to cc in the PubMLST database, rST = ribosomal sequence type, VR, VR1, VR2 = variable regions of the porA and fetA genes, Writing in red = allele defined as inactive in the PubMLST database, Yellow box = newly described allelic variant /new allele at the stage of registration in the PubMLST database/newly described sequence type/newly described ribosomal sequence type Tab. 3. Analýza genů antigenů MenB vakcín u 59 izolátů N. meningitidis, 2010–2019, Česká republika
Table 3. Analysis of MenB vaccine antigen genes in 59 N. meningitidis isolates, 2010–2019, Czech Republic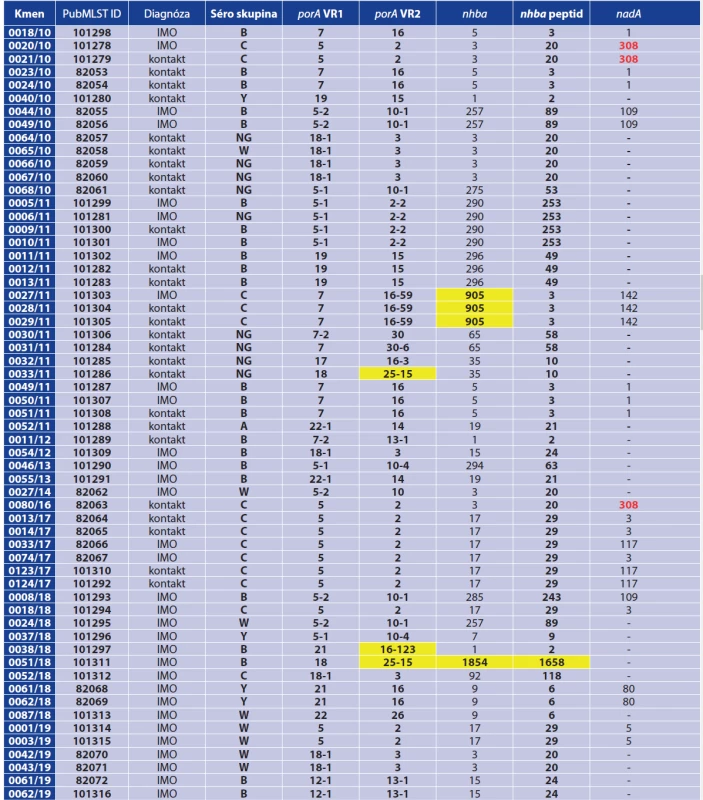
IMO = invazivní meningokokové onemocnění, NG (non-groupable) = izolát, u kterého nebylo možné určit séroskupinu, VR1, VR2 = variabilní regiony genu porA,
červené písmo = alela, která je v PubMLST databázi definována jako inaktivní (neprodukuje peptid), žluté pole = nově popsaná alelová nebo peptidová varianta
/ nově popsaný BAST typ, zelené pole = MenDeVAR index – izolát obsahuje jednu nebo více antigenních variant, které se nacházejí v MenB vakcínách, oranžové
pole = MenDeVAR index – izolát obsahujíce jednu nebo více antigenních variant, u kterých byla v experimentálních studiích pozorována zkřížená reaktivita, šedé
pole = MenDeVAR index – izolát, pro jehož antigenní varianty nejsou zatím k dispozici dostatečná data v databázi PubMLST
IMD = invasive meningococcal disease, NG (non-groupable) = serogroup could not be determined, VR1, VR2 = variable regions of the porA gene, Writing in red = allele defined as inactive in the PubMLST database (does not produce peptide), Yellow box = newly described allelic or peptide variant /newly described BAST type, Green box = MenDeVAR index – isolate contains one or more antigenic variants present in MenB vaccines, Orange box = MenDeVAR index – isolate contains one or more antigenic variants found to be cross- reactive in experimental studies, Grey box = MenDeVAR index – isolate, for antigenic variants of which sufficient PubMLST data have not been available. 2. 1. MLST geny a ST typy
U 59 studovaných izolátů bylo detekováno 28 různých sekvenačních typů, které příslušely k 13 různým klonálním komplexům. 8 izolátů (5 různých ST) nebylo ke konkrétnímu klonálnímu komplexu přiřazeno (UA, unassigned). Sedm různých sekvenačních typů bylo námi v PubMLST databázi nově registrováno. Ve čtyřech případech se jednalo o nové kombinace známých alel (ST-8527, ST-8794, ST-10793 a ST-14624), u tří izolátů byla nalezena nová alela jednoho z MLST genů. U izolátu 0030/11 (ST-8795) – nová alela 611 genu aroE a izolát 0046/13 (ST-10298) nesl novou alelu genu pgm (685). Jeden izolát (0018/18) obsahoval nové alelové varianty u dvou MLST genů – abcZ (877) a adk (857) a byl v PubMLST databázi registrován jako nový sekvenační typ ST-15753.
2. 2. Lokusy finetypingu a gen porA
U 59 izolátů bylo stanoveno 26 různých variant finetypingu, což je kombinace alelových variant variabilních regionů genů porA a fetA (porA_VR1, porA_VR2 a fetA_VR1). S výjimkou dvou nově popsaných variant lokusu porA_VR2 (16-123 a 25-15) byly všechny alelové varianty finetypingových lokusů již známy.
Alela genu porA byla nalezena u všech 59 izolátů v celkem 27 různých variantách. V případě zkrácených alelových variant ,porA to bylo 24 různých alel, protože 4 různé alely genu porA (1, 393, 2425 a 2970) obsahují stejnou sekvenci zkrácené varianty ’porA – 9. V databázi PubMLST jsme nově registrovali 3 alelové varianty u genu porA (2969, 2970 a 2971), u zkrácené alelové varianty ’porA to bylo 8 nových variant (208, 245, 246, 247, 248, 249, 250 a 251). U izolátu 0031/11 nebyla dosud nová alelová varianta lokusu ’porA registrována. Dvě alely genu porA (393 a nová 2970) získaly mutací interní stop kodon, a ztratily tak funkci produkovat kompletní protein.
2. 3. Ribozomální geny a rST typy
Již popsané kombinace alel ribozomálních genů byly zjištěny u 30 izolátů z 59 studovaných (15 různých ribozomálních sekvenačních typů – rST). 16 izolátů neslo dosud neznámé kombinace známých ribozomálních alel a bylo podle nich v databázi PubMLST popsáno 8 různých nových ribozomálních profilů (rST-149785, rST-149786, rST-163665, rST-163666, rST-163668, rST - -163672, rST-163674 a rST-163675). U zbývajících 13 izolátů byly nalezeny mutované, dosud nepopsané alelové varianty ribozomálních genů (14 různých genů). Po registraci nových alel v PubMLST databázi a následném přidělení číselných označení těmto alelám bylo definováno dalších 10 nových ribozomálních profilů (rST-149782, rST-149783, rST-149784, rST-163663, rST-163664, rST-163667, rST-163669, rST-163670, rST - -163671 a rST-163673).
2. 4. Gen penA
Gen penA se u 59 studovaných izolátů objevil v 18 různých alelových variantách. V případě zkrácených alelových variant ’penA se jednalo o 13 různých, protože 6 alel kompletního genu penA (1, 5, 24, 59, 89 a 205) obsahuje stejnou sekvenci zkrácené alelové varianty ’penA-1. Tři alely genu penA nebyly dosud v PubMLST registrovány: nová alela 2411 u izolátu 0046/13 (zkrácená alelová forma ’penA-403), která se lišila od popsané alely 83 subtitucí 3 nukleotidů, alela 2412 u izolátu 0031/11, která se lišila od alely 2207 osmi substitucemi (obsahovala známou zkrácenou formu ’penA-11) a alela 2413 u izolátu 0051/18 (’penA-171), která vznikla jednou bodovou mutací z alely 5.
2. 5. Geny MenB vakcín a BAST typy
Gen nhba byl detekován u všech izolátů. Jednalo se celkem o 19 různých alelových variant, které kódovaly 18 různých proteinů. Všechny alely byly aktivní a schopné produkovat příslušný protein. Námi nově popsaná alela 905 u izolátů 0027/11, 0028/11 a 0029/11 produkuje stejnou proteinovou variantu 3 jako známá alela 5. Izolát 0051/18 nesl pak nově popsanou variantu genu nhba 1854, která produkuje i novou proteinovou variantu – 1658.
Gen nadA byl nalezen u 26 izolátů z 59, což odpovídá faktu, že tento gen se nachází jen u části populace N. meningitidis. U 23 izolátů se jednalo o aktivní alely, schopné produkovat příslušný protein (7 různých alel a 7 různých peptidových variant). Tři izoláty našeho výběru (0020/10, 0021/10 a 008/16) disponovaly alelou 308, která je inaktivována IS elementem a funkční protein neprodukuje.
Alela genu fHbp byla přítomna u všech izolátů (22 různých alel). S výjimkou jedné byly všechny alely aktivní. Izolát 0080/16 nesl alelovou variantu 917 (’fHbp 669), která mutací získala interní stop kodon a funkční protein neprodukuje. Byly popsány čtyři nové alely genu fHbp – u izolátu 0052/18 alela 1228 (ve zkrácené formě ’fHbp 1741), která kóduje novou proteinovou variantu (1342) a nová alela 1227 u izolátu 0038/18 (’fHbp 1739), která obsahovala jednobodovou synonymní mutaci v kodonu pro glycin (GGG – GGC) na pozici 15 a neměla tedy vliv na alelovou variantu produkovaného proteinu (peptidová varianta 464). U izolátu 0046/13 byla nalezena nová alela 1271, která obsahuje stejnou sekvenci zkrácené varianty ’fHbp 13 jako známá alela 145. Izolát 0051/18 nesl pak gen fHbp v nové alelové variantě 1272 (’fHbp 312).
U všech 59 studovaných izolátů byl určen příslušný BAST typ (BEXSERO® antigen sequence typing), který je v PubMLST databázi definován jako kombinace alelových variant dvou variabilních regionů genů porA (porA_VR1, porA_VR2) a peptidových variant tří genů antigenů vakcíny MenB-4C (nhba, nadA, fHbp). Celkem bylo zjištěno 32 různých BAST typů, z toho 23 bylo v databázi PubMLST již registrováno. Deset izolátů z našeho souboru obsahovalo buď neznámou kombinaci BAST lokusů nebo neslo novou alelovou variantu v některém z nich. Podle těchto izolátů bylo námi popsáno 9 nových BAST typů: BAST-2939, BAST-3530, BAST-3887, BAST-3888, BAST-3889, BAST-3959, BAST-3960, BAST-3961 a BAST-3962.
Kromě určení BAST typu, který poskytuje informaci o pokrytí izolátu vakcínou Bexsero [4], byl recentně v databázi PubMLST představen index MenDeVAR (Meningococcal Deduced Vaccine Antigen Reactivity Index), který informuje o funkčním účinku obou MenB vakcín (Bexsero, Trumenba) na daný izolát [17]. Men - DeVAR index je založen na zpracování informací o přítomnosti jednotlivých antigenních variant MenB vakcín, jejich expresi a citlivosti těchto antigenních variant na protilátky v baktericidním testu. Na základě Men - DeVAR indexu jsou izoláty zařazeny do 4 různých, barevně odlišených skupin, a to ve vztahu k oběma MenB vakcínám. Zeleně jsou označeny izoláty obsahující jednu nebo více antigenních variant, které se nacházejí v MenB vakcínách. V případě vakcíny Bexsero to jsou: peptidová varianta 1 genu fHbp, peptidová varianta 2 genu nhba, peptidová varianta 8 genu nadA a varianta 4 lokusu porA_VR2. Vakcína Trumenba obsahuje peptidové varianty 45 a 551 genu fHbp. Oranžová barva značí izoláty obsahující jednu nebo více antigenních variant, u kterých byla v experimentálních studiích pozorována zkřížená reaktivita. Šedě jsou označeny izoláty, pro jejichž antigenní varianty nejsou zatím k dispozici dostatečná data a červená barva patří izolátům nesoucím antigenní varianty, u kterých zkřížená reaktivita v experimentálních studiích nebyla prokázána.
V našem souboru 59 izolátů byla u 14 zjištěna jedna nebo více antigenních variant, které se nacházejí ve vakcíně Bexsero – viz tabulka 3. Antigenní varianty, u kterých byla v experimentálních studiích pozorována zkřížená reaktivita, byly zjištěny u 12 izolátů pro vakcínu Bexsero a u 28 izolátů pro vakcínu Trumenba. Antigenní varianty, pro které nejsou zatím k dispozici dostatečná data, byly zjištěny u 33 izolátů pro vakcínu Bexsero a u 31 izolátů pro vakcínu Trumenba. Lze očekávat, že ve světové literatuře a databázi PubMLST budou přibývat další podklady k určování indexu Men - DeVAR. Tento index poskytuje rychlou evidence-based informaci o přítomnosti a možné zkřížené reaktivitě různých MenB antigenních variant daného izolátu v souvislosti s oběma MenB vakcínami. Dostupnost těchto informací je významným přínosem pro hodnocení vakcinačních programů a jejich aktualizaci.
ZÁVĚRY
Studium izolátů N. meningitidis metodou WGS poskytlo detailní molekulární charakterizaci (MLST geny, lokusy finetypingu, ribozomální geny a geny antigenů MenB vakcín). U velké části genů byly zjištěny nové, mutované alelové varianty, které byly registrovány v databázi PubMLST a následně anotovány kurátorem a očíslovány díky vkládacímu nástroji platformy BIGSdb. Získané výsledky prohlubují znalosti o přenosu invazivních a neinvazivních kmenů N. meningitidis v populaci. Metodou WGS byla získána detailní data o pokrytí těchto kmenů novými MenB vakcínami.
Podpora projektu
Podpořeno z programového projektu Ministerstva zdravotnictví ČR s reg. č. NV19-09-00319. Veškerá práva podle předpisů na ochranu duševního vlastnictví jsou vyhrazena.
Do redakce došlo dne 26. 4. 2021.
Adresa pro korespondenci:
MUDr. Pavla Křížová, CSc.
SZÚ Praha
Šrobárova 48
100 42 Praha 10
e-mail: pavla.krizova@szu.cz
Zdroje
1. Caugant DA, Brynildsrud OB. Neisseria meningitidis: using genomics to understand diversity, evolution and pathogenesis. Nat Rev Microbiol, 2020;18(2):84–96. DOI:10.1038/s41579-019-0282-6.
2. Jolley KA, Wilson DJ, Kriz P, et al. The influence of mutation, recombination, population history, and selection on patterns of genetic diversity in Neisseria meningitidis. Mol Biol Evol, 2005;22(3):562–569. DOI:10.1093/molbev/msi041.
3. Feil EJ, Enright MC, Spratt BG. Estimating the relative contributions of mutation and recombination to clonal diversification: a comparison between Neisseria meningitidis and Streptococcus pneumoniae. Res Microbiol, 2000;151(6):465–946. DOI:10.1016/ s0923-2508(00)00168-6.
4. Brehony C, Rodrigues CMC, Borrow R, et al. Distribution of Bexsero ® Antigen Sequence Types (BASTs) in invasive meningococcal disease isolates: Implications for immunisation. Vaccine, 2016;34(39):4690–4697. DOI:10.1016/j.vaccine.2016.08.015.
5. https://pubmlst.org/organisms/neisseria-spp/
6. Maiden MCJ, Bygraves JA, Feil E, et al. Multilocus sequence typing: a portable approach to the identification of clones within populations of pathogenic microorganisms. Proc Natl Acad Sci USA, 1998;95(6):3140–3145. DOI:10.1073/pnas.95.6.3140.
7. Brehony C, Jolley KA, Maiden MCJ. Multilocus sequence typing for global surveillance of meningococcal disease. FEMS Microbiol Rev, 2007;31(1):15–26. DOI:10.1111/j.1574 - 6976.2006.00056.x.
8. Jolley KA, Maiden MCJ. Using multilocus sequence typing to study bacterial variation: prospects in the genomic era. Future Microbiol, 2014;9(5):623–630. DOI:10.2217/fmb.14.24.
9. PCR for Detection and Characterization of Bacterial Meningitis Pathogens: Neisseria meningitidis, Haemophilus influenzae, and Streptococcus pneumoniae, in Laboratory Methods for the Diagnosis of Meningitis caused by Neisseria meningitidis, Streptococcus pneumoniae and Haemophilus influenzae, CDC manual, Chapter
10. Dostupné na www: https://www.cdc.gov/meningitis/ lab-manual/chpt10-pcr.html. 10. Manuál QIAamp DNA Mini Kit. Dostupné na www: http:// www.qiagen.com/Products/Catalog/Sample-Technologies/ DNA-Sample-Technologies/Genomic-DNA/ QIAamp-DNA-Mini-Kit#technicalspecification.
11. Zerbino DR. Using the Velvet de novo assembler for shortread sequencing technologies. Curr Protoc Bioinformatics, 2010;11(11.5). DOI:10.1002/0471250953.bi1105s31.
12. Jolley KA, Maiden MCJ. BIGSdb: Scalable analysis of bacterial genome variation at the population level. BMC Bioinformatics, 2010;11 : 595. DOI:10.1186/1471-2105-11-595.
13. Harrison OB, Claus H, Jiang, et al. Description and Nomenclature of Neisseria meningitidis Capsule Locus. Emerg Infect Dis, 2013;19(4):566–573. DOI:10.3201/eid1904.111799.
14. Marjuki H, Topaz N, Rodriguez-Rivera LD, et al. Whole-Genome Sequencing for Characterization of Capsule Locus and Prediction of Serogroup of Invasive Meningococcal Isolates. J Clin Microbiol, 2019;57(3). DOI: 10.1128/JCM.01609-18.
15. Jolley KA, Brehony C, Maiden MCJ. Molecular typing of meningococci: recommendations for target choice and nomenclature. FEMS Microbiol Rev, 2007;31(1):89–96. DOI: 10.1111/j.1574 - 6976.2006.00057.x.
16. Jolley KA, Bliss CM, Bennett JS, et al. Ribosomal multilocus sequence typing: universal characterization of bacteria from domain to strain. Microbiology (Reading), 2012;158(4):1005–1015. DOI: 10.1099/mic.0.055459-0.
17. Rodrigues ChMC, Jolley KA, Smith A, et al. Meningococcal Deduced Vaccine Antigen Reactivity (MenDeVAR) Index: a Rapid and Accessible Tool That Exploits Genomic Data in Public Health and Clinical Microbiology Applications. J Clin Microbiol, 2020;59(1). DOI: 10.1128/JCM.02161-20.
Štítky
Hygiena a epidemiologie Infekční lékařství Mikrobiologie
Článek vyšel v časopiseEpidemiologie, mikrobiologie, imunologie
Nejčtenější tento týden
2021 Číslo 3- Jak souvisí postcovidový syndrom s poškozením mozku?
- Stillova choroba: vzácné a závažné systémové onemocnění
- Perorální antivirotika jako vysoce efektivní nástroj prevence hospitalizací kvůli COVID-19 − otázky a odpovědi pro praxi
-
Všechny články tohoto čísla
- Pokud by byla k dispozici vakcína proti covid-19, chtěli byste být očkováni? A jste očkováni proti chřipce a jiným nemocem? Průzkum mezi studenty vysokých škol v době nouzového stavu
- Citlivost testů antigenu SARS-CoV-2 z hlediska testování ve velkém měřítku
- Evaluation of tuberculosis diagnostic tools, with extending MODS assay use to second line susceptibility testing
- Detailní molekulární charakterizace izolátů Neisseria meningitidis metodou sekvenace celého genomu (WGS), Česká republika, 2010–2019
- Možnosti diagnostiky lidských polyomavirů v klinické praxi
- Co víme a stále nevíme o klíšťové encefalitidě?
- Autozápalový proces v patogenéze generalizovanej pustulárnej psoriázy a perspektívy jej cielenej liečby
- Epidemiologie, rizikové faktory a možnosti prevence akutních leukemií
- Epidemiologie, mikrobiologie, imunologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Epidemiologie, rizikové faktory a možnosti prevence akutních leukemií
- Co víme a stále nevíme o klíšťové encefalitidě?
- Možnosti diagnostiky lidských polyomavirů v klinické praxi
- Autozápalový proces v patogenéze generalizovanej pustulárnej psoriázy a perspektívy jej cielenej liečby
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Autoři: MDDr. Eleonóra Ivančová, PhD., MHA
Autoři: prof. MUDr. Eva Kubala Havrdová, DrSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



