-
Medical journals
- Career
Využití molekulárně cytogenetických technik při analýze chromozomových aberací u hematologických malignit
Authors: J. Březinová 1; I. Šárová 1; Š. Ransdorfová 1; Z. Zemanová 2; K. Michalová 1,2
Authors‘ workplace: Ústav hematologie a krevní transfuze, Praha 1; Centrum nádorové cytogenetiky, Ústav lékařské biochemie a laboratorní diagnostiky VFN a 1. LF UK, Praha 2
Published in: Transfuze Hematol. dnes,23, 2017, No. Supplementum1, p. 48-53.
Category:
Overview
Cytogenetická analýza buněk kostní dřeně je jedním ze základních vyšetření nemocných s maligním hematologickým onemocněním. Přispívá k upřesnění diagnózy, určení stadia nemoci, sledování jejího průběhu a stanovení prognózy. Detekce chromozomových aberací je limitována rozlišovací schopností metody, proto jsou v současné době využívány i citlivější molekulárně cytogenetické techniky založené na fluorescenční in situ hybridizaci. Umožňují rychlou a přesnou analýzu početních a strukturních změn chromozomů, detekci kryptických přestaveb i určení zlomových míst a jednotlivých alterovaných genů. Různé modifikace molekulárně cytogenetických metod jsou využívány jak v rutinní laboratorní praxi, tak i ve výzkumu. S jejich využitím můžeme detekovat minimální zbytkovou chorobu, sledovat benigní proliferaci i případnou klonální evoluci, a posuzovat tak úspěšnost léčby. Nové objevy molekulárních změn přispívají k lepšímu porozumění patogenezi onemocnění. Stratifikují nemocné k cílené terapii a odhalují další genetické příčiny a mechanismy vzniku a vývoje leukemického klonu.
KLÍČOVÁ SLOVA:
klasická cytogenetická analýza – FISH – leukemie – preleukemie
Cytogenetická analýza buněk kostní dřeně je jedním ze základních vyšetření nemocných s preleukemiemi a leukemiemi, a to jak při diagnóze, tak i v průběhu onemocnění. Podává informace, sloužící k upřesnění diagnózy, stanovení prognózy a určení remise, případně relapsu choroby. Cytogenetické výsledky se proto staly nedílnou součástí mezinárodních prognostických skórovacích systémů i jejich revidovaných verzí. Například u myelodysplastického syndromu (MDS) byla dlouhou dobu tzv. „zlatým standardem“ práce Greenberga et al. z roku 1997, která rozdělovala nemocné do tří prognostických skupin – příznivé, střední a nepříznivé. S příznivou prognózou byly spojeny samostatné delece dlouhých ramen chromozomů 5 a 20, samostatná ztráta chromozomu Y a normální karyotyp. Aberace chromozomu 7 a komplexní karyotyp, zahrnující přestavby 3 a více chromozomů, poukazovaly na nepříznivou prognózu. Ostatní aberace představovaly prognózu střední [1]. Později byl tento systém revidován na velkém souboru 7 012 nemocných a na základě cytogenetického nálezu a dalších parametrů bylo vytvořeno pět prognostických skupin – velmi dobrá, dobrá, střední, špatná a velmi špatná [2]. Podobný mezinárodní prognostický skórovací systém byl vypracován a dále revidován i pro nemocné s akutní myeloidní leukemií (AML) [3, 4].
Genomové alterace, vedoucí k aktivaci onkogenů či ztrátě nádorových supresorových genů, jsou rozhodujícími mechanismy při vzniku a vývoji nádorového onemocnění. Detekce chromozomových změn metodou klasické cytogenetické analýzy není však vždy zcela informativní – ať už z důvodu nízkého počtu dělících se buněk, nedostatečné kvality mitos nebo přítomnosti kryptických aberací, které jsou pod rozlišovací schopností metody (přibližně 4 Mb). V těchto případech nacházejí uplatnění citlivější molekulárně cytogenetické techniky, založené na hybridizaci in situ s využitím fluorochromy značených DNA sond, tzv. fluorescenční in situ hybridizace (FISH) [5]. Principem metody je vazba specifické jednořetězcové DNA sondy ke komplementárnímu úseku cílové DNA pacienta na mikroskopickém preparátu. Podle lokalizace cílových míst lze sondy pro FISH analýzu dělit na satelitní, lokus specifické a malovací. Satelitní sondy hybridizují s repetitivními satelitními DNA sekvencemi, které se nacházejí především v oblasti centromer a telomer. Využívají se k vyšetření aneuploidí či k určení chromozomů neznámého původu (tzv. marker chromozomů). Lze jimi detekovat centromery, telomery nebo heterochromatinovou oblast na Y chromozomu. Lokus specifické sondy se váží na jedinečné sekvence DNA. Slouží k lokalizaci jednotlivých genů nebo specifických úseků DNA. Jde zpravidla o genomické klony, které se liší velikostí v závislosti na klonovacím vektoru. Tím může být plazmid (55 bp – 5 kb), kosmid (20–50 kb), bakteriofág lambda (8–15 kb), umělé kvasinkové chromozomy YAC (Yeast artificial chromosomes, 50–1 000 kb), umělé bakteriální klony BAC (Bacterial artificial chromosomes 150–350 kb) a další vektory [5]. Malovací sondy obsahují množství chromozomových sekvencí, a označují tak celý chromozom nebo celá dlouhá, případně celá krátká ramena. Slouží k detekci strukturních i početních odchylek a k identifikaci marker chromozomů. Oproti předchozím dvěma typům sond je nelze použít k analýze interfázních jader, ve kterých je díky despiralizaci chromozomů viditelný pouze rozptýlený, difuzní signál. Specifickým využitím malovacích sond je sonda pro mnohobarevnou FISH (mFISH) a mnohobarevné pruhování (mBAND). Princip metody mFISH (MetaSystems, Germany) spočívá ve značení jednotlivých chromozomových párů pěti různými fluorochromy a jejich kombinacemi, což umožňuje barevně odlišit všech 22 párů autozomů i dva pohlavní chromozomy. Marker chromozomy, komplexní chromozomové aberace i početní změny chromozomů tak lze současně identifikovat v jednom hybridizačním pokusu. Na podobném principu je založena i metoda mBAND (MetaSystems, Germany), kdy kombinací dvou a více různých fluorochromů není značen celý genom, ale pouze fragment DNA, získaný z jednoho chromozomového páru. Vyhodnocením zastoupení jednotlivých fluorochromů na aberovaném chromozomu pak můžeme přesně definovat jednotlivé intrachromozomové přestavby, zlomová místa, rozsah delece či inverze a další změny.
Pro vizualizaci sond se dříve využívalo radioaktivní značení, v současné době sondy obsahují již zabudované fluorochromy a identifikují se přímo ve fluorescenčním mikroskopu. Sondy pro hybridizaci lze připravovat nejrůznějšími způsoby v laboratoři, nejjednodušším a nejrozšířenějším postupem je však využití komerčně dostupných kitů, které obsahují značenou sondu s příslušným detekčním systémem.
První práce, využívající fluorescenčně značené DNA sondy v analýze chromozomových aberací, byly publikovány v 80. letech minulého století [6, 7]. Na našem pracovišti jsme metodu FISH zavedli v 90. letech a v současné době používáme všechny její dostupné modifikace. První kazuistiku, využívající k cytogenetické analýze buněk kostní dřeně tuto techniku, jsme publikovali v roce 1996. U pacientky s diagnózou AML jsme prokázali buněčný klon s nebalancovanou translokací, která měla za následek parciální trizomii dlouhých ramen chromozomu 1 v oblasti 1q22 až 1qter a parciální monozomii krátkých ramen chromozomu 6 v oblasti 6p21 až 6pter. Nález jsme ověřili pomocí celochromozomových malovacích sond pro chromozomy 1 a 6, které nám umožnily přesnější identifikaci přestavby i zlomových míst, než bychom byli schopni určit metodou klasické cytogenetické analýzy [8].
V současné době využíváme molekulárně cytogenetické techniky k analýze buněk kostní dřeně a periferní krve v běžné rutinní diagnostice, při screeningových vyšetřeních i ve výzkumu. Například u všech nemocných s nově diagnostikovanou AML aplikujeme ihned po zpracování dodaného vzorku FISH s panelem vybraných sond pro detekci delece 5q, monozomie 7/delece 7q, trizomie 8/9 a přestaveb genu KMT2A (MLL). Tyto aberace jsou prognosticky významné, a poskytují proto ošetřujícím lékařům důležité informace v kratší době než výsledky klasické cytogenetické analýzy. V současné době máme tímto panelem sond vyšetřený a zpracovaný soubor 245 nemocných s nově diagnostikovanou AML, u všech jsme při analýze buněk kostní dřeně použili lokus specifické sondy Vysis LSI EGR1/D5S23, D5S721 Dual Color Probe Kit, Vysis LSI 7q31/CEP 7, Vysis LSI MLL Dual Color, Break Apart Rearrangement Probe a centromerické sondy Vysis CEP 8 SpectrumOrange Direct Labeled Fluorescent DNA Probe Kit a Vysis CEP 9 SpectrumGreen Probe (Abbott Molecular, USA). Deleci chromozomové oblasti 5q31 jsme prokázali u 12 % nemocných, deleci/monozomii chromozomu 7 u 11 % nemocných. Přestavbu, nadpočetnou kopii či amplifikaci genu KMT2A (MLL) jsme identifikovali u 15 % nemocných, trizomii chromozomu 8 u 10 % nemocných (obr. 1).
Image 1. Výsledky screeningového vyšetření FISH při použití panelu sond pro detekci delece 5q, delece 7q, amplifikace KMT2A (MLL) a trizomie 8 (Abbott Molecular, USA) u nemocných s nově diagnostikovanou AML 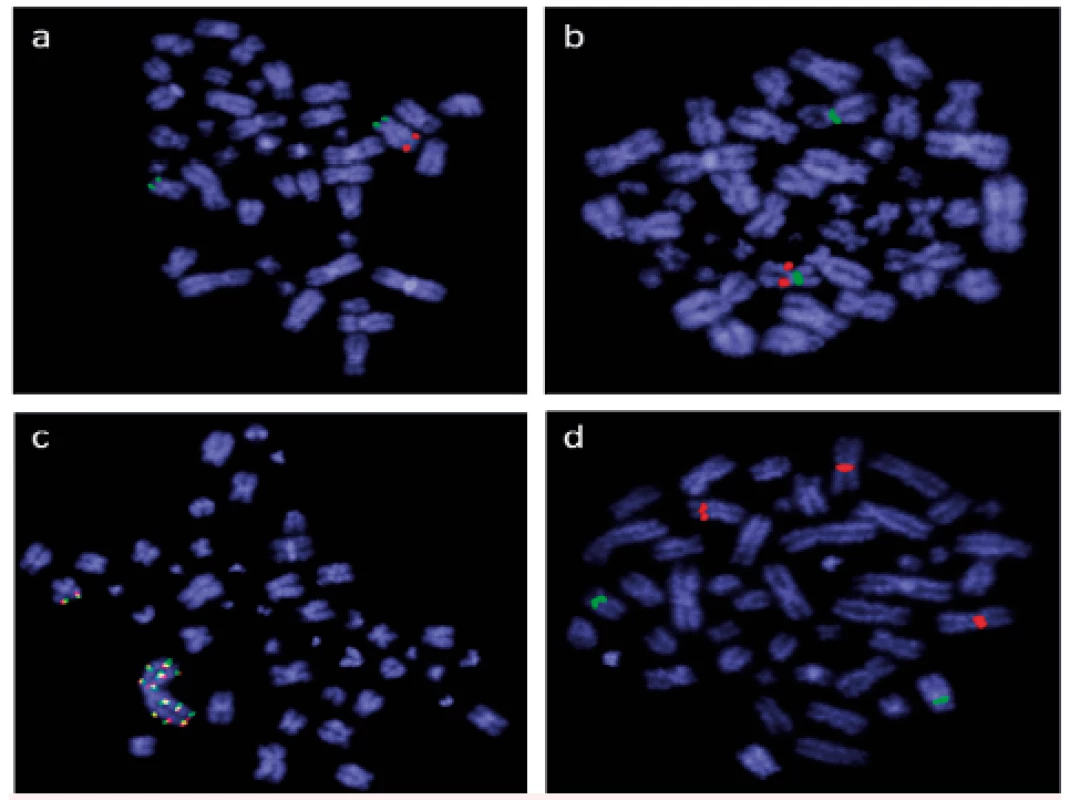
a) lokus specifická sonda Vysis LSI EGR1/D5S23, D5S721 Dual Color Probe Kit prokázala deleci oblasti 5q31 (pouze 1 červený signál) b) lokus specifická sonda Vysis LSI 7q31/CEP 7 identifikovala deleci oblasti 7q31 (pouze 1 červený signál, dvě centromery chromozomu 7 značeny zeleně) c) lokus specifická sonda Vysis LSI MLL Dual Color, Break Apart Rearrangement Probe odhalila amplifikaci genu KMT2A (MLL) d) centromerická sonda Vysis CEP 8 SpectrumOrange Direct Labeled Fluorescent DNA Probe Kit společně s centromerickou sondou Vysis CEP 9 SpectrumGreen Probe potvrdily nález trizomie chromozomu 8 (červeně značená centromera chromozomu 8, zeleně chromozomu 9) Do rutinní praxe jsme zavedli metodu interfázní FISH (I-FISH) také pro analýzu CD34+ buněk v periferní krvi u nemocných s myelodysplastickými syndromy jako alternativu k I-FISH analýze buněk kostní dřeně. Analýza separovaných CD34+ buněk pomocí I-FISH je vhodná metoda pro sledování aberantního klonu v periferní krvi. Jedná se o cílené vyšetření, které umožňuje monitorovat pouze konkrétní vybrané aberace. Tato metoda tak nenahrazuje počáteční vyšetření buněk kostní dřeně, kdy je nutné určit chromozomové změny v karyotypu pomocí klasické cytogenetiky. Neodráží ani klonální vývoj, neboť nedetekuje další změny karyotypu, a nemůže tedy nahradit klasickou cytogenetickou analýzu. Může ji ale vhodně doplnit v intervalech mezi odběry kostní dřeně. Zvláště u pacientů s nízkým rizikem transformace do akutní myeloidní leukemie je žádoucí častější sledování v průběhu léčby (nejlépe s odstupem 1 měsíce). V těchto případech je analýza chromozomových aberací prováděná z periferní krve méně invazivní než biopsie kostní dřeně a může pomoci efektivněji vést léčbu založenou na molekulárně cytogenetické odpovědi. Lze ji použít k šetrnému sledování průběhu nemoci, pokud nelze odebrat kostní dřeň pro suchou punkci, případně když pacient tento odběr odmítá [9].
Cílem našeho dlouhodobého výzkumu jsou delece chromozomů. V deletovaných úsecích jsou často lokalizovány nádorové supresorové geny, které mají důležitou úlohu v regulaci buněčné proliferace, diferenciace i v jiných buněčných a systémových procesech. Jejich produkty inhibují buněčný cyklus, mutace či delece těchto genů vede ke ztrátě inhibiční funkce a představuje jeden z kroků v procesu nádorové transformace buňky. V roce 2000 jsme publikovali studii 77 nemocných s hematologickým maligním onemocněním a s delecí dlouhých ramen chromozomu 5. Klasickou cytogenetickou analýzou jsme suspektní deleci 5q nalezli u 63 nemocných, u 14 dalších byla delece 5q provázena i komplexními přestavbami. Metodou FISH s lokus specifickými sondami pro oblast 5q31 (Oncor, USA) jsme samostatnou deleci 5q potvrdili u 23 nemocných, u 12 nemocných jako součást komplexních přestaveb karyotypu. Strukturní přestavby chromozomu 5 jsme u všech nemocných ověřovali malovací sondou pro chromozom 5. V práci jsme potvrdili nezbytnost FISH analýzy pro detekci malých patologických klonů [10]. Soubor nemocných s delecí 5q jsme v průběhu let neustále rozšiřovali a v roce 2014 výsledky analýz komplexně vyhodnotili v publikaci. Kombinací molekulárně cytogenetických technik jsme provedli detailní analýzu buněk kostní dřeně u 157 nemocných s nově diagnostikovaným MDS s komplexními přestavbami karyotypu a delecí 5q. U žádného z nemocných jsme nenalezli monozomii chromozomu 5 – ve všech případech, kdy bylo při klasické cytogenetické analýze podezření na tuto monozomii, prokázala metoda mFISH přítomnost alespoň části aberovaného chromozomu 5 na jiném místě v karyotypu. Potvrdili jsme tak hypotézu, že deletovaný chromozom 5 je značně nestabilní a často vstupuje do různých, převážně nereciprokých kryptických přestaveb. Všichni nemocní s komplexními přestavbami a delecí 5q měli velmi špatnou prognózu s mediánem přežití dva měsíce. U 47 % nemocných jsme prokázali chromozomové aberace, vzniklé v důsledku chromotripsis [11]. Při tomto procesu jeden či více chromozomů v nádorové buňce podléhá desítkám až stovkám přestaveb při jedné katastrofické události. Nejjednodušší vysvětlení jejich vzniku představuje fragmentace chromozomů na vysoký počet malých segmentů, které jsou zpětně nepřesně sestaveny pomocí DNA opravných mechanismů [12, 13]. Identifikace těchto změn je pod rozlišovací schopností klasické cytogenetické analýzy a je možná pouze na základě využití molekulárně cytogenetických metod a čipových technologií.
Zabýváme se rovněž delecemi dlouhých ramen chromozomu 20. V roce 2014 jsme mapováním pomocí BAC sond společně s molekulární analýzou DNA prokázali vznik fúzního genu ASXL1/TSHZ2 u nemocného s MDS a derivovaným chromozomem 20 [14]. Zatímco samostatná delece 20q řadí nemocné do skupiny s příznivou prognózou, jsou alterace ASXL1 genu u nemocných s MDS spojené se zhoršeným průběhem onemocnění. Cílem naší současné studie proto bylo identifikovat v souboru 34 nemocných se samostatnou delecí 20q zlomová místa v genu ASXL1, určit frekvenci parciálních/úplných delecí tohoto genu a vyhodnotit rozdíly v době přežití ve skupinách s alterovaným a nealterovaným genem ASXL1. Mapováním s využitím BAC sond, pokrývajících oblast 20q11.21 až 20q13.2 (BlueGnome, UK) jsme prokázali alteraci genu ASXL1 (parciální nebo úplnou deleci) celkem u 19 nemocných (56 %). Rozdíly v přežívání mezi oběma skupinami nejsou statisticky významné (p = 0,15), což je patrně dáno malým počtem vyšetřených nemocných. Souvislost alterací genu ASXL1 s průběhem maligních hematologických onemocnění je stále předmětem intenzivního studia, a proto i my budeme klinický a prognostický význam přestaveb genu ASXL1 nadále sledovat.
Cílem našeho současného výzkumu je analýza dicentrických chromozomů (chromozomy se dvěma centromerami). Jsou považovány za iniciátory genomové nestability nádorových buněk, vedoucí k dalším chromozomovým aberacím a klonální evoluci patologického klonu. V průběhu buněčného dělení putují centromery těchto aberantních chromozomů k opačným pólům buňky, s následnými zlomy a opětovnými spojeními, což má za následek vznik chromozomových přestaveb, především delecí, duplikací a amplifikací. Dicentrické chromozomy často podstupují další sekundární změny, které je během dělení stabilizují – je to inaktivace, případně delece či excize jedné z centromer. Dochází tak ke vzniku sekundárních monocentrických chromozomů, jehož důsledkem je monozomie příslušného chromozomu [15]. Klasickou cytogenetickou analýzou nelze dicentrické chromozomy často zcela identifikovat, ani odlišit skutečné ztráty celých chromozomů od parciálních monozomií, kdy části chromozomů zůstávají součástí karyotypu ve formě marker chromozomů či jiných aberací. K detekci je nutné využít molekulárně cytogenetické metody. Jejich kombinaci jsme uplatnili ve studii, jejímž cílem bylo za pomoci kombinace mnohobarevné multicentromerické sondy XCen mFISH, mFISH a mBAND (MetaSystems, Germany) revidovat karyotypy pacientů s AML, u nichž byla klasickou cytogenetickou analýzou nalezena suspektní přítomnost dicentrických chromozomů (obr. 2). Naše studie odhalila dicentrické chromozomy, které nebylo možné identifikovat klasickou cytogenetickou analýzou a prokázala vyšší frekvenci dicentrických chromozomů, než se předpokládá na základě literárních údajů. Prokázali jsme rovněž asociaci mezi počtem dicentrických chromozomů v karyotypu a dobou přežívání nemocných [16]. Vzhledem k významné úloze dicentrických chromozomů v procesu iniciace i progrese nádorových onemocnění chceme nadále pokračovat v jejich analýze, vyhodnotit výskyt a zastoupení těchto aberací při diagnóze i v závislosti na léčbě a průběhu onemocnění. Komplexní analýzou všech získaných cytogenetických i molekulárně cytogenetických dat můžeme přispět k zpřesnění diagnostiky, prognostického rizika a tím i k efektivnější léčbě nemocných.
Image 2. Využití kombinace molekulárně cytogenetických technik při analýze dicentrických chromozomů 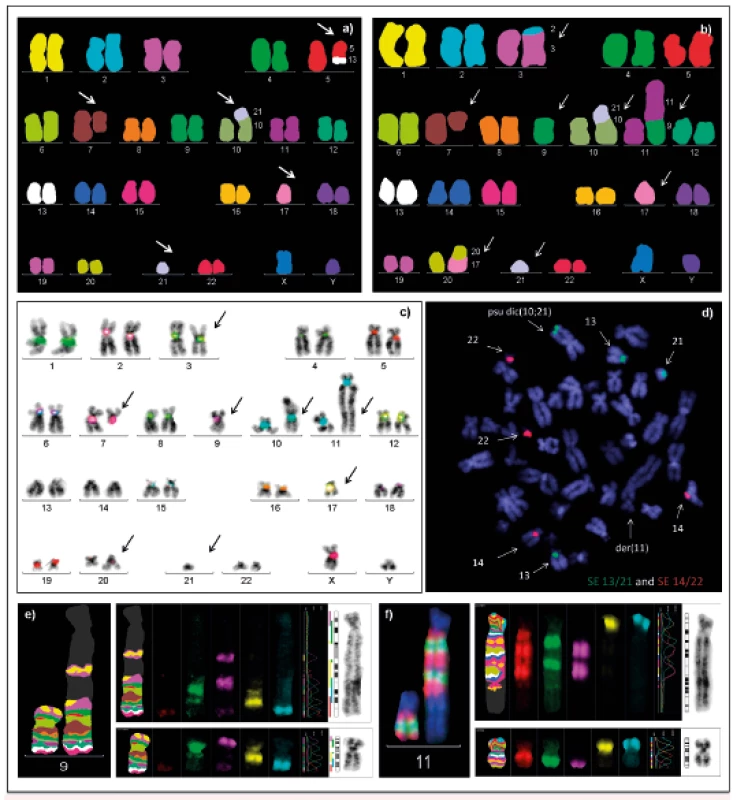
a) b) mFISH c) FISH s multicentromerickou sondou (XCen mFISH) d) FISH analýzy s kombinací centromerických sond pro chromozomy 13, 21 (SpectrumGreen) a 14, 22 (SpectrumOrange) e) mBAND pro chromozom 9 f) mBAND pro chromozom 11 I přes rychlý rozvoj molekulárně genetických metod, jako jsou např. čipové technologie a sekvenování nové generace (NGS), mají molekulárně cytogenetické metody v současné době stále své nezastupitelné místo jak v rutinní diagnostice buněk kostní dřeně a periferní krve, tak i v oblasti výzkumu nádorových onemocnění. Doplňují výsledky klasické cytogenetické analýzy, umožňují získat informace o změnách genomu v případě nedostatečného počtu dělících se buněk nebo špatné kvality mitos a přinášejí komplexní pohled na změny v genomu nádorových buněk. Určení specifických alterací pomocí molekulárně cytogenetických metod přispívá k identifikaci genů a odhalení dalších možných mechanismů, které se podílejí na nádorové transformaci buňky, a tím i na vzniku a rozvoji onemocnění.
Použité zkratky
AML – akutní myeloidní leukemie
ASXL1 – additional sex combs like 1
BAC – bacterial artificial chromosome
FISH – fluorescenční in situ hybridizace
I-FISH – interfázní fluorescenční in situ hybridizace
LSI – locus specific identification
mBAND – multicolor banding
mFISH – multicolor FISH
MDS – myelodysplastický syndrom
MLL – myeloid/lymphoid leukaemia
NGS – next generation sequencing
TSHZ2 – teashirt zinc finger homeobox 2
YAC – yeast artificial chromosome
Podíl autorů na přípravě rukopisu
JB – provedení a vyhodnocení mFISH analýz, zpracování podkladů a literatury, napsání rukopisu
IŠ – provedení a vyhodnocení FISH a mFISH analýz, připomínkování a úprava rukopisu, kontrola a schválení finální verze
ŠR – provedení a vyhodnocení FISH a mFISH analýz, připomínkování a úprava rukopisu, kontrola a schválení finální verze
ZZ – provedení a vyhodnocení mFISH analýz, připomínkování a úprava rukopisu, kontrola a schválení finální verze
KM – připomínkování rukopisu, kontrola a schválení finální verze
Čestné prohlášení autora
Autor práce prohlašuje, že v souvislosti s tématem, vznikem a publikací tohoto článku není ve střetu zájmů a vznik ani publikace článku nebyly podpořeny žádnou farmaceutickou firmou. Toto prohlášení se týká i všech spoluautorů.
Poděkování
Podpořeno projektem MZČR koncepčního rozvoje výzkumné organizace 00023736 ÚHKT a granty RVO-VFN0064165, GAČR P302/12/G157 a ProgresQ28/LF1.
RNDr. Jana Březinová, Ph.D.
Oddělení cytogenetiky
Ústav hematologie a krevní transfuze
U Nemocnice 1
128 20 Praha 2
e-mail: Jana.Brezinova@uhkt.cz
Sources
1. Greenberg P, Cox C, LeBeau MM, et al. International scoring system for evaluating prognosis in myelodysplastic syndromes. Blood 1997;89 : 2079–2088.
2. Greenberg PL, Tuechler H, Schanz J, et al. Revised international prognostic scoring system for myelodysplastic syndromes. Blood 2012;120 : 2454–2465.
3. Grimwade D, Walker H, Oliver F, et al. The importance of diagnostic cytogenetics on outcome in AML: analysis of 1,612 patients entered into the MRC AML 10 trial. The Medical Research Council Adult and Children‘s Leukaemia Working Parties. Blood 1998;92 : 2322–2333.
4. Grimwade D, Hills RK, Moorman AV, et al. Refinement of cytogenetic classification in acute myeloid leukemia: determination of prognostic significance of rare recurring chromosomal abnormalities among 5876 younger adult patients treated in the United Kingdom Medical Research Council trials. Blood 2010;116 : 354–365.
5. Michalová K, Zemanová Z. Molekulární cytogenetika v diagnostice nádorových onemocnění. Cas Lek Ces 2006;145 : 532–537.
6. Pinkel D, Straume T, Gray JW. Cytogenetic analysis using quantitative, high-sensitivity, fluorescence hybridization. Proc Natl Acad Sci U S A 1986;83 : 2934–2938.
7. Fan YS, Davis LM, Shows TB. Mapping small DNA sequences by fluorescence in situ hybridization directly on banded metaphase chromosomes. Proc Natl Acad Sci USA 1990;87 : 223–227.
8. Michalová K, Lemez P, Bartsch O, et al. Derivative (6)t(1;6)(q22;p21) revealed in bone marrow cells by FISH 9 months before diagnosis of acute T-lymphoblastic leukemia. Cancer Genet Cytogenet 1996;86 : 131–135.
9. Ransdorfová Š, Březinová J, Šárová I, et al. Využití interfázní fluorescenční in situ hybridizace pro analýzu CD34+ buněk v periferní krvi u nemocných s myelodysplastickými syndromy. Transfuze Hematol dnes 2016;22 : 90–96.
10. Brezinová J, Zemanová Z, Cermák J, Michalová K. Fluorescence in situ hybridization confirmation of 5q deletions in patients with hematological malignancies. Cancer Genet Cytogenet 2000;117 : 45–49.
11. Zemanova Z, Michalova K, Buryova H, et al. Involvement of deleted chromosome 5 in complex chromosomal aberrations in newly diagnosed myelodysplastic syndromes (MDS) is correlated with extremely adverse prognosis. Leukemia Res 2014;38 : 537–544.
12. Kloosterman WP, Guryev V, van Roosmalen M, et al. Chromothripsis as a mechanism driving complex de novo structural rearrangements in the germline. Hum Mol Genet 2011;20 : 1916–1924.
13. Stephens PJ, Greenman CD, Fu B, et al. Massive genomic rearrangement acquired in a single catastrophic event during cancer development. Cell 2011;144 : 27–40.
14. Brezinova J, Sarova I, Buryova H, et al. Fusion of the additional sex combs like 1 and teashirt zinc finger homeobox 2 genes resulting from ider(20q) aberration in a patient with myelodysplastic syndrome. Br J Haematol 2014;164 : 153–155.
15. MacKinnon RN, Campbell LJ. The role of dicentric chromosome formation and secondary centromere deletion in the evolution of myeloid malignancy. Genet Res Int 2011;2011 : 643628.
16. Sarova I, Brezinova J, Zemanova Z, et al. Molecular cytogenetic analysis of dicentric chromosomes in acute myeloid leukemia. Leukemia Res 2016;43 : 51–57.
Labels
Haematology Internal medicine Clinical oncology
Article was published inTransfusion and Haematology Today

2017 Issue Supplementum1-
All articles in this issue
- Genetické testy pro výběr dárce hematopoetických kmenových buněk a potransplantační monitorování
- Centrum pro vzácné choroby červené krevní řady v Ústavu hematologie a krevní transfuze
- Transfuze a imunohematologie v ÚHKT
- Standardní a velkoobjemové separace krvetvorných buněk z periferní krve za použití nového protokolu pro kontinuální sběr mononukleárních buněk Spectra Optia
- Dysfibrinogenemie a afibrinogenemie v České republice
- Molekulárně genetická vyšetření prováděná v Ústavu hematologie a krevní transfuze u pacientů s myelodysplastickým syndromem
- Funkční následky mutací v genu pro nukleofosmin u akutní myeloidní leukemie
- Současné trendy v léčbě a diagnostice chronické myeloidní leukemie
- Využití molekulárně cytogenetických technik při analýze chromozomových aberací u hematologických malignit
- Transplantace krvetvorných buněk v Ústavu hematologie a krevní transfuze (1986–2016)
- Transfusion and Haematology Today
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Dysfibrinogenemie a afibrinogenemie v České republice
- Centrum pro vzácné choroby červené krevní řady v Ústavu hematologie a krevní transfuze
- Transfuze a imunohematologie v ÚHKT
- Funkční následky mutací v genu pro nukleofosmin u akutní myeloidní leukemie
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

