-
Medical journals
- Career
První zkušenosti s preimplantačním genetickým screeningem chromozomových abnormalit pomocí komparativní genomové hybridizace na oligonukleotidových DNA mikročipech
Authors: Petr Kuglík 1,2; Jan Smetana 1,2; Darja Němcová 3; Vladimíra Vallová 1,2; Aneta Mikulášová 1,2; Renata Gaillyová 2; Vít Hubinka 3; Marek Koudelka 3
Authors‘ workplace: Laboratoř molekulární cytogenetiky, Ústav experimentální biologie, Přírodovědecká fakulta, MU, Brno 1; Oddělení lékařské genetiky FN, Brno 2; Reprofit International, Brno 3
Published in: Čas. Lék. čes. 2015; 154: 127-131
Category: Special Articles
Overview
Preimplantační genetická diagnostika představuje soubor genetických nebo cytogenetických vyšetření, pomocí kterých můžeme odhalit genetické abnormality embrya před jeho přenosem do dělohy matky. I v této oblasti se začínají využívat moderní genomické technologie založené na DNA mikročipech. Cílem práce je informovat o možnostech i prvních zkušenostech s celogenomovým vyšetřením chromozomových abnormalit v buňkách trofoektodermu pětidenních embryí pomocí techniky single cell array-CGH (komparativní genomová hybridizace na mikročipech) s využitím 60-merové oligonukleotidové platformy DNA mikročipů CytoSure 8 × 15 K Aneuploidy Array. I když byla tato platforma původně vyvinuta pro genetická vyšetření prováděná na neamplifikované DNA, současné pokroky v oblasti genomiky jedné buňky umožnily využít tuto technologii v oblasti preimplantačních genetických analýz. Metoda single cell array-CGH byla validována testováním 30 negativních a pozitivních kontrol, tj. amplifikací a následným vyšetřením DNA izolované z 5–10 buněk s normálním karyotypem a ze vzorků buněk se známými početními i strukturními abnormalitami chromozomů. Následně jsme úspěšně amplifikovali a vyšetřili vzorky 118 embryí a detekovali aneuploidie u 26,7 % a segmentální nebalancované změny u 6,8 % embryí. Naše zkušenosti potvrzují, že oligonukleotidové platformy DNA mikročipů s vysokým rozlišením jsou vhodné jak pro preimplantační genetický screening aneuploidií všech 23 párů chromozomů embrya, tak pro citlivou preimplantační diagnostiku segmentálních nebalancovaných aberací typu delecí, duplikací či amplifikací.
Klíčová slova:
chromozomové aberace – preimplantační genetická diagnostika – oligonukleotidové DNA mikročipy – array-CGHÚVOD
Preimplantační genetická diagnostika (PGD) představuje soubor genetických nebo molekulárně cytogenetických vyšetření, pomocí kterých můžeme odhalit genetické abnormality embrya před jeho přenosem do dělohy matky. Toto specializované vyšetření je založeno na nejnovějších pokrocích genetiky a asistované reprodukce. Do PGD je zahrnováno vyšetření gamet, polárních tělísek, blastomer a blastocyst (1). První klinické výsledky cyklů s PGD publikoval Handyside et al. (2), který prováděl preimplantační stanovení pohlaví embryí u párů s rizikem chorob vázaných na pohlaví. PGD je v humánní reprodukci nutně spojena s metodou in vitro fertilizace (IVF), a i když jsou vypracovány technologie pro genetické vyšetření polárních tělísek, nejčastěji se provádějí na izolovaných blastomerach 3. den po IVF a v poslední době stále častěji na buňkách trofoektodermu z pětidenních blastocystů (3).
Obecně lze říci, že po provedení PGD lze k transferu do dělohy lze vybrat pouze nepostižená embrya bez genetické zátěže. I když se pojem preimplantační genetická diagnostika používá v souvislosti s genetickým vyšetřením embryí více než 20 let, v současné době se tento soubor diagnostických metod a postupů rozlišuje na dvě skupiny. Při preimplantační genetické diagnostice (PGD) provádíme cílené genetické vyšetření, které umožňuje transfer geneticky nepostiženého embrya rodičům, u nichž byla prokázána konkrétní chromozomová aberace (např. reciproká či robertsonovská translokace) nebo konkrétní monogenně dědičná dominantní, recesivní či X-vázaná choroba vyšetřovaná nejčastěji pomocí technik PCR. Cílem PGD je tedy výběr takového embrya, které není postiženo zkoumanou genetickou vadou přítomnou u některého z rodičů. Preimplantační genetický screening (PGS) bývá prováděn u párů s normálním karyotypem a je zaměřen především na vyšetření aneuploidií chromozomů, což umožňuje zvýšit úspěšnost metod asistované reprodukce ženám vyšších věkových skupin nebo ženám s opakovanými aborty či neúspěchy v předchozích IVF cyklech (4, 5).
Klasicky byla k vyšetření chromozomových odchylek embryí používána od devadesátých let 20. století technika interfázní fluorescenční in situ hybridizace (I-FISH). Pro účely PGS byly vytvořeny speciální komerčně dostupné kity se směsí několika centromerických a lokusově specifických DNA sond určené pro vyšetření aneuploidií chromozomů v polárních tělískách a blastomerách, pomocí kterých bylo možné analyzovat početní odchylky 5–15 chromozomů v jedné buňce (6, 7). Pro tento typ diagnostiky byly nejčastěji využívány sondy hybridizující ke chromozomům 13, 16, 18, 21, 22, X a Y. Prostřednictvím metody I-FISH však nemůžeme vyšetřit všechny chromozomy buňky, což je považováno za největší nevýhodu této techniky. Aneuploidií může být postižen kterýkoliv chromozom v karyotypu. Tento cílený screening aneuploidií také neodhalí segmentální přestavby, jako jsou duplikace či delece části ramen chromozomů. Získané zkušenosti řady pracovišť i retrospektivní analýzy publikované v nedávné době prokázaly, že screeningové vyšetření 1–2 blastomer odebraných 3. den po IVF založené na technice FISH nezvyšuje úspěšnost implantace a následného otěhotnění (8, 9), což může souviset též s vysokým podílem mozaicismu a mitotické chromozomové nestability přítomné v raných embryích (10).
V současné době proto nahrazují metodu I-FISH v oblasti preimplantačních genetických analýz stále více celogenomové technologie založené na DNA mikročipech, jejichž předností je možnost vyšetřit početní změny všech 23 párů chromozomů embrya a které umožňují též odhalit též segmentální nebalancované strukturní změny všech chromozomů. Ty mohou vzniknout de novo v důsledku postzygotických mitotických poruch, nebo mohou být zděděny v důsledku meiotické segregace od rodičů – nositelů translokačních přestaveb. Techniky založené na principu komparativní genomové hybridizace na DNA mikročipech (array-CGH) mohou být proto použity jak pro účely PGD nebalancovaných chromozomových abnormalit, tak pro provádění PGS zaměřeného na vyšetření aneuploidií chromozomů. Pro širší uplatnění celogenomových analýz embryí v oblasti PGS svědčí i první povzbudivé poznatky a zkušenosti zahraničních i tuzemských pracovišť, které prokazují, že vyšetření embryí s využitím technologie single cell array-CGH vede ke zvýšení úspěšnosti implantace a následného otěhotnění, snižuje četnost abortů a přináší výrazný klinický benefit z hlediska otěhotnění i u žen vyšších věkových skupin (11–13).
VYUŽITÍ METODY ARRAY-CGH PŘI PREIMPLANTAČNÍCH GENETICKÝCH ANALÝZÁCH
Techniku array-CGH poprvé popsal v roce 1997 Solinas-Toldo et al. (14) a tato metoda se postupně se díky své vysoké citlivosti a celogenomovému přístupu stala hojně využívanou v oblasti postnatální a nádorové cytogenetické diagnostiky. Metody využívající array-CGH jsou založeny na hybridizaci nukleových kyselin k DNA mikročipům, což jsou zpravidla speciálně upravená mikroskopická skla obsahující desítky až stovky tisíc sekvencí vybraných lidských genů. V principu je tato metoda založena na srovnání počtu kopií DNA sekvencí u dvou genomů – testovaného (pacienta) a referenčního (normální). DNA každého genomu je po izolaci naznačena odlišným fluorochromem, pak jsou DNA smíchány a hybridizovány na DNA sondy imobilizované na povrchu hybridizačního skla. Výsledek hybridizace je odečítán pomocí čtecího zařízení (scanneru), který hodnotí poměr intenzit zelené a červené fluorescence, a získané údaje jsou zpracovávány speciálními programy, které provádí statistické vyhodnocení naměřených dat. Výsledkem vyšetření je grafický celogenomový profil zkoumaného vzorku, na jehož základě určujeme zisky či ztráty jednotlivých chromozomů (aneuploidie), jejich částí (segmentální změny) nebo dokonce zisky nebo ztráty jednotlivých genů.
Podle druhu použitých sond navázaných na nosném médiu rozlišujeme dva hlavní typy DNA mikročipů – tzv. BAC a oligonukleotidové mikročipy, které se liší zejména v rozlišovací schopnosti (15).
Mikročipy založené na BAC sondách (bacterial arteficial chromosomes) obsahují několik tisíc DNA sond izolovaných z klonů umělých bakteriálních chromozomů o velikosti 150–200 kb a rozlišení těchto čipů je omezeno na 50–100 kb (16). Oligonukleotidové mikročipy obsahují DNA sondy o velikosti 25–85 nukleotidů a jejich rozlišovací schopnost je tak výrazně vyšší. Vzhledem k snadnější přípravě poskytují mikročipy založené na oligonukleotidech také širší možnosti výběru různých platforem, které se liší různou hustotou pokrytí genomu (od 15 000 až po miliony DNA sond), a tím i citlivostí záchytu genetických abnormalit. Platformy oligonukleotidových DNA mikročipů založené na využití jednonukleotidových polymorfizmů (tzv. SNP čipy) umožňují navíc detekovat genetické abnormality typu uniparentální dizomie (UPD) či ztráty alelové heterozygotnosti (LOH) a stanovit genotyp buněk (17).
Se zavedením celogenomových analýz pomocí array-CGH se významným způsobem posunula rychlost i citlivost určování změn v počtu kopií DNA sekvencí. I když byly DNA mikročipy původně vyvinuty pro genetická vyšetření prováděná na neamplifikované DNA, rychlé pokroky v oblasti genomiky jedné buňky (single cell genomics) vedly v uplynulých letech k vytvoření několika komerčně dostupných platforem DNA mikročipů, které umožňují rychlé vyšetření chromozomových odchylek z jedné či několika buněk embrya a které jsou určeny pro využití v oblasti preimplantačních genetických analýz (tzv. single cell array-CGH). Molekulárně cytogenetické analýzy embryí prováděné těmito technikami bývají označovány jako preimplantační genetický screening II. generace (12). Přestože genomické metody založené na mikročipových platformách mají oproti technice FISH řadu nesporných výhod, existují i zde určitá omezení.
Obecnou limitací analýzy embryonální DNA pomocí moderní celogenomové techniky single cell array-CGH je především dostatečné množství genetického materiál vyžadovaného pro hybridizační reakci na mikočipu; množství DNA, které vyizolujeme z jedné buňky embrya činí asi 7 pg DNA, avšak mikročipové technologie vyžadují jako vstupní materiál stovky ng DNA, tj. vyžadují řádově tisícinásobnou amplifikaci. Z těchto důvodů byly postupně vypracovány účinné protokoly celogenomových amplifikací (WGA) DNA získané z jedné buňky, které jsou optimalizovány s ohledem na celkový výnos, věrohodnost amplifikované DNA ve srovnání s templátovou DNA, kompletní nezkreslené pokrytí genomu a na to, aby provedení takovéto amplifikace bylo co nejjednodušší (18). Současný trh nabízí několik metodicky odlišných kitů určených pro celogenomovou amplifikaci DNA z jedné buňky, zejména amplifikace založené na metodě PCR nebo technice MDA (multiple displacement amplification). Řada studií prokázala, že právě kvalita celogenomové amplifikace je pro úspěšné vyšetření genomu embrya pomocí techniky single cell array-CGH či jiných genomických technik klíčovým faktorem a artefakty, které při ní vznikají např. v důsledku různého obsahu GC párů v genomu, či při amplifikaci buněk v nacházejících se v S-fázi buněčného cyklu, představují největší úskalí těchto metod (5,19).
K dalším stimulům, které umožnily současný rozmach využití mikročipových technologií pro účely PGD, patří vyšetření buněk odebíraných ve vyšších embryonálních stádiích a zdokonalení technik kryokonzervace embryí metodou vitrifikace. V poslední době bylo prokázáno, že biopsie blastomer z třídenního embrya může významně snížit implantační potenciál embrya (20), a proto stále více pracovišť namísto vyšetření 1-2 blastomer z 3 denního embrya přechází na odběry a následné genetické vyšetření 5–10 buněk trofoektodermu z blastocysty odebíraných 5. až 6. den embryonálního vývoje. Vyšetření prováděné z většího množství materiálu buněk poskytuje nejenom více DNA, ale vede též ke zpřesnění a zvýšení účinnosti diagnostiky a to zejména s ohledem na přítomnost blastocyst s mozaikami. Zdokonalení vitrifikačních metod umožňuje efektivní uchování bioptovaných blastocyst a jejich transfer v následujícím cyklu, což dovoluje provádět genetická vyšetření v průběhu delšího časového intervalu.
VYUŽITÍ OLIGONUKLEOTIDOVÝCH DNA ČIPŮ PRO ÚČELY PGS/PGD
V případě použití metody single cell array-CGH pro účely PGS/PGD jsou v současné době na mnoha pracovištích úspěšně používány zejména DNA mikročipy založené na BAC sondách. Tyto mikročipy jsou validované jak pro detekce aneuploidií, tak pro vyšetřování nebalancovaných translokací a jejich výhodou je menší citlivost k artefaktům vznikajícím v důsledku celogenomové amplifikace a zkrácená doba laboratorního protokolu na 24 h (21, 22). Avšak v posledních letech se pozornost obrací též na mikročipy oligonukleotidové, které mají hustší pokrytí genomu jednotlivými sondami, a tedy i vyšší citlivost a reprodukovatelnost (23, 24). Hellani el al. v roce 2008 (24) prezentovali narození prvního dítěte po provedení celogenomového PGS s využitím oligonukleotidových DNA mikročipů obsahujících 44 000 DNA sond. Výsledky dalších studií prokázaly, že po amplifikaci DNA je možné pomocí oligonukleotidových DNA mikročipů v rámci preimplantačních genetických analýz spolehlivě odhalit nejenom aneuploidie jednotlivých chromozomů, ale i drobné mikrodelece a mikroduplikace o velikosti 1,2 Mb (25).
Na našem pracovišti byla ve spolupráci s firmou Oxford Gene Technology zavedena a použita metoda preimplantačního genetického screeningu pomocí nové generace oligonukleotidových DNA mikročipů Single Cell Aneuploidy Array 8 × 15 K, které byly speciálně vyvinuty a zvalidovány pro detekci aneuploidií všech chromozomů získaných amplifikací DNA z jedné buňky. Tyto čipy obsahují 15 000 DNA sond o velikosti 60 oligonukleotidů a umožňují analyzovat osm vzorků na jednom sklíčku, co snižuje náklady na jedno vyšetření. Aplikace speciálního programu dovoluje jednoduchou a přehlednou interpretaci získaných výsledků, tj. hodnocení počtu kopií jednotlivých chromozomů. Pro provádění PGD a detekci aneuploidií byly tyto DNA mikročipy nejprve validovány samotným výrobcem, a to porovnáním dat získaných na uvedené oligonukleotidové platformě s výsledky získanými pomocí komerčně používané BAC platformy; úspěšnost dosáhla 99,5 % (Oxford Gene Technology, Oxford, Velká Británie).
Celý proces přípravy array-CGH byl navíc validován i v naší laboratoři dle mezinárodních doporučení (26), a to v několika krocích. Nejprve jsme v naší laboratoři optimalizovali protokol založený na „single cell“ celogenomové amplifikaci, který dokáže z 5–25 pg DNA dosáhnout výtěžku v řádu mikrogramů (typicky 2,5–4 µg DNA). Pro „single cell“ amplifikaci jsme použili PicoPLEX WGA Kit (Rubicon Genomic), kontrola DNA vzorků i reference (Human Genomic DNA: Male; Promega Inc) byla provedena na fluorimetru Qubit (Life Technologies) a délka amplifikovaných fragmentů byla ověřena v 2% agarovém gelu. Amplifikovaná DNA byla poté naznačena fluorescenčními barvami (Alexa 5, Alexa 3) dle standardního protokolu (Life Technologies) a hybridizována. Analýza dat byla provedena pomocí software CytoSure 4.6.2 a speciálního vyhodnocovacího algoritmu „PGD analysis“ (Oxford Gene Technology). Při těchto validačních experimentech jsme rovněž optimalizovali parametry kontroly kvality DNA mikročipu (QC metrics) pro preimplantační genetické analýzy.
Účinnost celogenomové amplifikace i samotná platforma čipů byly následně ověřovány testováním 10 negativních a 12 pozitivních kontrol, tj. amplifikací a následným vyšetřením DNA izolované z 5–10 buněk s normálním karyotypem či naředěné DNA získané ze vzorků buněk s nadpočetným chromozomem 21 a dalšími trizomiemi (tab. 1). Ve druhém kroku byla testována amplifikace DNA i oligonukleotidové mikročipy na vzorcích blastomer a blastocyst se známými početními chromozomovými změnami stanovenými metodou FISH. V posledním kroku byla prověřována rozlišovací schopnost námi vybraných mikročipů prostřednictvím analýz osmi vzorků s různě velkými potvrzenými strukturními nebalancovanými chromozomovými změnami (duplikace, delece). Pro tyto účely byly rovněž testovány oligonukleotidové mikročipy s hustším pokrytím DNA sond (8 × 60 K, Oxford Gene Technology) a (8 × 60 K, Agilent).
Table 1. Karyotyp vzorků použitých po „single cell amplifikaci“ jako negativní i pozitivní kontroly pro validaci oligonukleotidových DNA mikročipů Single Cell Aneuploidy Array 8 × 15 K (Oxford Gene Technology) 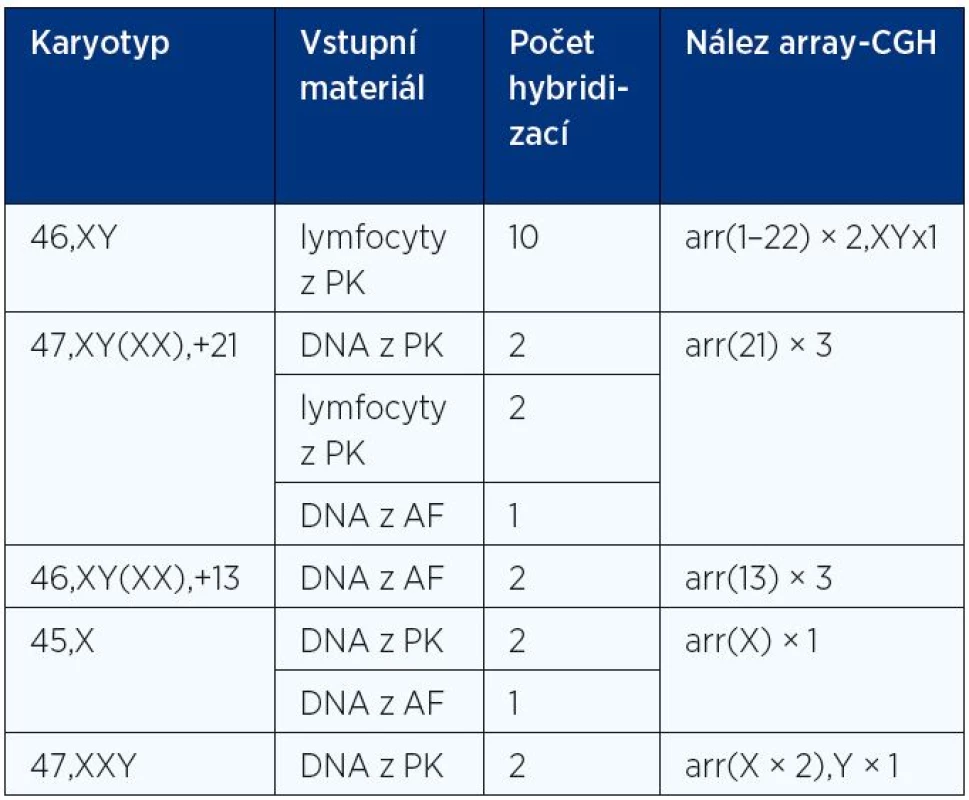
PK – periferní krev, AF – amniová tekutina V našich validačních experimentech jsme byli schopni s těmito platformami DNA mikročipů potvrdit všechny testované aneuploidie a prokázat mikrodeleci na chromozomu 22q11 o velikosti asi 2 Mb. Součástí zavádění této technologie byly i mezilaboratorní kontroly založené na srovnávání účinnosti záchytu aneuploidií pomocí BAC a oligonukleotidových DNA mikročipů.
Během roku 2013 jsme následně úspěšně amplifikovali a pomocí oligonukleotidových DNA vyšetřili vzorky bioptované z trofoektodermu 118 embryí. Aneuploidie jsme přitom prokázali u 26,7% a segmentální nebalancované změny typu delecí či duplikací u 6,8 % embryí. Mezi nejčastější pozorované chromozomové abnormality patřily aneuploidie chromozomů 8, 13, 18 a 21 a segmentální abnormality v oblastech 5q, 8p, 14q a 16p (obr. 1).
Image 1. Trizomie chromozomů 3 a 14 a monozomie chromozomu 19 detekovaná pomocí techniky array-CGH s využitím oligonukleotidových DNA mikročipů Single Cell Aneuploidy Array 8 × 15 K (Oxford Gene Technology) u buněk z blastocysty 
Naše zkušenosti i první výsledky tedy potvrzují, že oligonukleotidové typy DNA mikročipů lze velmi dobře využít nejen pro spolehlivý screening aneuploidií všech 23 párů chromozomů, ale i pro citlivou diagnostiku mikrodelecí, mikroduplikací a dalších segmentálních nebalancovaných strukturních chromozomových změn vyskytujících se u embryí. Tyto moderní celogenomové analýzy umožňují rozšířit spektrum vyšetřovaných chromozomových abnormalit v rámci PGS, a tím i zlepšit výběr optimálních embryí pro účely IVF (27, 28). Při analýze 28 transferů provedených po PGS pomocí DNA mikročipů bylo u 14 pacientek prokázáno biochemické těhotenství. I po provedení PGS/PGD pomocí oligonukleotidových DNA mikročipů s vysokým rozlišením je však doporučováno ověření nálezů pomocí metod prenatální genetické diagnostiky.
ZÁVĚR
Závěrem můžeme shrnout, že diagnostika genetických abnormalit na genomické a molekulární úrovni je v současné době nezbytným předpokladem ke správné léčbě již u řady diagnóz. Lze předpokládat, že i komplexní diagnostika a léčba neplodných párů bude stále více ovlivněna rozvojem a využitím moderních genomických technologií, a to nejen využitím DNA mikročipů, ale v budoucnu i sekvenováním nové generace (NGS), a to i přes řadu nesporných problémů, které s sebou přináší nutnost amplifikace vstupního materiálu. Rozvoj a aplikace těchto moderních genomických metod však bude vyžadovat co nejužší spolupráci mezi obory asistované reprodukce a klinické genetiky, a to jak na úrovni lékařů, tak na úrovni vysoce erudovaných laboratorních pracovníků, zejména embryologů a genetiků.
Zkratky
I-FISH interfázní fluorescenční in situ hybridizace
NGS sekvenováním nové generace
PGD preimplantační genetická diagnostika
PGS preimplantační genetický screening
Práce byla podpořena projekty OP VK CZ.1.07/2.3.00/20.0183 a OP VK CZ.1.07/2.4.00/31.0155.
Konflikt zájmů: žádný.
ADRESA PRO KORESPONDENCI:
doc. RNDr. Petr Kuglík, CSc.
Laboratoř molekulární cytogenetiky, Ústav experimentální biologie
Přírodovědecká fakulta MU
Kotlářská 2, 611 37 Brno
e-mail: kugl@sci.muni.cz
Sources
1. Harper JC, SenGupta SB. Preimplantation genetic diagnosis: State of the art 2011. Hum Genet 2012; 131 : 175–186.
2. Handyside A. Sex and the single cell. New Sci 1990; 126 (1713): 34–35.
3. Kokkali G, Traeger-Synodinos J, Vrettou C, et al. Blastocyst biopsy versus cleavage stage biopsy and blastocyst transfer for preimplantation genetic diagnosis of beta-thalassaemia: a pilot study. Hum Reprod 2007; 22 : 1443–1449.
4. Fragouli E. Preimplantation genetic diagnosis: present and future. J Assist Reprod Genet 2007; 24(6): 201–207.
5. Van der Aa N, Esteki MZ, Vermeesch JR, et al. Preimplantation genetic diagnosis quided by single-cell genomics. Genome Medicine 2013; 5(8): 71.
6. Munné S, Lee A, Rosenwaks Z, et al. Diagnosis of major chromosome aneuploidies in human preimplantation embryos. Hum Reprod 1993; 8(12): 2185–2191.
7. Baart EB, Van den Berg I, Martini E, et al. FISH analysis of 15 chromosomes in human day 4 and 5 preimplantation embryos: the added value of extended aneuploidy detection. Prenat Diagn 2007; 27(1): 55–63.
8. Harper J, Coonen E, De Rycke M, et al. What next for preimplantation genetic screening (PGS)? A position statement from the ESHRE PGD Consortium steering committee. Hum. Reproduction 2010; 24(4): 821–833.
9. Mastenbroek S, Twisk M, van der Veen F, Repping S. Preimplantation genetic screening: a systematic review and meta-analysis of RCTs. Hum Reprod Update 2011; 17(4): 454–466.
10. Vanneste E, Voet T, Le Caignet C, et al. Chromosome instability is common in human cleavage-stage embryos. Nature Medicine 2009; 19(5): 577–583.
11. Schoolcraft WB, Fragouli E, Stevens J, et al. Clinical application of comprehensive chromosomal screening at the blastocyst stage. Fertil Steril 2010; 94(5): 1700–1706.
12. Harton GL, Munne S, Surrey M, et al. Diminished effects of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertil Steril 2013; 100 (6): 1695–1703.
13. Slamova I, Horinova V, Okenkova K, et al. Preimplantation genetic screening with used 24sure microarrays, our results and the success of in vitro fertilization cycles. Eur J of Hum Genet 2013; 21(Suppl 2): 441–442.
14. Solinas-Toldo S, Lampel S, Stilgenbauer S, et al. Matrix-based comparative genomic hybridization: biochips to screen for genomic imbalances. Genes Chromosome Cancer 1997; 20 : 399–407.
15. Pinkel D, Segraves R, Sudar D, et al. High resolution analysis of DNA copy number variation using comparative genomic hybridization to microarrays. Nat Genet 1998; 20(2): 207–211.
16. Oostlander AE, Meijer GA, Ylstra B. Microarray-based comparative genomic hybridization and its applications in human genetics. Clin Genet 2004; 66(6): 488–495.
17. Vanneste E, Bittman L, Van der Aa N, et al. New array approaches to explore single cells genomes. Front Genet 2012; 3(44): 1–6.
18. Hughes S, Arneson N, Done S, Squire J. The use of whole genome amplification in the study of human disease. Prog Biophys Mol Biol 2005; 88(1): 173–189.
19. Van der Aa N, Cheng J, Mateiu L. Genome-wide copy numbere profiling of single cells in S-phase reveals DNA-replication domains. Nucl Acid Res 2013; 41(6): 1–13.
20. Scott RT, Upham KM, Forman EJ, et al. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trials. Fertil Steril 2013; 100(3): 624–630.
21. Harper JC, Harton G. The use of arrays in preimplantion genetic diagnosis and screening. Fertil Steril 2010; 94(4): 1173–1177.
22. Fiorentino F, Spizzichino L, Bono S, et al. PGD for reciprocal and Robertsonian translocations using array comparative genomic hybridization. Hum Reprod 2011; 26 : 1925–1935.
23. Chang L-J, Chen S-U, Tsai Y-Y, et al. An update of preimplantation genetic diagnosis in gene disease, chromosomal translocation, and aneuploidy screening. Clin Exp Reprod Med 2011; 38(3): 126–134.
24. Hellani A, Abu-Amero K, Azouri J, El-Akoum S. Successful pregnancies after application of array-comparative genomic hybridization in PGS-aneuploidy screening. Reprod Biomed Online 2008; 17(6): 841–847.
25. Bi W, Breman A, Shaw CA, et al. Detection of ≥ 1 Mb microdeletions and microduplications in a single cell using custom oligonucleotide arrays. Prenat Diagn 2012; 32(1): 10–20.
26. Vermeesch JR, Brady PD, Sanlaville D, et al. Genome-wide arrays: quality criteria and platforms to be used in routine diagnostics. Hum Mutat 2012; 33(6): 906–915.
27. Liang L, Wang CT, Sun X. Identification of chromosomal errors in human preimplantation embryos with oligonucleotide DNA microarray. PLoS One 2013; 8(4): 1–11
28. Adler A, Lee LH, McCulloh DH. Blastocyt culture selects for euploid embryos: comparison of blastomere and trophoectoderm biopsies. Reprod Biomed Online 2013, S1472-6483(13)00636-6. doi: 10.1016/j.rbmo.2013.11.018.
Labels
Addictology Allergology and clinical immunology Angiology Audiology Clinical biochemistry Dermatology & STDs Paediatric gastroenterology Paediatric surgery Paediatric cardiology Paediatric neurology Paediatric ENT Paediatric psychiatry Paediatric rheumatology Diabetology Pharmacy Vascular surgery Pain management Dental Hygienist
Article was published inJournal of Czech Physicians

-
All articles in this issue
- Kmenové buňky v ortopedii
- Histologické hodnocení vlivu in vivo aplikace biomateriálů na hojení chrupavky, kosti a kůže
-
Spiritualita a etika v psychosomatické medicíně –
biopsychosociospirituální vztahy - miRNA jako nový ukazatel u diabetes mellitus a u rozvoje karcinomu pankreatu
- První zkušenosti s preimplantačním genetickým screeningem chromozomových abnormalit pomocí komparativní genomové hybridizace na oligonukleotidových DNA mikročipech
- Připravenost izraelského zdravotnictví na krizové situace
- Historie výroby inzulinu v Československu
- Zásluhy prof. Josefa Hally o rozvoj pražské lékařské fakulty
- Journal of Czech Physicians
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Historie výroby inzulinu v Československu
-
Spiritualita a etika v psychosomatické medicíně –
biopsychosociospirituální vztahy - Kmenové buňky v ortopedii
- Připravenost izraelského zdravotnictví na krizové situace
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

