-
Medical journals
- Career
Nejčastěji využívané metodiky k analýze DNA metylačních změn
Authors: Marcela Chmelařová; Vladimír Palička
Authors‘ workplace: Univerzita Karlova v Praze, Lékařská fakulta a FN Hradec Králové, Ústav klinické biochemie a diagnostiky
Published in: Čas. Lék. čes. 2011; 150: 442-445
Category: Review Articles
Overview
Vzhledem k významnosti vztahu mezi stupněm metylace DNA a genovou aktivitou, zejména u nádorových onemocnění, byla popsána celá řada technik sledujících metylací DNA. Cílem této práce bylo zpracovat přehled o metodikách, které mohou být využívány k monitorování DNA metylačních změn. V práci je kladen důraz na monitorování změn metylace DNA v oblasti CpG ostrůvků promotorových oblastí genů. Přehled popisuje výhody, nevýhody a potenciální využití jednotlivých metod.
Klíčová slova:
metylace DNA, modifikace hydrogensiřičitanem, MSP, MS-MLPA.ÚVOD
Při mnoha procesech v organismu, zvláště pak v procesu karcinogeneze často dochází k epigenetickým změnám. Epigenetické změny jsou změny v genové expresi, při nichž nedochází ke změnám kódujících sekvencí bází v polynukleotidovém řetězci DNA. Zahrnují DNA metylaci, modifikaci histonů, přestavbu chromatinu a posttranskripční genovou regulaci pomocí miRNA. Nejčastěji studovanou epigenetickou alterací je DNA metylace.
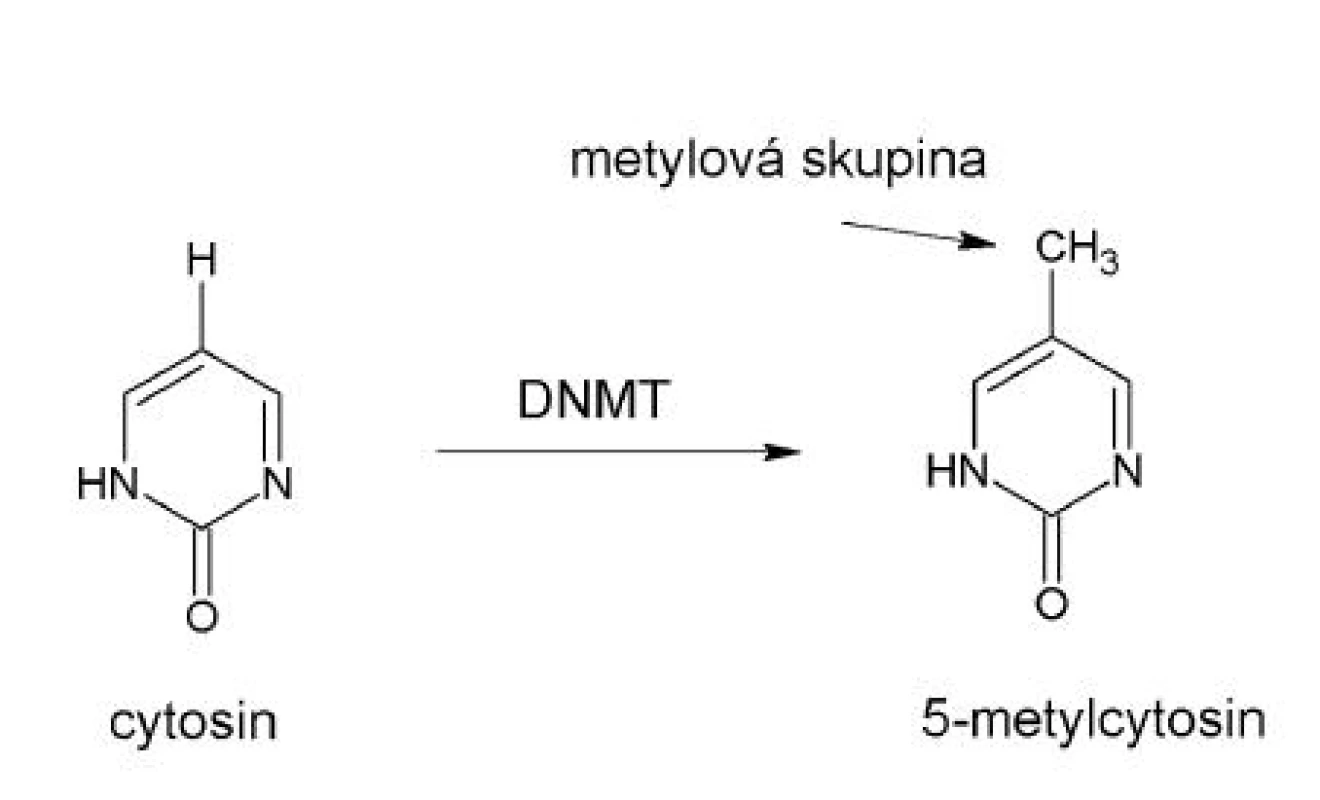
Metylace DNA je kovalentní modifikací DNA, která je katalyzována enzymy skupiny DNA metyltransferáz (DNMTs). Při metylaci dochází k vazbě metylové skupiny na 5ę uhlík cytosinu za vzniku 5-metylcytosinu (obr. 1). Děje se tak přenosem metylu z S-adenosyl-methioninu na C5 pozici cytosinu v dinukleotidovém kontextu CpG. Distribuce 5-metylcytosinu v genomu je specifická pro každý buněčný typ a je ustavena během embryonálního vývoje (1). Metylace DNA není ovšem stálá, v organismu může docházet k její zesílení metylací de novo. Bohužel o mechanismu metylace de novo v normálních a nádorových buňkách není mnoho známo. K hromadění změn v metylaci DNA může docházet v průběhu několika let před vypuknutím maligního bujení, proto by tyto změny mohly být potenciálně používány v časné diagnostice nádorových onemocnění.
DNA metylace hraje nepostradatelnou roli v kontrole genové aktivity a je důležitá pro architekturu buněčného jádra (2), podílí se na regulaci transkripce, stabilitě chromozomů, genomickém imprintingu a inaktivaci X-chromozomu (3), dále má důležitou roli ve vývoji a chrání genom před transpozony. Stupeň metylace příslušného úseku DNA ovlivňuje aktivitu chromatinu. Transkripčně inaktivní chromatin je charakterizován přítomností metylovaného cytosinu, přičemž tato konfigurace brání transkripci. Metylace promotorových částí DNA může způsobovat transkripční inaktivaci tumorsupresorových genů, a to zejména tím, že snižuje vazebnou afinitu transkripčních faktorů, které interagují s DNA ve specifických sekvencích, což často vede k progresi tumorů či rezistenci k chemoterapeutikům.
V současné době známe několik genově specifických metod (tab. 1), které mohou být využity k monitorování změn v metylaci DNA. Při výběru metody záleží zejména na cíli studie. Důležité je, zda sledujeme celkovou hypermetylaci DNA, kde je metodou první volby HPLC (high-performance liquid chromatography) nebo pouze metylaci určitých částí DNA. V tomto článku je kladen důraz na monitorování změn v metylaci v oblasti CpG ostrůvků.
Table 1. Nejčastěji používané genově-specifické DNA metylační techniky 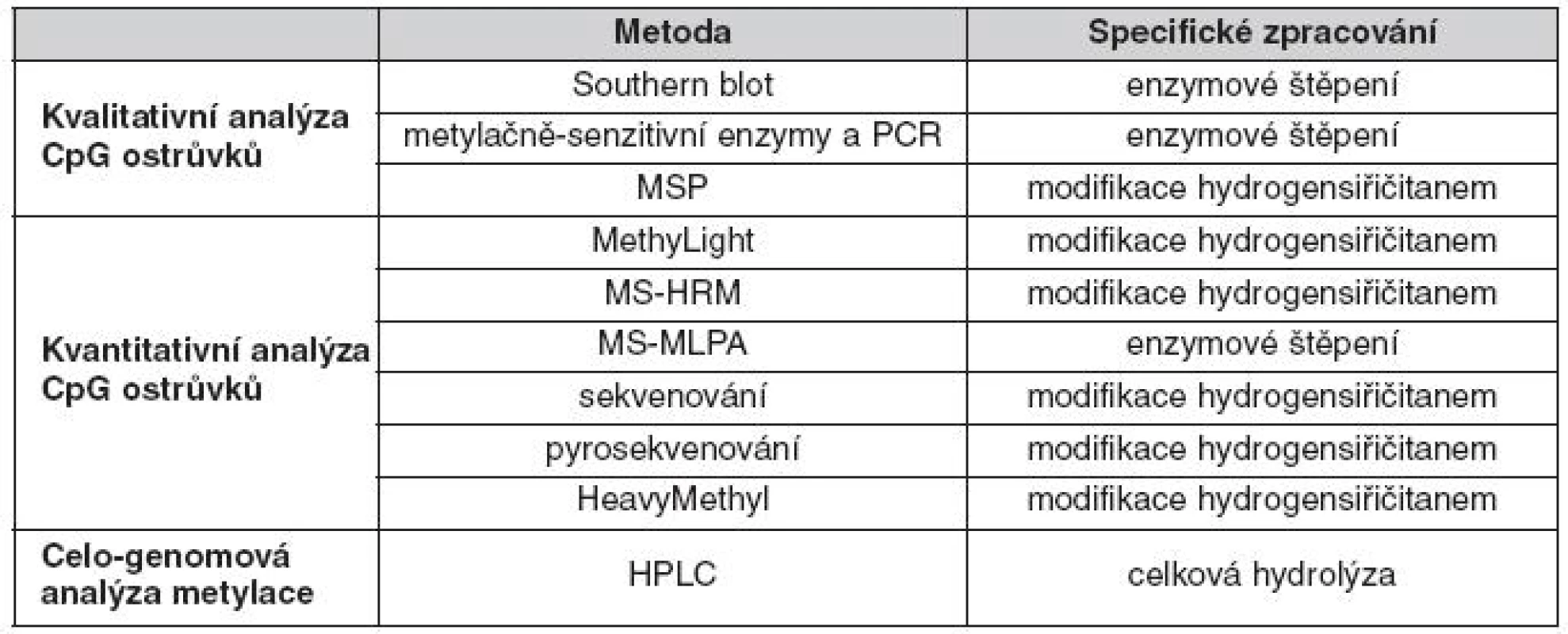
METODY ZALOŽENÉ NA MODIFIKACI DNA HYDROGENSIŘIČITANEM
U metod založených na modifikaci DNA hydrogensiřičitanem (bisulfite conversion) je prvním krokem chemická přeměna analyzované DNA. Při modifikaci dochází k přeměně cytosinu na uracyl pomocí hydrogensiřičitanu sodného na jednovlákné DNA. Dochází k záměně všech cytosinů na uracyly, pouze metylované cytosiny (5-metylcytosin) jsou k záměně rezistentní a zůstávají jako cytosiny (4). Při procesu přeměny nemetylovaného cytosinu na uracyl je prvním krokem denaturace DNA zahřátím v alkalickém prostředí. Chemický průběh modifikace DNA zahrnuje sulfonaci a hydrolytickou deaminaci, kdy vzniká meziprodukt (uracyl sulphonát), který musí být převeden na uracyl alkalickou desulfonací (obr. 2). Chemická modifikace takto vytvoří odlišnosti v sekvenci mezi metylovanou a nemetylovanou DNA.
Image 2. Chemický průběh modifikace cytosinu na uracil 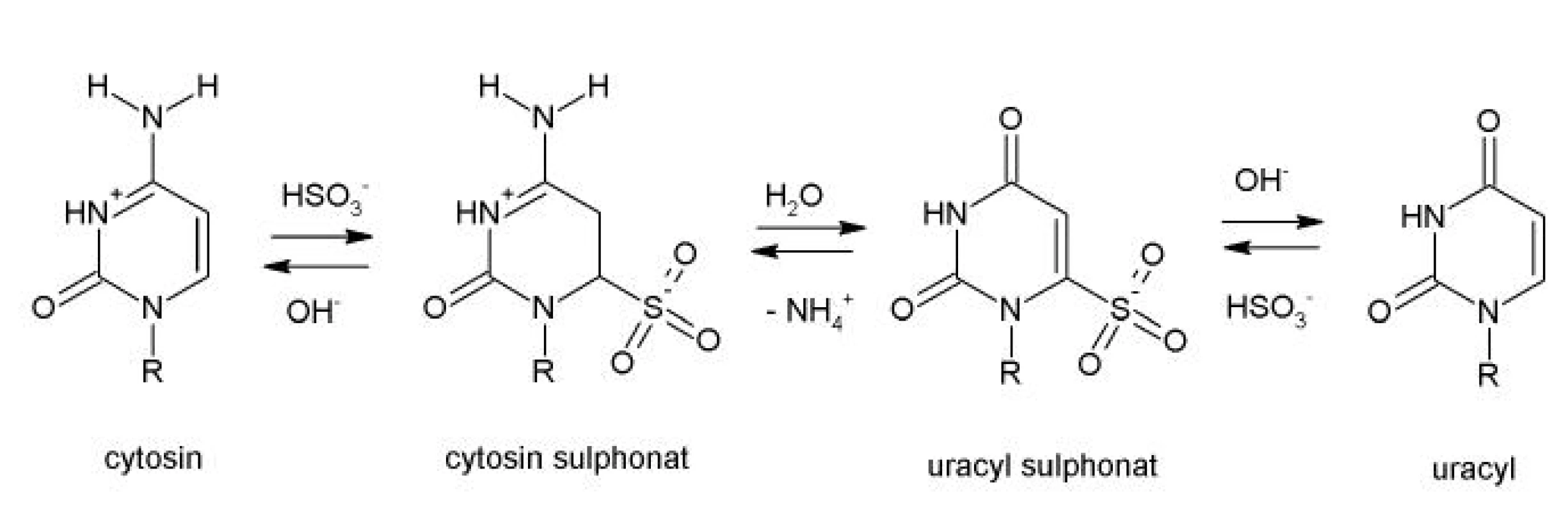
Modifikace DNA hydrogensiřičitanem je poměrně levná a jednoduchá, nevýhodou je ovšem časté poškození DNA, které se projeví zejména u málo kvalitní DNA izolované z parafínových bločků.
Metylačně specifická PCR (MSP)
MSP je specifickou a senzitivní metodou využívanou zejména ke sledování abnormálních genových metylací ve vybraných genových sekvencích, zejména CpG ostrůvcích (4). Její nespornou výhodou je proveditelnost při nízkých koncentracích DNA a relativně nízká cena. Nevýhodou této metody je ovšem falešná pozitivita některých výsledků.
U MSP hydrogensiřičitanovou modifikaci následuje amplifikace pomocí primerů specifických pro metylovanou (nemetylovanou) DNA. Ideálně jsou navrhnuty tři sety primerů přisedající k DNA na základě odlišností v sekvenci. Jeden set primerů (U) přisedá k nemetylované DNA, která podlehla chemické modifikaci. Druhý set primerů (M) přisedá k metylované DNA, která také podlehla chemické modifikaci. A třetí set primerů (W) přisedá ke každé DNA (metylované nebo nemetylované), která nepodlehla chemické modifikaci. Tento set primerů by měl sloužit jako kontrola efektivity chemické modifikace. Pravidla pro navrhování standardních primerů platí i pro navrhování primerů pro MSP. Primery by měly být přibližně 20–21 bp dlouhé a měly by mít podobnou disociační teplotu. Produkt MSP reakce s každým setem primerů by měl mít velikost 100–200 bp. K zabránění tvorby dimerů primerů by primery neměly být komplementární, a to zejména na 3’ konci.
K vlastní amplifikaci se doporučuje využití „hot start“ PCR. Během reakce dojde k amplifikaci produktu. Pokud je vzorek DNA v místě CpG ostrůvků původně nemetylovaný, reakční produkt je detekovatelný pouze s využitím setu primerů U, primery navržené jako komplementární k nemetylované DNA sekvenci, v tomto případě pak žádný produkt nebude vznikat v reakci se setem primerů M, primery navržené jako komplementární k metylované DNA sekvenci. Naopak produkt MSP s použitím M primerů bude vznikat při reakci s původně metylovanou DNA. Nemodifikovaná DNA bude poskytovat produkt pouze v případě využití setu primerů W. Jako u každé PCR je nutné dbát na zabránění kontaminace PCR produktem (oddělené laboratoře na přípravu amplifikační směsi a detekci PCR produktu). Vyhodnocení PCR reakce může být následně kvalitativní pomocí elektroforézy (obr. 3) nebo kvantitativní, založené na detekci změn fluorescence (MethyLight, Syber green based, HeavyMethyl).
Image 3. Elektroforetické vyhodnocení MSP Legenda: Je-li DNA nemetylovaná, vzniká PCR produkt po reakci se setem primerů U, který je specifický pro nemetylovanou DNA. Je-li DNA metylovaná, vzniká PCR produkt po reakci se setem primerů M, který je specifický pro metylovanou DNA. 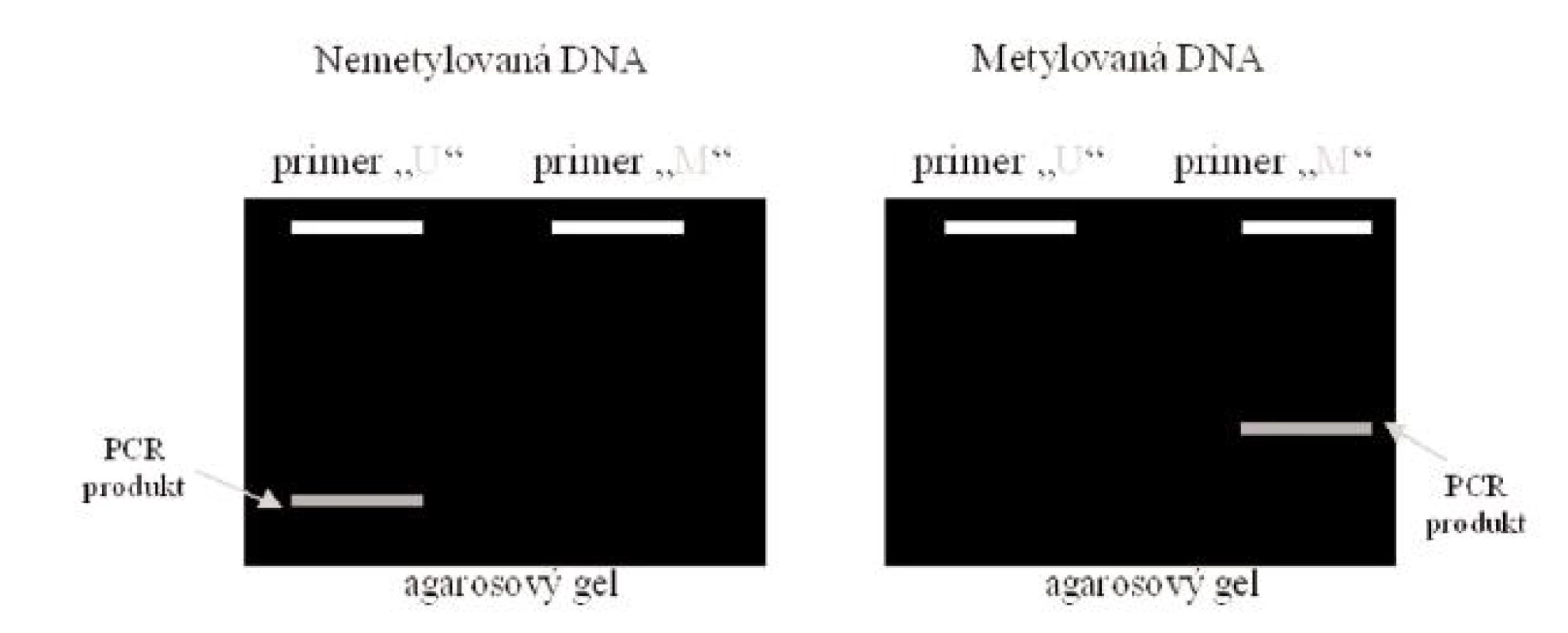
Sekvenování
Jednou z největších předností sekvenování amplifikovaných produktů je cenná kontrola hydrogensiřičitanové přeměny.
Pro určení přesné sekvence nukleotidů v úseku DNA se využívají zejména dvě metody – Sangerova a Maxam-Gilbertova. Sekvenování amplifikovaných produktů je nejčastěji provedeno Sangerovou metodou. Sangerova metoda využívá speciální vlastnosti dideoxinukleotidů, které nemají na 3’ uhlíku ribózy OH skupinu. Dideoxynukleotid je schopen se začlenit do replikující se DNA, ale následně zastaví elongaci řetězce. Označíme-li každý dideoxinukleotid jinou fluorescenční barvou, bude v reakční směsi vznikat směs různě dlouhých a různě barevně značených sekvencí. Výsledná sekvence je pak určena kapilární elektroforézou. Sangerova metoda je použitelná zejména k sekvenování krátkých sekvencí jednovlákné DNA.
V současné době je v souvislosti se sekvenací často zmiňováno pyrosekvenování. Jedná se o velice senzitivní technologii, která může být použita i ke kvantifikaci sekvenčních variací. Pyrosekvenování může detekovat a kvantifikovat i malé změny ve stupni metylace. Pyrosekvenování je založeno na detekci emitovaného světla během syntézy komplementárniho vlákna exonukleasově-deficientní DNA polymerázou. Technologie pyrosekvenování již byla použita pro korelaci metylace DNA nádorových buněk a genové exprese, k měření buněčné odpovědi na léčbu látkami, které zabraňují DNA metylaci, k posouzení změn v metylaci DNA při vzniku nádorů a genetickém imprintingu (5).
MS-HRM (methylation specific high-resolution melting analysis)
HRM je metoda založená na změnách teploty tání DNA v roztoku. HRM je realizována postupným zahříváním dvouvlákného amplikonu DNA z přibližně 50 °C na 95 °C. V určitém bodě tohoto procesu je dosaženo teploty tání amplikonu a dvě vlákna DNA se od sebe oddělí.
Nejčastěji se HRM provádí v real-time uspořádání. Toho je dosaženo využitím fluorescenčního barviva. Využívána jsou interkalační barviva, která se specificky vážou na dvouvláknou DNA, a pokud jsou vázána fluoreskují jasně. Při absenci dvoušroubovice DNA se nemohou vázat a fluoreskují na nízké úrovni. Na začátku HRM analýzy je tedy vysoká úroveň fluorescence ve vzorku, protože jsou zde přítomny miliardy kopií dvouvlákného amplikonu. Během zahřívání vzorku přítomnost dvouvlákné DNA klesá, a proto se fluorescence snižuje.
Principem MS-HRM je, že DNA ovlivněná hydrogensiřičitanem má v případě metylovaného templátu více cytosinů než DNA, která byla původně nemetylovaná (cytosiny se při modifikaci přeměnily na uracyly), proto se změní i křivka teploty tání takovéto DNA. PCR produkty pocházející z templátu, který byl původně nemetylovaný, budou mít nižší bod tání než ty, které pocházejí z metylovaného templátu. HRM nabízí také možnost určení podílu metylace v daném vzorku porovnáním s kalibrační křivkou, která vzniká mícháním různých poměrů univerzálně metylované a nemetylovaný DNA dohromady. Při analýze je porovnávána teplota tání univerzálně metylované a nemetylované DNA s vyšetřovaným vzorkem.
HRM je senzitivní a specifická (6), relativně jednoduchá a levná metoda. Předností této metody je analýza všech CpG ostrůvků amplikonu.
VYUŽITÍ RESTRIKČNÍCH ENZYMŮ
Metylačně specifické restrikční endonukleázy jsou enzymy specificky využívané k DNA metylační analýze. Většina z nich je inhibována metylací jejich rozpoznávacího místa, kde specificky štěpí DNA.
Mezi nejčastěji užívané restrikční endonukleázy patří izoschizomery HpaII, MspI, které rozeznávají sekvenci CCGG. Každý z těchto emzymů je blokován metylací specifického místa. HpaII je blokován metylací jednoho z cytosinu uvedené sekvence, zatímco MspI je blokován pouze metylací vnějšího cytosinu (7).
Vystavíme-li sledovanou DNA působení restrikčních endonukleáz, dojde v případě, kdy není metylována, k jejímu rozštěpení. Následně můžeme takto naštěpenou DNA analyzovat pomocí elektroforézy a Southern blotu. Southern blot byl použit jako jedna z prvních metod pro analýzu metylace DNA. Použijeme-li po naštěpení DNA PCR reakci, bude vznikat PCR produkt pouze v případě, kdy DNA je metylovaná (nedochází k rozštěpení). Využití kombinace restrikční endonukleázy s PCR se jeví jako senzitivnější metoda než Southern blot.
Využití restrikčních enzymů, které rozpoznávají metylované sekvence od nemetylovaných, může být aplikováno pouze u sekvencí, které obsahují jejich rozpoznávací sekvence, v čemž lze vidět určitou limitaci této metody ve srovnání s metodami využívajícími modifikaci DNA hydrogensiřičitanem.
Nedávná studie popisuje „jednokrokovou metylační analýzu“ (8). Tato metoda je založena na kombinaci metylačně senzitivní endonukleázy a kvantitativní real-time PCR v jedné reakci. Prvním krokem je DNA štěpení, následované inaktivací endonukleázy a nakonec real-time PCR.
MS-MLPA
MS-MLPA (methylation specific multiplex ligation-dependent probe amplification) je v současné době široce používaná metoda pro detekci epigenetických změn. MS-MLPA představuje semikvantitativní metodu pro sledování metylačních změn DNA. MS-MLPA je velmi podobná klasické MLPA, kdy ligace proby je kombinována se štěpením komplexu DNA-proba metylačně specifickou endonukleázou (9). Nejčastěji využívaným restrikčním enzymem je Hha1, tento enzym rozpoznává a specificky štěpí pouze nemetylovanou DNA sekvenci GCGC.
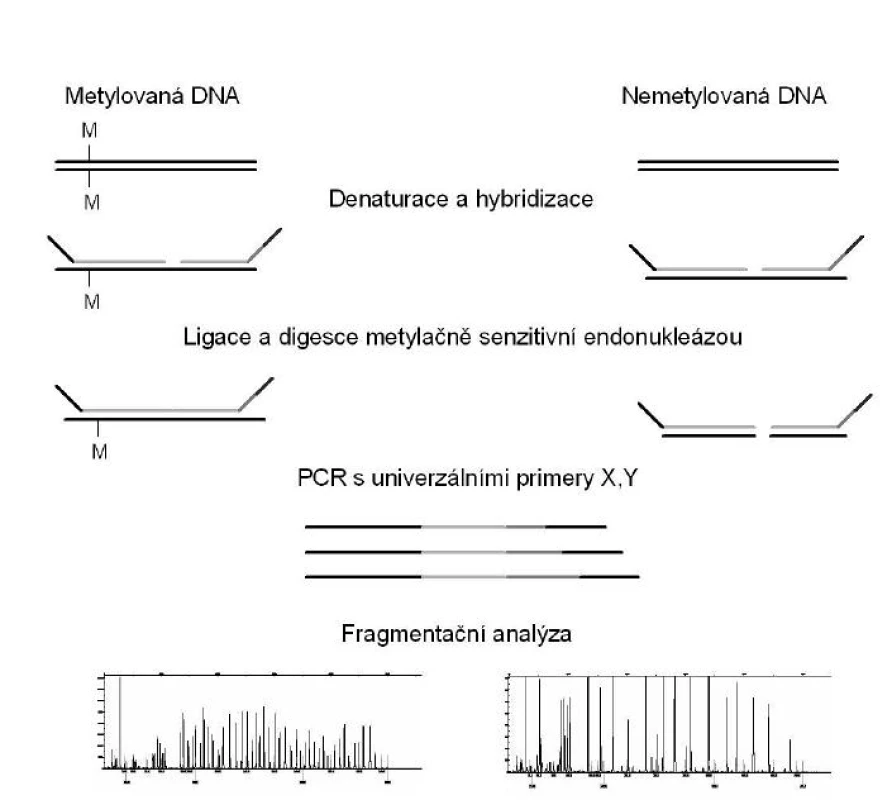
MS-MLPA lze provést v pěti základních krocích (obr. 4). Prvním krokem je denaturace DNA a hybridizace specifických MLPA sond. Následuje ligace a digesce s využitím metylačně specifické endonukleázy. Třetím krokem je PCR s užitím univerzálních primerů, při které dochází k exponenciální amplifikaci pouze ligovaných a neštěpených prob. Čtvrtým krokem je oddělení amplifikovaných produktů pomocí kapilární elektroforézy. Jelikož štěpené proby (hybridované s nemetylovanou DNA) nejsou během PCR amplifikovány, nebudou tedy při kapilární elektroforéze dávat žádný signál. Naopak, pokud byl vzorek metylovaný, hybridizovaná proba je chráněna před digescí, bude vznikat amplifikační produkt a následně bude vznikat pík při kapilární elektroforéze. Posledním, pátým krokem je analýza dat.
MS-MLPA je metoda, která je velmi citlivá a může být využita již při malých koncentracích izolované DNA. Nespornou výhodou je i její použití u analýzy DNA izolované z parafínových bločků, která často bývá málo kvalitní. Další výhodou je analýza několika sekvencí v jedné reakci.
MIKROČIPY
Mikročipy jsou používány jak k celkové analýze DNA, tak k detekci metylace v promotorových oblastech genů. Principem mikročipů je imobilizace fragmentů jednovlákné DNA na pozitivně nabité nylonové membráně a hybridizace za vhodných podmínek. Hybridizační signály jsou nejčastěji detekovány s využitím chemiluminiscence.
Mikročipy jsou komerčně připravované a poskytované různými firmami jako například Illumina, NimbleGen, Agilent a Affymetrix. DNA sondy, nebo prvky, které tvoří čip, jsou klíčovým faktorem při výběru nejvhodnější platformy k řešení dané otázky. Některé biočipy jako například od firmy Illumina jsou navrhované pro analýzu DNA po hydrogensiřičitanové modifikaci a některé se hodí pro analýzu po DNA restrikci.
IMUNOPRECIPITAČNÍ METODY
Imunoprecipitační metodiky umožňují izolovat metylovanou DNA, což usnadní následnou analýzu. Principem imunoprecipitačních metod je imobilizace denaturované DNA na DEAE (diethylaminoethyl) membráně a následná inkubace s monoklonální protilátkou.
Imunoprecipitace metylované DNA je běžně dosažena použitím jedné ze dvou metod: izolace DNA metylovaných fragmentů pomocí monoklonální protilátky specifické k 5-metylcytosinu (anti-5mC) nebo izolace DNA fragmentů obsahujících metylované CpG dinukleotidy za použití proteinu vázajícího metyl doménu (MBD protein – methyl binding domain protein) (10). Imunoprecipitovaná DNA může být následně analyzována různými výše zmíněnými metodami.
ZÁVĚR
Důležitost DNA metylace je jasná zejména u nádorových onemocnění. Důkazem je množství publikované literatury v posledních letech. V literárních pramenech je popsáno velké množství metod, které mohou být využívány ke sledování změn v metylaci DNA. Žádná z těchto metod ovšem nemůže být použita univerzálně, proto je důležitý správný výběr metody. Při volbě metody musíme dbát zejména na množství a kvalitu izolované DNA a možnosti a vybavení laboratoře. Mezi často analyzovanou DNA patří DNA izolovaná z parafínových bločků. Pro analýzu takovéto DNA se jeví nejvhodnější metodou MS-MLPA, musí být ovšem zváženy možnosti laboratoře, jelikož kromě běžně užívaného termocykléru je zapotřebí systém kapilární elektroforézy. V případě analýzy DNA izolované z krve je vhodné využít hydrogensiřičitanovou přeměnu následovanou MSP a případně zakončenou sekvenováním amplifikovaných produktů. Je důležité mít na paměti, že správný výběr metody zajišťuje reprodukovatelné výsledky.
Zkratky
- DEAE – diethylaminoethyl
- DNA – deoxyribonucleic acid
- DNMT – DNA metyltransferáza (methylatransferase)
- HPLC – high-performance liquid chromatography
- MBD – methyl binding domain
- miRNA – microRNA
- MS-HRM – methylation specific high-eesolution melting analysis
- MS-MLPA – methylation specific multiplex ligation-dependent probe amplification
- MSP – methylation specific PCR
- PCR – polymerase chain reaction
- RNA – ribonucleic acid
Práce byla podpořena výzkumným záměrem MZO 00179906.
ADRESA PRO KORESPONDENCI:
Mgr. Marcela Chmelařová
ÚKBD LF UK a FN
Sokolská 581, 500 05 Hradec Králové
e-mail: chmelarovamarcela@seznam.cz
Sources
1. Mareš J, Sedláček Z, Goetz P. Metylace DNA a nádorová onemocnění. Čas Lék čes 2000; 139(10): 291–294.
2. Esteller M. Epigenetics in Cancer. N Engl J Med 2008; 358(11): 1148–1159.
3. Bird A. DNA methylation patterns and epigenetic memory. Genes Dev 2002; 16(1): 6–21.
4. Herman JG, Graff JR, Myöhänen S, Nelkin BD, Baylin SB. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands. Proc Natl Acad Sci USA 1996; 93(18): 9821–9826.
5. Dejeux E, El Abdalaoui H, Gut IG, Tost J. Identification and quantification of differentially methylated loci by the pyrosequencing technology. Methods Mol Biol 2009; 507 : 89–205.
6. Wojdacz TK, Dobrovic A. Methylation-sensitive high resolution melting (MS-HRM): a new approach for sensitive and high--throughput assessment of methylation. Nucleic Acids Res 2007; 35(6): e41.
7. Waalwijk C, Flavell RA. MspI, an isoschizomer of hpaII which cleaves both unmethylated and methylated hpaII sites. Nucleic Acids Res 1978; 5(9): 3231–3236.
8. von Kanel T, Gerber D, Schaller A, Baumer A, Wey E, Jackson CB, Gisler FM, Heinimann K, Gallati S. Quantitative 1-step DNA methylation analysis with native genomic DNA as template. Clin Chem 2010; 56(7): 1098–1106.
9. Nygren AO, Ameziane N, Duarte HM, Vijzelaar RN, Waisfisz Q, Hess CJ, Schouten JP, Errami A. Methylation-specific MLPA (MS-MLPA): simultaneous detection of CpG methylation and copy number changes of up to 40 sequences. Nucleic Acids Res 2005; 33(14): e128.
10. Thu KL, Pikor LA, Kennett JY, Alvarez CE, Lam WL. Methylation analysis by DNA immunoprecipitation. J Cell Physiol 2010; 222(3): 522–531.
Labels
Addictology Allergology and clinical immunology Angiology Audiology Clinical biochemistry Dermatology & STDs Paediatric gastroenterology Paediatric surgery Paediatric cardiology Paediatric neurology Paediatric ENT Paediatric psychiatry Paediatric rheumatology Diabetology Pharmacy Vascular surgery Pain management Dental Hygienist
Article was published inJournal of Czech Physicians

-
All articles in this issue
- Reaktivní kyslíkové a dusíkové sloučeniny v klinické medicíně
- Současné možnosti léčení poranění pánevního kruhu
- Klinický význam nemoci spojené s imunoglobuliny IgG4
- Nejčastěji využívané metodiky k analýze DNA metylačních změn
- Hazard u dětí a dospívajících
- Naše zkušenosti s maravirocem u HIV pozitivních pacientů
- Antiepileptické léky a lékové interakce: klinická kazuistika
- Journal of Czech Physicians
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Současné možnosti léčení poranění pánevního kruhu
- Klinický význam nemoci spojené s imunoglobuliny IgG4
- Reaktivní kyslíkové a dusíkové sloučeniny v klinické medicíně
- Nejčastěji využívané metodiky k analýze DNA metylačních změn
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career