-
Články
- Vzdělávání
- Časopisy
Top články
Nové číslo
- Témata
- Kongresy
- Videa
- Podcasty
Nové podcasty
Reklama- Kariéra
Doporučené pozice
Reklama- Praxe
Fylogenetická analýza a genotypizace virů chřipky A/H3N2 izolovaných od pacientů hospitalizovaných s příznaky ILI (Influenza-Like Illnesses) v Nemocnici Na Bulovce v sezoně 2011/2012
Phylogenetic Analysis and Genotyping of A/H3N2 Influenza Viruses Isolated from Patients Hospitalised with Influenza-Like Illness Symptoms in the Na Bulovce Hospital in the Season 2011/2012
Influenza A virus is an important cause of acute respiratory infections (ARI). Clinical manifestations of ARI vary from mild or moderate to life-threatening conditions requiring intensive care. Given the segmented genome, a large natural reservoir of other influenza virus subtypes, and antibody selection pressure in the population, the virus is variable and genetically unstable. The phylogenetic analysis and genotyping of A/H3N2 influenza viruses isolated from patients hospitalised with influenza-like illness symptoms in the Na Bulovce Hospital in the season 2011/2012 support the assumption that the pathogenicity is a polygenic trait modifiable by the host health status and seems not to be unambiguously associated with any specific mutations.
Keywords:
influenza A virus, phylogenetic analysis – genotyping – hospitalization – clinical symptoms
Autoři: Nagy Alexander 1; Jiřincová Helena 1; Havlíčková Martina 1; Džupová Olga 2,4; Herrmannová Kristýna 3,4; Trojánek Milan 3,4; Marešová Vilma 3,4; Nováková Ludmila 1; Kynčl Jan 1
Působiště autorů: Státní zdravotní ústav, Centrum epidemiologie a mikrobiologie, Praha 1; 3. lékařská fakulta, Univerzita Karlova v Praze 2; 2. lékařská fakulta, Univerzita Karlova v Praze 3; Klinika infekčních nemocí, Nemocnice Na Bulovce, Praha 4
Vyšlo v časopise: Epidemiol. Mikrobiol. Imunol. 62, 2013, č. 1, s. 4-8
Souhrn
Virus chřipky typu A je důležitým vyvolavatelem akutních respiračních infekcí (ARI). Klinické projevy této infekce se pohybují od mírných či středně těžkých průběhů až po život ohrožující stavy vyžadující intenzivní péči. S ohledem na segmentovaný genom, velký přírodní rezervoár dalších subtypů i selekční tlak protilátek v populaci se jedná o virus proměnlivý a geneticky nestabilní. Fylogenetická analýza a genotypizace virů chřipky A/H3N2 izolovaných od pacientů hospitalizovaných s příznaky ILI v Nemocnici Na Bulovce v sezoně 2011/2012 podporuje názor, že patogenita je polygenní znak, který je modifikován aktuálním zdravotním stavem hostitele a pravděpodobně není jednoznačně svázaný s konkrétními mutacemi.
Klíčová slova:
virus chřipky typu A – fylogenetická analýza – genotypizace – hospitalizace – klinické příznakyÚVOD
Chřipkové viry typu A jsou důležitými vyvolavateli infekcí u lidí i zvířat, které svými klinickými projevy oscilují od lehkých forem až po závažné, život ohrožující stavy. Chřipkový virus je charakteristický segmentovaným genomem tvořeným 8 molekulami jednovláknové RNA. Segmentovaný charakter genomu v kombinaci s absencí editačních mechanismů při replikaci virové RNA vytváří základ pro ohromnou genetickou proměnlivost. Tato genetická variabilita spočívá buď v postupné kumulaci bodových mutací vedoucích ke změně antigenních vlastností, zvýšení patogenity či rezistence na antivirotika, nebo umožňuje hybridizaci odlišných virionů při koinfekci. Hybridizace virionů, tzv. genetický reassortment je podle současných znalostí základním mechanismem mezidruhového přenosu infekce a vzniku pandemických variant viru [3]. V současnosti v lidské populaci cirkulují tři základní subtypy viru chřipky A, a to H3N2, H1N1 a od roku 2009 tzv. pandemická H1N1. Chřipková sezona 2011/2012 byla v ČR stejně jako v jiných evropských státech charakterizována dominancí subtypu A/H3N2 s minoritní cirkulací typu B [1]. Subtyp A/H3N2 se v této sezoně prosadil jako epidemický kmen téměř po dvouleté odmlce, během které cirkulovala pandemická varianta A/H1N1. Cílem našeho krátkého sdělení je charakteristika genetické variability virů chřipky typu A/H3N2, které byly izolovány od pacientů přijatých k hospitalizaci v Nemocnici Na Bulovce v sezoně 2011/2012.
SOUBOR A METODIKA
Soubor vzorků pocházel od 19 pacientů pozitivních na virus chřipky A/H3N2. Všichni pacienti měli příznaky ILI a byli přijati k hospitalizaci do Nemocnice Na Bulovce v období únor až květen 2012. Při příjmu byl u pacientů odebrán stěr z nosohltanu a z obou nosních průduchů a použit pro molekulární detekci a izolaci viru chřipky A. Molekulární detekce a typizace byla provedena s využitím metody RT-qPCR (Center for Disease Control, Atlanta, USA) a izolace viru pak pomocí buněčného systému MDCK SIAT-1 [10]. Nakonec byly získané izoláty podrobeny celogenomovému sekvenování a následné fylogenetické analýze a genotypizaci. Pro celogenomové sekvenování byly použity vybrané oligonukleotidy z naší knihovny primerů pokrývající všechny segmenty viru chřipky A. Po editaci, sestavě a kvalitativní analýze sekvencí všech izolátů bylo provedeno jejich srovnání [6] a určení stupně sekvenční identity na nukleotidové i aminokyselinové úrovni [5]. Fylogenetická analýza pro každý segment byla provedena s využitím Neighbour-Joining nebo Maximum Likelihood metod [2]. Dendrogramy se graficky upravily s využitím TreeExplorer aplikace programu MEGA 4 [9]. Výsledky fylogenetické analýzy se shrnuly s využitím principů digitální genotypizace [8]. Všechny sekvence byly zadány do databáze GenBank.
VÝSLEDKY
Z 19 A/H3N2 pozitivních vzorků bylo úspěšně izolováno deset reprezentativních kmenů, na základě kterých se provedla fylogenetická analýza a genotypizace. Průměrný věk těchto deseti hodnocených pacientů byl 57 let, věkové rozmezí 28–95 let. Tři nejstarší pacientky měly základní interní chronické onemocnění (ischemická choroba srdeční, aterosklerotická demence). Nejčastěji pozorovanými symptomy byly kašel a horečka, klinický průběh byl mírný až středně závažný. Všichni pacienti byli léčeni symptomatickou terapií, virostatikum nebylo podáváno. U dvou pacientek byl průběh komplikován sekundární bakteriální infekcí dýchacích cest typu bronchitidy, které byly léčeny antibiotiky. Nebyl evidován ani jeden fatální průběh. Doba hospitalizace se pohybovala v rozmezí dva až pět dní (tab. 1). Porovnání sekvencí získaných kmenů na nukleotidové úrovni odhalilo překvapivě vysokou sekvenční variabilitu mezi určitými segmenty (tab. 2). Na aminokyselinové úrovni byla sekvenční variabilita výrazně nižší, s rozdíly hlavně u proteinu NS2, H3, PB2 a N2. Mapování molekulárních markerů majících vztah k patogenitě v genomu H3N2 izolátů indikovalo uniformní přítomnost celé škály patogenních mutací popsaných u jiných subtypů viru chřipky A a v jiných, převážně ptačích hostitelských systémech. Za pozornost stojí mutace PB2 E627K a dále mutace v proteinech NA a M1 indikujících rezistenci na antivirotika. Dalším zajímavým zjištěním je diferenciální exprese proteinu PB1-F2 mezi izoláty, a to úplné a zkrácené verze (34 aminokyselin) v poměru 4 : 6.
Tab. 1. Demografické a klinické ukazatele pacientů, u nichž byl izolován a geneticky analyzován virus chřipky A/H3N2 Table 1. Demographic and clinical characteristics of patients from whom A/H3N2 influenza virus was isolated and genetically analyzed 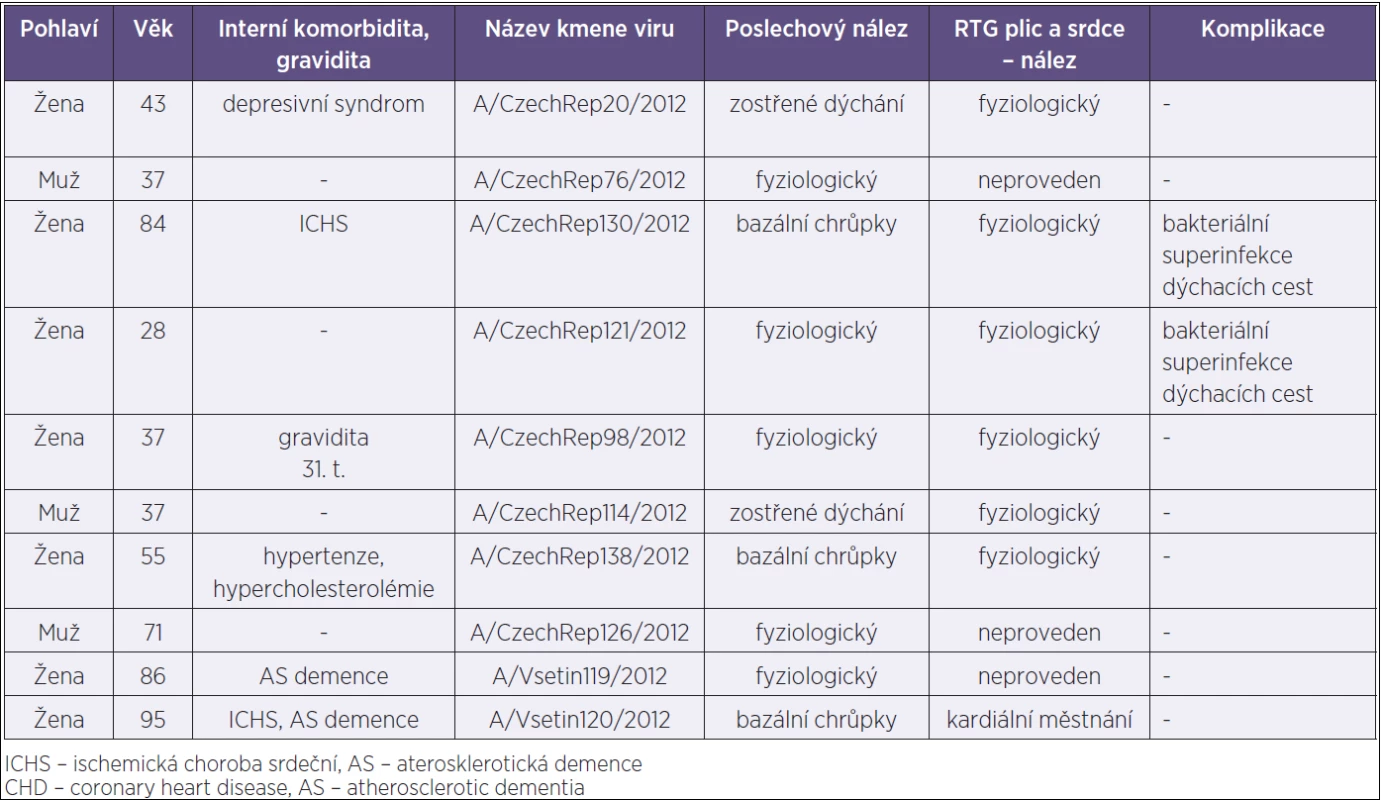
Tab. 2. Sekvenční variabilita analyzovaných kmenů chřipky A/H3N2 na nukleotidové a aminokyselinové úrovni v absolutních a procentuálních hodnotách Table 2. Sequence variability in A/H3N2 influenza strains at the nucleotide and amino acid level given in the absolute and percent values 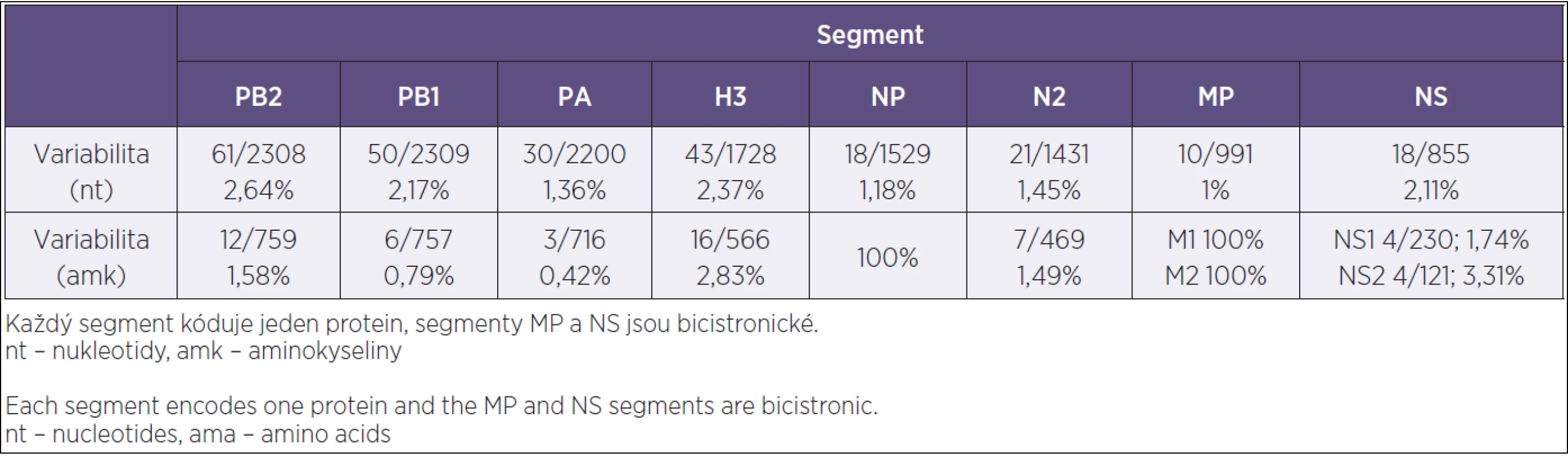
Nukleotidová variabilita se odrážela i v analýze fylogenetických vztahů, kde v závislosti na segmentu byla pozorována separace sekvencí na dvě až čtyři příbuzné, ale jasně rozlišitelné sublinie. Dendrogram zkonstruovaný na základě vybraných H3 sekvencí [4] odhalil, že všech deset izolátů patří do linie A/Victoria/208 (s referenčním kmenem A/Victoria/208/09), sublinií 3B, 3C a 6 a výrazně se odchylují od vakcinačního kmene A/Perth/16/09 (obr. 1). Porovnání sekvencí HA1 domén našich izolátů oproti A/Perth/16/09 odkrylo 11–15 aminokyselinových rozdílů, z nichž šest až osm bylo lokalizovaných ve známých antigenních oblastech molekuly H3. Shrnutí výsledků fylogenetických analýz všech genomických segmentů ve formě genotypizace indikovalo přítomnost pěti rozlišitelných genotypů, přičemž jeden vykazoval původ z intrasubtypového reassortmentu v poměru 7 : 1.
Obr. 1. Fylogenetická analýza molekuly hemaglutinínu H3 Referenční kmeny v jednotlivých liniích (Perth/16 a Victoria/208) a subliniích jsou zvýrazněný tlustě. Analyzované A/H3N2 izoláty z chřipkové sezony 2011/2012 jsou zvýrazněné podtržením. Fig. 1 Phylogenetic analysis of the H3 hemagglutinin molecule Reference strains in particular lines (Perth/16 and Victoria/208) and sublines are highlighted in bold. TheA/H3N2 isolates from the influenza season 2011/2012 are highlighted by underlining. 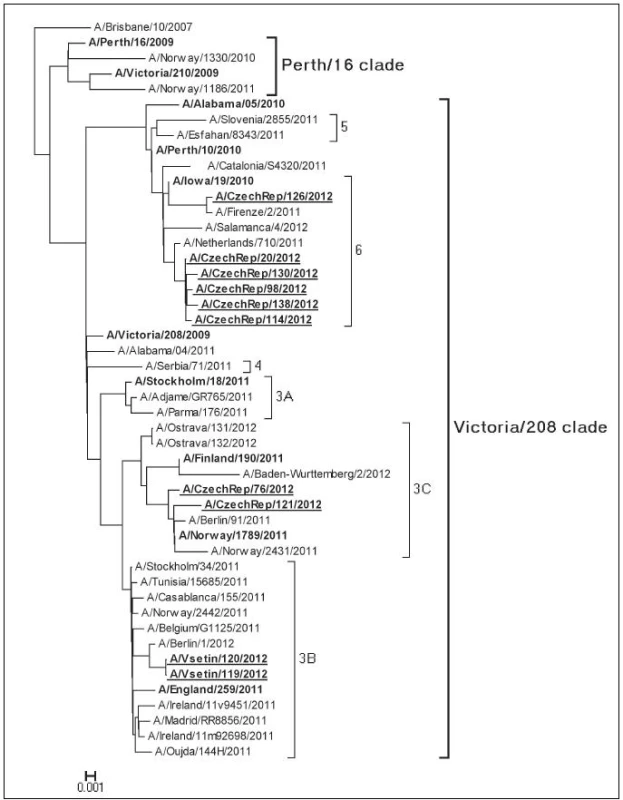
DISKUSE A ZÁVĚR
Ačkoliv prezentovaný soubor je v důsledku mírné epidemické sezony početně velmi limitovaný, předkládáme následující pracovní závěry:
- Analýza chřipkových kmenů ze sezony 2011/2012 ukázala, že byly již evolučně jasně odděleny od vakcinálního kmene A/Perth/16/2009.
- Relativně výrazná nukleotidová, ale nízká aminokyselinová variabilita indikuje převládající synonymní kodonovou variabilitu mezi analyzovanými kmeny. To ukazuje na kocirkulaci funkčně i antigenně relativně ekvivalentních segmentů, které se odlišují na genotypové úrovni.
- Fenotypově identické, ale genotypově rozdílné segmenty umožňují intenzivní intrasubtypový reassortment, pozorovaný i v našem souboru, jehož intenzita a význam v evoluci viru chřipky A se teprve objasňuje.
- Identifikace pěti rozdílných genotypů indikuje významnou genetickou heterogenitu viru chřipky A na relativně malém geografickém území s vysokou hustotou a intenzivním pohybem obyvatelstva (Praha).
- Dichotomie v expresi proteinu PB1-F2 je vzhledem k jeho polyfunkčnímu charakteru [7] nejasná.
- Přítomnost mnohých markerů patogenity popsaných v jiných systémech kontrastuje s benigním charakterem našich izolátů. Z toho plyne, že patogenita je pravděpodobně polygenní znak, který zůstává stále v mnoha směrech neobjasněn a přímo souvisí s aktuálním zdravotním stavem hostitele.
- Mutace, které jsou popisovány u některých těžkých průběhů chřipky, jsou nacházeny i u průběhů klinicky mírných a jejich interpretace je proto nejednoznačná. V současné době nelze jednoznačně doložit, že by některé mutace – s výjimkou těch, které nesou rezistenci na inhibitory neuraminidázy – byly jasně a pravidelně spjaty s určitou klinickou formou infekce.
Tato práce vznikla za podpory grantu IGA MZ ČR 12493-3: Virologická a genetická charakteristika viru chřipky A ve vztahu ke klinické závažnosti infekce.
Do redakce došlo dne 8. 1. 2013.
Adresa pro korespondenci:
RNDr. Alexander Nagy
Národní referenční laboratoř pro chřipku
Státní zdravotní ústav
Šrobárova 48
100 42 Praha
e-mail: alexandernagy17@hotmail.com
Zdroje
1. European Centre for Disease Prevention and Control Weekly Influenza Surveillance Reports. Dostupné na www:
<http://www.ecdc.europa.eu/en/healthtopics/seasonal_influenza/epidemiological_data/Pages/Weekly_Influenza_Surveillance_Overview.aspx>.
2. Felsenstein, J. PHYLIP (Phylogeny Inference Package) Version 3.6. Distributed by the Author. Department of Genome Sciences, University of Washington, Seattle, 2004. Dostupné na www: <http://evolution.genetics.washington.edu/phylip.html>.
3. Garten, R. J. et al. Antigenic and genetic characteristics of swine-origin 2009 A(H1N1) influenza viruses circulating in humans. Science, 2009, 325, pp. 197–201.
4. GISAID (Global Initiative of Sharing Avian Influenza Data) 2011. Dostupné na www: <http://platform.gisaid.org/dante-cms/struktur.jdante?aid=1131>.
5. Hall, T. A. BioEdit: a user-friendly biological sequence alignmentu editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 1999, 41, pp. 95–98. Dostupné na www: <http://www.mbio.ncsu.edu/RNaseP/info/programs/BIOEDIT/bioedit.html>.
a7. Krumbholz, A., Philipps, A., Oehring, A., Schwarzer, K., Eitner, A., Wutzler, P., Zell, R. Current knowledge on PB1-F2 of influenza A viruses. Med. Microbiol. Immunol., 2011, 200, pp. 69–75.
8. Nagy, A. Černíková, L., Křivda, V., Horníčková, J. Digital genotyping of avian influenza viruses of H7 subtype detected in central Europe in 2007–2011. Virus Res., 2012, 165, pp. 126–133.
9. Tamura, K., Dudley, J., Nei, M., Kumar, S. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0. Mol. Biol. Evol., 2007, 24, pp. 1596–1599.
10. World Health Organization Manual for the Laboratory diagnosis and virological surveillance of influenza [on line]. 2011 [cit.2012-12-18]. Dostupné na www:
ISBN: 9789241548090. Štítky
Hygiena a epidemiologie Infekční lékařství Mikrobiologie
Článek ÚvodníkČlánek RNDr. Marie Vacková, CSc.
Článek vyšel v časopiseEpidemiologie, mikrobiologie, imunologie
Nejčtenější tento týden
2013 Číslo 1- Stillova choroba: vzácné a závažné systémové onemocnění
- Perorální antivirotika jako vysoce efektivní nástroj prevence hospitalizací kvůli COVID-19 − otázky a odpovědi pro praxi
- Jak souvisí postcovidový syndrom s poškozením mozku?
-
Všechny články tohoto čísla
- Odlišnosti výskytu virových hepatitid A a E v České republice
- Bakteriální infekce jako příčina neplodnosti u lidí
- Úvodník
- Antimalarická profylaxe při cestách do zahraničí
- Fylogenetická analýza a genotypizace virů chřipky A/H3N2 izolovaných od pacientů hospitalizovaných s příznaky ILI (Influenza-Like Illnesses) v Nemocnici Na Bulovce v sezoně 2011/2012
- RNDr. Marie Vacková, CSc.
- Incidence onemocnění klíšťovou encefalitidou v České republice v letech 2001–2011 v jednotlivých krajích a obcích s rozšířenou působností
- Epidemiologie, mikrobiologie, imunologie
- Archiv čísel
- Aktuální číslo
- Informace o časopisu
Nejčtenější v tomto čísle- Bakteriální infekce jako příčina neplodnosti u lidí
- Antimalarická profylaxe při cestách do zahraničí
- Odlišnosti výskytu virových hepatitid A a E v České republice
- Incidence onemocnění klíšťovou encefalitidou v České republice v letech 2001–2011 v jednotlivých krajích a obcích s rozšířenou působností
Kurzy
Zvyšte si kvalifikaci online z pohodlí domova
Autoři: prof. MUDr. Vladimír Palička, CSc., Dr.h.c., doc. MUDr. Václav Vyskočil, Ph.D., MUDr. Petr Kasalický, CSc., MUDr. Jan Rosa, Ing. Pavel Havlík, Ing. Jan Adam, Hana Hejnová, DiS., Jana Křenková
Autoři: MUDr. Irena Krčmová, CSc.
Autoři: MDDr. Eleonóra Ivančová, PhD., MHA
Autoři: prof. MUDr. Eva Kubala Havrdová, DrSc.
Všechny kurzyPřihlášení#ADS_BOTTOM_SCRIPTS#Zapomenuté hesloZadejte e-mailovou adresu, se kterou jste vytvářel(a) účet, budou Vám na ni zaslány informace k nastavení nového hesla.
- Vzdělávání



