-
Medical journals
- Career
Možnosti implementace sekvenace celého genomu (WGS) Neisseria meningitidis do molekulární surveillance invazivního meningokokového onemocnění v České republice
Authors: Z. Vacková; P. Křížová; Z. Jandová; M. Musílek; J. Kozáková
Authors‘ workplace: Centrum epidemiologie a mikrobiologie, Státní zdravotní ústav, Praha
Published in: Epidemiol. Mikrobiol. Imunol. 66, 2017, č. 1, s. 50-52
Category: Short Communication
Overview
Zavedení molekulární surveillance u invazivního meningokokového onemocnění je celosvětově doporučeno. Nově se do popředí dostává metoda sekvenace celého genomu (Whole Genome Sequencing, WGS), která poskytuje možnosti další detailní charakterizace Neisseria meningitidis a umožňuje efektivní spojení všech „klasických“ sekvenačních metodik v tuto jednu metodu. Pro studium novou WGS metodou bylo vybráno šest izolátů N. meningitidis z let 2013 a 2015, které již byly charakterizovány klasickým Sangerovým přístupem amplikonové sekvenace. Analýza WGS dat potvrdila správnost této metody při určování epidemiologických markerů. Cílem sdělení je upozornit na možnost implementace WGS do molekulární surveillance invazivního meningokokového onemocnění v České republice. Národní referenční laboratoř pro meningokokové nákazy (NRL/MEN) bude nadále využívat WGS k molekulární charakterizaci vybraných izolátů N. meningitidis a ke zpřesnění molekulární surveillance invazivního meningokokového onemocnění v České republice.
KLÍČOVÁ SLOVA:
sekvenace celého genomu – WGS – Neisseria meningitidis – molekulární surveillanceÚVOD
Invazivní meningokokové onemocnění je stále jedním z nejzávažnějších infekčních onemocnění, vzhledem k přetrvávající vysoké smrtnosti i vysokému procentu celoživotních závažných následků u přeživších pacientů. Nejvyšší nemocnost a smrtnost tohoto onemocnění je u nejmenších dětí pod jeden rok věku a u adolescentů. Přesná epidemiologická data, včetně dat molekulární charakterizace izolátů Neisseria meningitidis, jsou nezbytná pro zavedení účinných preventivních opatření včetně vakcinačních doporučení. Zavedení molekulární surveillance u invazivního meningokokového onemocnění je celosvětově doporučeno včetně doporučení Evropským centrem pro kontrolu nemocí (ECDC), které doporučuje implementaci WGS do surveillance invazivního meningokokového onemocnění na evropské úrovni [1].
Po identifikaci a určení séroskupin izolátů N. meningitidis je v Národní referenční laboratoři pro meningokokové nákazy (NRL/MEN) k charakterizaci běžně používána metoda multilokusové sekvenace (Multilocus Sequence Typing, MLST) se Sangerovým přístupem.
MLST vykazuje pro izoláty N. meningitidis vysokou diskriminační schopnost [2], když pomocí ní byly v řadě zemí včetně České republiky zjištěny klonální komplexy, které působí častěji invazivní meningokokové onemocnění než nosičství, tzv. hypervirulentní klonální komplexy [3].
Další používanou molekulární metodou v NRL/MEN je sekvenační identifikace genů antigenů čtyřsložkové vakcíny proti meningokoku B (4CMenB), která umožňuje predikci pokrytí českých izolátů N. meningitis touto vakcínou [10].
Nově se do popředí dostává metoda sekvenace celého genomu (Whole Genome Sequencing, WGS), která poskytuje možnost další detailní charakterizace N. meningitidis. WGS umožňuje efektivní spojení všech „klasických“ sekvenačních metodik v tuto jednu metodu a získání celkových genetických charakteristik izolátů. Metoda WGS může být použita jak v oblasti základního výzkumu, například při studiu faktorů virulence N. meningitidis, tak v oblasti aplikovaného výzkumu, například při studiu epidemiologických markerů N. meningitidis. Tento aplikovaný výzkum může perspektivně vyústit do rutinního použití WGS v molekulární surveillance invazivního meningokokového onemocnění [4]. Také byla vyvinuta metoda whole-genome MLST (wgMLST), která poskytuje efektivní přístup k interpretaci WGS dat [5]. Analýza WGS dat s použitím platformy Bacterial Isolate Genome Sequence Database (BIGSdb) [6, 7] se ukázala velmi užitečnou při analýze ohniska invazivního meningokokového onemocnění [8]. Ukládání a analýza WGS dat N. meningitidis v databázi PubMLST, která uchovává MLST údaje získané v celosvětovém rámci a další sekvenční data za více než 15 let, se osvědčilo jako efektivní při využití těchto dat pro detailní molekulární typizaci [9].
Využití molekulárních metod v surveillance invazivního meningokokového onemocnění má v České republice již dlouhou tradici a molekulární charakterizace N. meningitidis je nezbytným předpokladem pro aktualizaci vakcinačních doporučení [10]. Metoda WGS, která umožní detailnější charakterizaci tohoto agens, byla nově aplikována v rámci řešení výzkumného projektu. Cílem tohoto krátkého sdělení je upozornit na možnost implementace WGS do molekulární surveillance invazivního meningokokového onemocnění v České republice. Předkládáme první výsledky WGS provedené v NRL/MEN u vybraných izolátů N. meningitidis.
Pro studium novou WGS metodou bylo vybráno šest izolátů N. meningitidis z let 2013 a 2015, které již byly charakterizovány klasickou Sangerovou metodou amplikonové sekvenace: čtyři izoláty od pacientů s invazivním meningokokovým onemocněním (dva izoláty séroskupiny C, po jednom séroskupiny B a Y) a dva izoláty od zdravých osob, které byly v kontaktu s invazivním meningokokovým onemocněním (po jednom izolátu séroskupiny B a Y).
Kmeny N. meningitidis vybrané pro testování WGS byly kultivovány na čokoládovém agaru Mueller-Hinton přes noc v 37 oC ve zvýšené tenzi CO2. Následovala manuální izolace DNA pomocí QIAmp DNA Mini Kitu (QIAGEN) s použitím enzymu RNasy A (DYNEX). Kontrola koncentrace vzorků byla prováděna měřením absorbance na přístroji Nanodrop 1000-ND (THERMO) a fluorescence na přístroji INFINITE 200 PRO (TECAN). K ověření kvality degradace DNA byla použita agarosová gelová elektroforéza. Před provedením WGS vzorků v IAB (Institute of Applied Biotechnologies, Olomouc, ČR) probíhalo nejprve hodnocení jejich kvality. Koncentrace DNA byla testována na přístroji QUBIT 2.0 (THERMO) a degradace pomocí agarosové gelové elektroforézy. Příprava knihovny vzorků probíhala podle standardního protokolu enzymatickou metodou (NEXTERA XT DNA kit, Illumina). Kvalita knihovny byla detailně testována přístroji QUBIT 2.0, Bioanalyzer Agilent 2100 (High Senzitivity DNA Assay) a metodou qPCR (KAPA kit). V případě potřeby byla použita purifikace fragmentů DNA pomocí magnetických kuliček (Agencourt AMPure beads) podle purifikačního protokolu Illuminy. WGS sekvenace probíhala na přístroji MiSeq (Illumina) za použití odpovídající sekvenační chemie (MiSeq V2 Reagent kit, 2x250). Přenos dat a primární hodnocení kvality sekvenačních běhů probíhalo přes BaseSpace (Illumina). Sekundární analýzy WGS dat byly provedeny v NRL/MEN pomocí softwarů CGE (Center for Genomic Epidemiology, http://www.genomicepidemiology.org/).
WGS data byla analyzována z pohledu epidemiologického, kdy byly hodnoceny alely lokusů abcZ, adk, aroE, fumC, gdh, pdhC, pgm, jež jsou zařazeny v základním MLST schématu Neisseria meningitidis a na jejichž podkladě se rozlišují sekvenční typy (ST), které jsou zařazovány do klonálních komplexů (cc). Analýza WGS dat potvrdila stejné epidemiologické markery N. meningitidis zjištěné metodou MLST: u dvou izolátů séroskupiny B byl zjištěn ST-10126, náležící do cc35, u dvou izolátů séroskupiny C byl zjištěn ST-3346, náležící do hypervirulentního cc41/44, u dvou izolátů séroskupiny Y byl zjištěn ST-23, náležící do hypervirulentního cc23 – tabulka 1. Byla tedy potvrzena úplná shoda ve zjištění sekvenčních typů a klonálních komplexů N. meningitidis klasickou sekvenací MLST a novou metodou WGS.
Table 1. Molekulární charakterizace zjištěná celogenomovou sekvenací u vybraných izolátů Neisseria meningitidis Česká republika Table 1. Molecular characterization of Neisseria meningitidis selected isolates using the whole genome sequencing in the Czech Republic 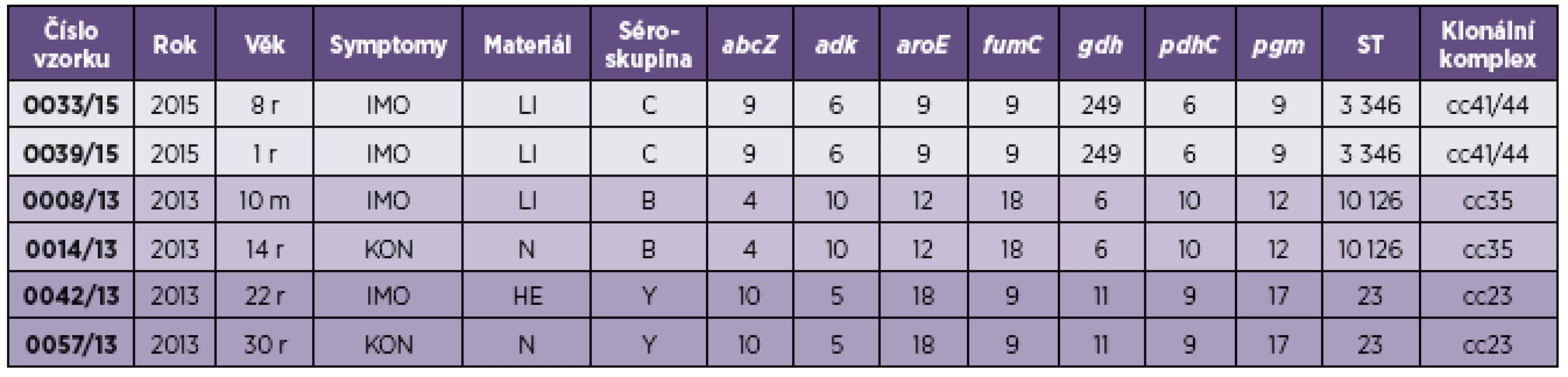
IMO = invazivní meningokokové onemocnění, KON = kontakt invazivního meningokokového onemocnění, LI = likvor, HE = hemokultura, N = výtěr nasopharyngu, ST = sekvenční typ, abcZ, adk, aroE, fumC, gdh, pdhC, pgm = geny sedmi lokusů zjišťované v multilokusové sekvenační analýze (MLST). IMO = invasive meningococcal disease, KON = contact with invasive meningococcal disease, LI = liquor, HE = blood culture, N = nasopharynx swab, ST = sequence type, abcZ, adk, aroE, fumC, gdh, pdhC, pgm = genes of seven loci analyzed in the multilocus sequencing analysis (MLST). Pro studium metodou WGS byly vybrány izoláty N. meningitidis, se stejnými epidemiologickými markery, ale z různých klinických symptomů, tj. od pacienta s invazivním meningokokovým onemocněním a od asymptomatické osoby, která byla v kontaktu s invazivním meningokokovým onemocněním, s cílem nejen potvrdit identifikaci molekulárních epidemiologických markerů novou WGS metodou, ale i testovat možnost studia faktorů virulence N. meningitidis.
Údaje získané metodou WGS umožnily u izolátů N. meningitidis identifikovat více než 422 genů kódujících popsané známé či hypotetické proteiny. Studie těchto genů a jejich významu bude dále probíhat.
Závěrem lze konstatovat, že metoda WGS N. meningitidis byla úspěšně poprvé provedena v České republice v NRL/MEN u šesti vybraných izolátů. Analýza WGS dat potvrdila správnost této metody při určování epidemiologických markerů. NRL/MEN bude nadále využívat WGS k molekulární charakterizaci vybraných izolátů N. meningitidis, ke zpřesnění molekulární surveillance invazivního meningokokového onemocnění v České republice. WGS data budou poskytnuta do mezinárodních databází.
Poděkování
Podpořeno z programového projektu Ministerstva zdravotnictví ČR s reg. č. 15-34887A. Veškerá práva podle předpisů na ochranu duševního vlastnictví jsou vyhrazena.
Díky Joël Mossongovi (Laboratoire National de Santé, Surveillance and epidemiology of infectious diseases, Luxembourg) za konzultace při sekundární analýze WGS dat.
Do redakce došlo dne 28. 7. 2016.
Adresa pro korespondenci:
Mgr. Zuzana Vacková
SZÚ Praha
Šrobárova 48
100 42 Praha 10
e-mail: zuzana.vackova@szu.cz
Sources
1. European Centre for Disease Prevention and Control. Expert opinion on whole genome sequencing for public health surveillance. Stockholm: ECDC; 2016; s. 1–14. ISBN 978-92-9193-888-9.
2. Maiden MC, Bygraves JA, Feil E, et al. Multilocus sequence typing: a portable approach to the identification of clones within populations of pathogenic microorganisms. Proc Natl Acad Sci USA, 1998;95 : 3140–3145.
3. Jandová Z, Musílek M, Vacková Z, et al. Klonální analýza populace meningokoky z invazivního onemocnění a od zdravých nosičů izolovaných v České republice v období 1971–2014 (květen). Epidemiol Mikrobiol Imunol, 2015;64(3):147–152.
4. Hill DM, Lucidarme J, Gray SJ, et al. Genomic epidemiology of age-associated meningococcal lineages in national surveillance: an observational cohort study. Lancet Infect Dis, 2015;15(12):1420–1428. doi: 10.1016/S1473-3099(15)00267-4. Epub 2015 Oct 27.
5. Jolley KA, Maiden MCJ. Using MLST to study bacterial variation: prospects in the genomic era. Future Microbiol, 2014;9(5):623–630.
6. Jolley KA, Maiden MCJ. BIGSdb: Scalable analysis of bacterial genome variation at the population level. BMC Bioinformatics, 2010;11 : 595.
7. Jolley KA, Maiden MCJ. Automated extraction of typing information for bacterial pathogens from whole genome sequence data: Neisseria meningitidis as an exemplar. Euro Surveill, 2013;18(4):20379.
8. Jolley KA, Hill DM, Bratcher HB, et al. Resolution of a meningococcal disease outbreak from whole-genome sequence data with rapid Web-based analysis methods. J Clin Microbiol, 2012;50 : 3046–3053.
9. Maiden MC, Jansen van Rensburg MJ, Bray JE, et al. MLST revisited: the gene-by-gene approach to bacterial genomics. Nat Rev Microbiol, 2013;11(10):728–736.
10. Křížová P, Musílek M, Vacková Z, et al. Sekvenční analýza antigenů zařazených v čtyřkomponentní vakcíně proti meningokoku B v Českých izolátech Neisseria meningitidis v období 2007–20013. Epidemiol Mikrobiol Imunol, 2014;63(1):61–68.
Labels
Hygiene and epidemiology Medical virology Clinical microbiology
Article was published inEpidemiology, Microbiology, Immunology

2017 Issue 1-
All articles in this issue
- Prevalence protilátek proti viru hepatitidy E v České republice – sérologický přehled
- Infekční choroby a laboratorní diagnostika v podmínkách Židovského ghetta Terezín
- Nespecifická imunoterapie inhibuje angiogenezi – výsledky monitorování sérových hladin vaskulárního endotelového růstového faktoru a matrixmetaloproteinázy 8 v průběhu adjuvantní léčby vysokodávkovaným interferonem u pacientů s maligním melanomem
- Management akutní anafylaxe v klinické praxi v kontextu doporučených postupů
- Alimentárny botulizmus – staronová výzva pre verejné zdravotníctvo
- Možnosti implementace sekvenace celého genomu (WGS) Neisseria meningitidis do molekulární surveillance invazivního meningokokového onemocnění v České republice
- Trend a výzva v prenose syfilisu z matky na dieťa
- Epidemiology, Microbiology, Immunology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Management akutní anafylaxe v klinické praxi v kontextu doporučených postupů
- Prevalence protilátek proti viru hepatitidy E v České republice – sérologický přehled
- Alimentárny botulizmus – staronová výzva pre verejné zdravotníctvo
- Infekční choroby a laboratorní diagnostika v podmínkách Židovského ghetta Terezín
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

