-
Medical journals
- Career
Mobilní genetické elementy v epidemiologii rezistence bakterií k antibiotikům
: J. Hrabák; A. Zemanová; E. Chudáčková
: Ústav mikrobiologie, LF UK a FN v Plzni
: Epidemiol. Mikrobiol. Imunol. 59, 2010, č. 2, s. 55-66
Mobilní elementy a mobilizace genů rezistence jsou nedílnou součástí poznatků pro pochopení mechanismů epidemiologie antibiotické rezistence. Tento souhrnný článek popisuje recentní znalosti o inserčních sekvencích, transpozonech, integronech a plazmidech ve vztahu k přenosu genů zodpovědných za rezistenci bakterií k antibiotikům.
Klíčová slova:
antibiotická rezistence – inerční sekvence – transpozon – integron – plazmid.1. Úvod
Mikrobi obývají naší planetu přibližně 3,5 miliardy let. Ve srovnání s dobou výskytu člověka je to čas takřka nesouměřitelný. Během té doby byli mikrobi nuceni vzájemně soutěžit o životní prostředí. Proto se někteří z nich naučili produkovat toxické látky, jiní se účinkům těchto látek naučili naopak bránit. Produkci antibiotik, látek toxických pro jiné druhy mikrobů, objevili lidé až v minulém století. I když se za první použitelné antibiotikum považuje penicilin, již v roce 1899 popsali čeští bakteriologové Honl a Bukovský objev antibiotických účinků pyocyaninu, produktu bakterie Pseudomonas aeruginosa [23]. Velmi rychle se však začaly objevovat kmeny bakterií k antibiotikům rezistentní a dnes se může zdát, že stojíme na začátku konce antibiotické éry. Vyřazením, případně významnou redukcí účinnosti antibiotik by byly limitovány mnohé lékařské obory, jejichž rozvoj si nelze bez efektivní antibiotické terapie vůbec představit.
Rezistence bakterií k antibiotikům vzniká obecně dvěma způsoby [2, 4]:
-
Mutacemi
- a. genů jejichž produkty jsou cílovými místy antibiotik,
- b. genů kódujících enzymy (příp. obecně proteiny) inaktivující účinek antibiotika, jenž způsobí změnu jejich substrátové specificity,
- c. promotorů způsobujících hyperprodukci inaktivujícího enzymu, efluxních pump, resp. mutacemi dalších strukturálních složek buněčné stěny (např. inaktivace porinů snižujících permeabilitu vnější buněčné stěny).
- Akvizicí nových genů. Zde dochází k expanzi genetické
informace bakterie.
Geny rezistence mohou být mobilizovány několika různými mechanismy – přenosem na plazmidech, konjugativních transpozonech, případně mohou být přeneseny bakteriofágy – transdukcí. Zároveň je důležitá jejich mobilizace uvnitř bakteriální buňky tak, aby k intercelulárnímu přenosu mohlo vůbec dojít.
Následující text je věnován mechanismům mobilizace a horizontálního přenosu genetické informace v bakteriálních populacích v souvislosti s geny zodpovědnými za rezistenci k antibiotikům.
U těchto typů rezistence se jedná pouze o změny ve stávajícím genomu a lze ji označit jako rezistenci adaptivní.
2. Inserční sekvence
Inserční sekvence (IS) jsou krátké úseky DNA (< 2,5 kb), schopné nezávislé transpozice v mikrobiálním genomu. Jejich funkce je obecně omezena pouze na zajištění jejich mobility [32]. Mimo bakterií se vyskytují také u eukaryot. Díky svým vlastnostem způsobují inserční mutace, rekombinace, případně mobilizují geny virulence nebo rezistence.
1. Obvyklá struktura inserční sekvence. IRL – invertovaná repetice levá, IRR – invertovaná repetice pravá, Pt – promotor genu transposázy, I – doména rozpoznávaná transposázou jako štěpné místo, II – vazebná doména transposázy, XYZ – generované repetice. Volně podle [32]. <b>Fig. 1. </b>General structure of the insertion sequence. IRL – inverted repeat left, IRR – inverted repeat right, Pt – transposase gene promoter, I – transposase-recognized insertion domain, II – transposase-binding domain, XYZ – generated repeats. Based on [32]. ![Obvyklá struktura inserční sekvence. IRL – invertovaná
repetice levá, IRR – invertovaná repetice pravá, Pt – promotor
genu transposázy, I – doména rozpoznávaná transposázou
jako štěpné místo, II – vazebná doména transposázy, XYZ
– generované repetice. Volně podle [32].
<b>Fig. 1. </b>General structure of the insertion sequence. IRL –
inverted repeat left, IRR – inverted repeat right, Pt – transposase
gene promoter, I – transposase-recognized insertion
domain, II – transposase-binding domain, XYZ – generated
repeats. Based on [32].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/8a744673dd40f8a0808d7d7cb8831313.jpeg)
Běžná struktura IS je znázorněna na obrázku 1. Základní složkou je gen transposázy, který je ohraničen krátkými úseky označovanými jako invertované repetice levá a pravá (IRL a IRR – Inverted Repeat Left, Inverted Repeat Right). Jedná se o místa rozpoznávaná transpsoázou, která se skládají ze dvou podjednotek (domén). Doména I je úsekem, který obsahuje krátký element (2-3 bp), rozpoznávaný transpsoázou jako štěpné místo. Doména II je vazebným místem transposázy. Nutno podotknout, že IRR a IRL nejsou obecně inverzní komplementární sekvence. IRL rovněž obsahuje promotor genu transposázy. Poslední součástí nalézanou u většiny IS jsou generované repetice, v anglické literatuře běžně označované jako DRs (directly repeated sequences). Tyto sekvence, dlouhé 2-14 bp, jsou generovány při inserci inserční sekvence do cílové molekuly DNA [32].
K transpozici inserční sekvence dochází přímou vazbou transposázy na invertované repetice s následným rozpojením vazby a přenesením na cílové místové recipientní molekuly DNA. V případě jednoduché inserce je přenesena IS kompletním vystřižením. Volné konce na donorové molekule DNA jsou následně spojeny. Ve druhém případě dojde po následné replikaci ke ko-integraci – spojení donorové a recipientní molekuly DNA (viz obr. 2) [32].
2. Mechanismy přenosu inserční sekvence mezi jednotlivými molekulami DNA. DRs – generované repetice. Volně podle [32] <b>Fig. 2. </b>Mechanisms of insertion sequence transfer between DNA molecules. DRs – generated repeats. Based on [32] ![Mechanismy přenosu inserční sekvence mezi jednotlivými molekulami DNA. DRs – generované repetice. Volně podle [32]
<b>Fig. 2. </b>Mechanisms of insertion sequence transfer between DNA molecules. DRs – generated repeats. Based on [32]](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/50d200b133837416b7750d968d106c97.jpeg)
Některé inserční sekvence jsou schopny vyvolat na své mateřské molekule DNA stav „imunity“, kdy tatáž inserční sekvence nemůže být na tuto molekulu opětovně vložena [32].
Specificita rozpoznávání cílového místa na molekule DNA, kam má být IS vložena, se různí. Některé IS, respektive jejich transposázy, se vyznačují nízkou specificitou (v řádu několika nukleotidů), jiné vyžadují přesné, relativně dlouhé sekvence [32].
Inserční sekvence jsou klasifikovány podle své struktury – složení transposázy, invertovaných a generovaných repetic, atp. Do současnosti bylo u prokaryot popsáno celkem 25 různých rodin, které čítají několik set různých IS. Databázi IS lze najít na internetové stránce:
2.1. Inserční sekvence rodiny IS91
Inserční sekvence této rodiny se vyznačují jiným mechanismem transpozice než je běžné u ostatních IS [32, 68]. Tento mechanismus je nazýván RC transpozice (Rolling-Circle transposition) (obr. 3). V epidemiologii antibiotické rezistence se jedná o významné mobilní elementy, které nedisponují klasickými invertovanými repeticemi. Sekvence, jež se nachází v blízkosti 3’ konce genu transposázy je označována jako oriIS. Je rozpoznávána transposázou a místně specificky napojena na recipientní molekulu DNA (obvykle rozpoznává sekvenci 5’-GTTC-3’). Následně dochází k replikaci, která je ukončena v místě terIS, nacházejícím se v blízkosti 5’ konce genu transposázy (obr. 3A). Pokud však není místo terIS rozpoznáno, replikace pokračuje (obr. 3B). Na recipientní molekulu je poté přenesen delší úsek DNA, který může obsahovat další geny, např. geny rezistence k antibiotikům. Ke stejné situaci může dojít při chybném rozpoznání sekvence oriIS [68].
3. Mechanismus RC transpozice. A – při replikaci je rozpoznáno místo terIS a dochází k přenosu pouze samotné inserční sekvence. B – terIS není rozpoznáno a replikace pokračuje. Následně dochází k přenosu delšího úseku DNA než je samotná IS. <b>Fig. 3.</b> RC transposition mechanism. A – the terIS site is recognized during replication and only the insertion sequence is transposed. B – the terIS site is not recognized and replication is going on. Subsequently a longer DNA part than the single IS is transposed. 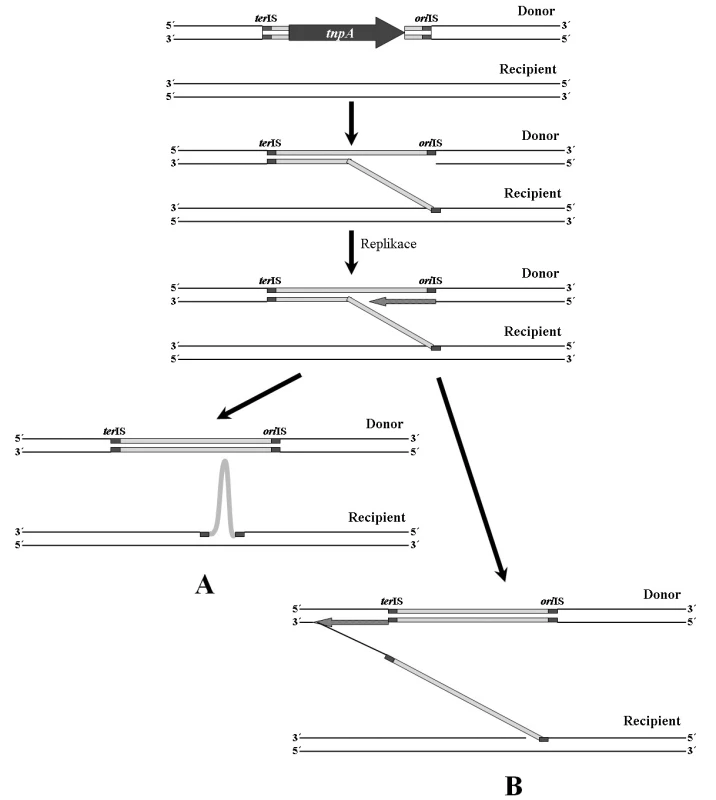
Při mobilizaci genů rezistence se významně uplatňují inserční sekvence ISCR (CRs – Common Regions), které byly dlouho známy pod označením orf513 (ISCR1). Tyto IS rovněž náleží do rodiny IS91 [32, 68].
2.2. Inserční sekvence a mobilizace genů
Pokud se na molekule DNA obsahující IS v její blízkosti vyskytují sekvence podobné invertovaným repeticím (IR-like), mohou být tyto „nesprávně“ rozpoznány a místo přenosu pouze inserční sekvence, dojde k transportu celého elementu (viz obr. 4) [30, 49, 50]. Stejně tak, pokud je určitý úsek ohraničen dvěma shodnými IS, může dojít k přenosu celého tohoto úseku. Taková struktura je nazývána kompozitní transpozon (viz dále) [17, 65].
4. Transpozice inserční sekvence nesprávným rozpoznáním IRR podobné sekvence. IRR-like – sekvence podobná pravé invertované repetici, IRL – invertovaná repetice levá, IRR – invertovaná repetice pravá. A – přenos pouze samotné inserční sekvence, B – transposázou chybně rozpoznaná IRR-like místo IRR, vedoucí k přenosu celého segmentu. <b>Fig. 4.</b> Insertion sequence transposition due to misrecognition of an IRR-like sequence. IRR-like sequence – right inverted repeat-like, IRL – left inverted repeat, IRR – right inverted repeat. A – transposition of the insertion sequence alone, B – an IRR-like sequence misrecognized by transposase as the IRR sequence and subsequent transposition of the whole segment. 
Mezi nejčastější inserční sekvence spjaté s mobilizací genů rezistence patří ISEcp1 (náležející do rodiny IS1380) [30, 49, 50] a ISCR [40, 53, 68]. ISEcp1 je zodpovědná za mobilizaci některých genů β-laktamáz, především skupiny CTX-M [30, 49, 50], ale byly nalezeny také v souvislosti s geny qnr, způsobujícími rezistenci k fluorochinolonům [39, 68]. Například v případě ESBLA podskupiny CTX-M-1 lze zjištěním vzdálenosti ISEcp1 od genu blaCTX-M mapovat geografickou příbuznost jednotlivých izolátů.
Elementy ISCR mobilizují celou řadu genů rezistence. Jako příklad lze uvést β-laktamázy DHA-1, CMY, OXA-1, OXA-4, atd., dále pak například geny dfr zodpovědné za rezistenci k trimetoprimu, fluorochinolové rezistence qnr atp. [39, 53, 68, 70, 71]. Tyto inserční sekvence se obvykle nachází v 3’ CS regionu komplexních sul1 integronů. Jejich význam je popsán v souhrnném článku [68]. Spolu s ISCR byly nalezeny v jejich blízkosti další inserční sekvence, např. IS26 a IS6100 [70, 71], jejichž význam však není dosud plně objasněn [68].
2.3. Inserční sekvence jako promotor genů rezistence
Významnou úlohou inserčních sekvencí v epidemiologii antibiotické rezistence je zvýšení exprese genů rezistence. To je způsobeno tím, že některé části IS, především invertované repetice, tvoří silné promotory. Lokalizace takového promotoru v oblasti IRR vede ke zvýšené expresi genů nacházejících se za IS [50, 51].
Typickým příkladem může být opět ISEcp1 u enterobakterií a ISAba1 u acinetobakterů [35, 51]. V případě již zmíněných genů ESBLA typu CTX-M (blaCTX-M) není důsledkem pouze mobilizace. Tyto geny jsou u svých mateřských druhů z rodu Kluyvera exprimovány v zanedbatelné kvantitě, tzn. nezpůsobují klinicky významnou rezistenci k cefalosporinům s širokým spektrem. Jestliže však dojde k transpozici ISEcp1 před gen blaCTX-M, dojde ke zvýšení jejich exprese a manifestním projevům rezistence k cefalosporinům [30, 49, 50].
Rovněž u druhu Acinetobacter baumannii byl popsán vliv inserční sekvence na expresi karbapenemáz skupiny OXA. Inserční sekvence ISAba1, která náleží do rodiny IS4, je často identifikována před genem blaOXA-23, jehož produkt, karbapenemáza OXA-23, vede ke snížené citlivosti daného kmene ke karbapenemům. Genu karbapenemázy OXA-58, jež je také typická pro acinetobaktery, rovněž často poskytují promotor inserční sekvence – již zmíněná ISAba1, dále ISAba2, ISAba3, a IS18. Tyto elementy však za mobilizaci genu blaOXA-58 pravděpodobně zodpovědné nejsou [51].
3. Transpozony
Stejně jako inserční sekvence, jsou transpozony mobilní genetické elementy schopné intracelulárního přenosu v rámci jedné molekuly, popř. mezi různými molekulami DNA (např. chromozom vs plazmid atp.) [57]. Zvláštní skupinu tvoří konjugativní transpozony, které disponují systémem pro jejich konjugativní přenos [60]. Na rozdíl od inserčních sekvencí kódují další geny, které přímo nesouvisí s jejich funkcí.
Transpozony obecně obsahují tři funkční jednotky: gen transposázy, která umožňuje vlastní transpozici, a invertované repetice na obou koncích transpozonu, které jsou – stejně jako v případě IS – transposázou rozpoznávány (obr. 5A) [57, 65].
Zvláštní skupinu transpozonů tvoří tzv. kompozitní transpozony, ohraničené dvěma inserčními sekvencemi, obvykle v opačné orientaci. Tyto IS poskytují transpozonu enzym transposázu a invertované repetice, označované někdy jako vnější konec (OE – Outside End, obvykle levá invertovaná repetice) a vnitřní konec (IE – Inside End, obvykle pravá invertovaná repetice). Struktura kompozitních transpozonů je zobrazena na obrázku 5B.
5. Transpozon. A – Obecná struktura transpozonu, tnp – gen transposázy, IRL – invertovaná repetice levá, IRR – invertovaná repetice pravá; B – kompozitní transpozon se dvěma opačně orientovanými inserčními sekvencemi na koncích, OE – vnější konec, IE – vnitřní konec. Šrafovaně jsou vyznačena místa obsahující genetickou informaci nepotřebnou k transpozici transpozonu (např. geny rezistence). <b>Fig. 5.</b> Transposon. A – General structure of the transposon, tnp – transposase gene, IRL – inverted repeatleft, IRR – inverted repeat right; B – composite transposon with two terminal inverted insertion sequences, OE – outer end, IE – inner end. There are shaded the parts containing genetic information that is not needed for the transposition of transposon (e.g. resistance genes). 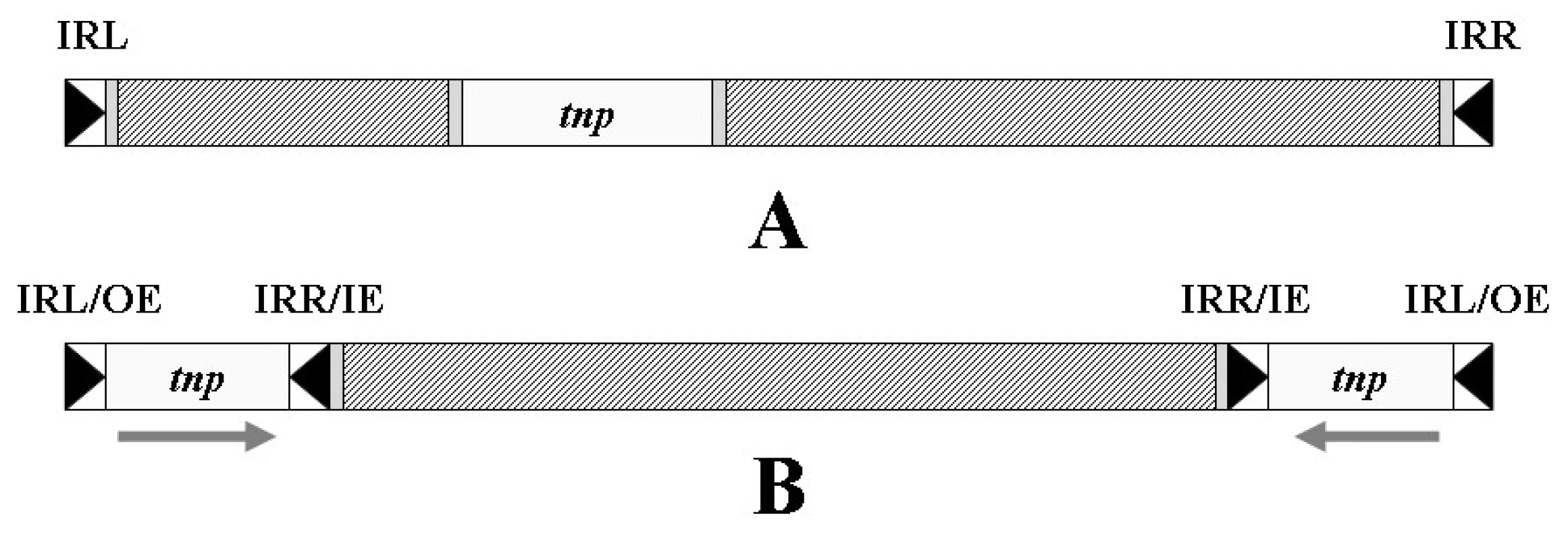
Různé transpozony se dále mohou do sebe vnořovat a vytvářet tak složité struktury [66].
Klasifikace transpozonů je, stejně jako v případě inserčních sekvencí, prováděna podle jejich struktury [57]. Databázi lze nalézt na webové stránce http://www.ucl.ac.uk/eastman/tn/.
Transpozice transpozonu probíhá v několika krocích. Obvyklý mechanismus je zobrazen na obrázku 6. V první fázi je transposáza navázána na invertované repetice. Následně jsou nekovalentně propojeny oba konce transpozonu a dochází k jeho excizi z donorové molekuly. Ve formě cirkulární, avšak kovalentně nepropojené molekuly je přenesen na cílové místo recipientní molekuly [65].
6. Transpozice transpozonu. Transpozon je znázorněn šrafovaně, tnp – transposáza, 1 – vazba transposázy na invertované repetice, 2 – synapse, 3 - excize, 4,5 – vazba transpozonu do recipientní molekuly. Podle [65]. <b>Fig. 6.</b> Transposition of the transposon. Transposon is shaded, tnp – transposase, 1 – binding of transposase to the terminal inverted repeats, 2 – synapsis, 3 - excision, 4,5 – transposon inserted into the recipient molecule. According to [65]. ![Transpozice transpozonu. Transpozon je znázorněn
šrafovaně, tnp – transposáza, 1 – vazba transposázy na invertované
repetice, 2 – synapse, 3 - excize, 4,5 – vazba transpozonu
do recipientní molekuly. Podle [65].
<b>Fig. 6.</b> Transposition of the transposon. Transposon is shaded,
tnp – transposase, 1 – binding of transposase to the terminal
inverted repeats, 2 – synapsis, 3 - excision, 4,5 – transposon
inserted into the recipient molecule. According to [65].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/459836b3af711179ff685db6c833be59.jpeg)
Kvůli nebezpečí letálních mutací způsobených nadměrnou transpozicí, je tento mechanismus regulován různými mechanismy [20], stejně jako je tomu v případě samotných inserčních sekvencí.
3.1. Konjugativní transpozony
Jedná se o neobvyklé mobilní genetické elementy, schopné intercelulárního přenosu konjugací [60]. Při excizi transpozonu z donorové molekuly DNA dojde k vytvoření kovalentně uzavřeného kruhu dvoušroubovice DNA. Tato struktura je podobná plazmidu, avšak není schopná autonomní replikace. Poté následuje konjugativní přenos jednoho vlákna DNA do recipientní buňky. Mechanismus konjugace není shodný s běžným konjugativním přenosem plazmidů, neboť na konjugativních transpozonech nejsou kódovány geny pro sex pilus a ostatní proteiny potřebné k běžnému konjugativnímu přenosu. Předpokládá se, že za konjugaci transpozonů jsou zodpovědné peptidové feromony. Po přenosu DNA dochází v obou buňkách (donoru i recipienta) k replikaci druhého vlákna DNA a opětovnému vytvoření původní kruhové formy dvoušroubovice DNA transpozonu. Ten je poté integrován do genomu v obou buňkách (viz obrázek 7).
7. Konjugativní transpozice. 1 – excize, 2 – konjugativní přenos, 3 – „replikace“, 4 – integrace. Podle [54]. <b>Fig. 7.</b> Conjugative transposition. 1 – excision, 2 – conjugative transposition, 3 – „replication“, 4 – integration. According to [54]. ![Konjugativní transpozice. 1 – excize, 2 – konjugativní
přenos, 3 – „replikace“, 4 – integrace. Podle [54].
<b>Fig. 7.</b> Conjugative transposition. 1 – excision, 2 – conjugative
transposition, 3 – „replication“, 4 – integration. According
to [54].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/ca501243d5079d04e126112e20d1e943.jpeg)
3.2. Transpozony a mobilizace genů rezistence
Transpozony jsou důležité především při mobilizaci integronů, jak je popsáno v odstavci 4.3. Například gen ESBLAblaCTX-M-9 byl nalezen na komplexním sul1 integronu, který byl pravděpodobně mobilizován transpozonem Tn402 [40]. Dalším typickým příkladem je kompozitní transpozon Tn21, který sdružuje integron třídy 1 a dále geny zodpovědné za rezistenci ke rtuti a jejím sloučeninám [31, 46]. Rovněž kompozitní transpozon ohraničený dvěma inserčními sekvencemi IS26 mobilizuje integron sdružující různé geny rezistence, např. gen pro ESBLAblaVEB-1, geny aminoglykosidové adenyltransferázy, a arr 2 – gen pro ADP-ribosyltransferázu inaktivující rifampicin, inherentní u Mycobacterium smegmatis, atp. [37].
U grampozitivních mikrobů se jedná o významné mobilní elementy. Řídí přenos van genů, jejichž produkty způsobují rezistenci k vankomycinu a genů zopovědných za rezistenci k aminoglykosidům u enterokoků [12, 13, 56]. Jako příklad lze uvést kompozitní transpozony ohraničené inserčními sekvencemi IS1216V (Tn1546) a IS256 (Tn5384) [56]. Transpozon Tn1546 byl rovněž nalezen v USA u meticilin rezistentních kmenů Staphylococcus aureus současně rezistentních k vankomycinu (VRSA) [8]. Na transpozonu Tn5406 u stafylokoků byly popsány determinanty způsobující rezistenci k streptograminu A včetně dalfopristinu [22]. Dalším příkladem může být mobilizace genů zodpovědných za rezistenci streptokoků k makrolidům erm (ribosomální metyláza) a geny mef kódující efluxní systém. Tyto geny byly nalezeny na různých transpozonech (Tn1207, Tn2009, Tn2010, atp.) [19].
4. Integrony
Integrony jsou genetické elementy sdružující otevřené čtecí rámce a konvertující je do funkčních genů – poskytují jim obvykle silné promotory [1, 5, 14, 34]. Každý integron obsahuje enzym integrázu, vazebné místo (attI) [45] a promotor (Pc), zajišťující expresi integrovaných genových kazet (struktura integronu viz obr. 8) [1, 5, 14, 34]. Integrony nejsou samy o sobě mobilními elementy, avšak mohou být mobilizovány, obvykle jako součást transpozonů (viz odstavec 4.3) [11, 38, 44, 46, 52]. Z biologického hlediska lze integrony řadit do dvou hlavních skupin – superintegrony (často označované zkratkou SI) a integrony sdružující geny rezistence k antibiotikům (se zkratkou RI) [14, 58]. RI jsou některými autory popisovány jako mobilní integrony, kvůli skutečnosti, že většina těchto struktur byla nalezena na mobilních elementech. Pokud nebude v následujících odstavcích zmíněno jinak, bude pojednáváno o této skupině integronů (RI).
Významnou složkou, která se podílí na formování integronu, jsou genové kazety. Ty jsou tvořeny otevřeným čtecím rámcem a sekvencí, která je obvykle označována jako attC, případně jako 59-be (be – Base Element, jehož velikost však může být různá – od 57 do 141 bp [1, 14, 34, 61]). Genové kazety jsou mobilními elementy cirkulární DNA, které se mohou vyskytovat volně v cytoplazmě. Narozdíl od plazmidů však nejsou schopny autonomní replikace. Integráza zajišťuje integraci volných genových kazet do struktury integronu na pozici attI. Je zároveň schopna vzájemné rekombinace genových kazet mezi různými integrony. Vazebnou pozicí integrázy na genové kazetě je místo attC [5, 9, 11, 47].
Integráza je enzym, který není endonukleázou, ale chová se podobně jako topoizomeráza, tzn. k úplné integraci genové kazety dochází až při následné replikaci molekuly DNA [5, 11, 14]. Mechanismus integrace genových kazet je zobrazen na obr. 8.
8. Schéma integronu a integrace genových kazet u integronu třídy 1. Int1 – gen pro integrázu, P<sub>int</sub> – promotor genu pro integrázu, P<sub>1</sub> , P<sub>2</sub> – promotory genových kazet integrovaných v integronu, attI1 – vazebné místo integrázy na integronu, attc – vazebné místo integrázy na genové kazetě, 5’CS – 5’ konzervativní sekvence, 3’CS – 3’ konzervativní sekvence. Volně podle [5]. <b>Fig. 8.</b> Schematic representation of the integron and gene cassette integration in the class 1 integron. Int1 – integrase gene, P<sub>int</sub> – integrase gene promoter, P<sub>1</sub> , P<sub>2</sub> – promoters of the gene cassettes integrated into the integron, attI1 – integrase binding site in the integron, attc – integrase binding site in the gene cassette, 5’CS – 5’ conserved sequence, 3’CS – 3’ conserved sequence. Based on [5]. ![Schéma integronu a integrace genových kazet u integronu třídy 1. Int1 – gen pro integrázu, P<sub>int</sub> – promotor genu pro
integrázu, P<sub>1</sub> , P<sub>2</sub> – promotory genových kazet integrovaných v integronu, attI1 – vazebné místo integrázy na integronu, attc –
vazebné místo integrázy na genové kazetě, 5’CS – 5’ konzervativní sekvence, 3’CS – 3’ konzervativní sekvence. Volně podle [5].
<b>Fig. 8.</b> Schematic representation of the integron and gene cassette integration in the class 1 integron. Int1 – integrase gene, P<sub>int</sub>
– integrase gene promoter, P<sub>1</sub> , P<sub>2</sub> – promoters of the gene cassettes integrated into the integron, attI1 – integrase binding site
in the integron, attc – integrase binding site in the gene cassette, 5’CS – 5’ conserved sequence, 3’CS – 3’ conserved sequence.
Based on [5].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/fb8a60cf3aa7a5ee76ec4cf997964f86.jpeg)
4.1. Klasifikace integronů
Jak bylo zmíněno na začátku, lze integrony členit na superintegrony, které čítají mnoho desítek genových kazet a jsou součástí chromozomu některých bakterií (Vibrio cholerae, Vibrio spp., Pseudomonas spp., Schewanella spp., Treponema denticola, Xanthomonas spp. atd. [14]). Vzhledem k jejich velikosti je jejich mobilizace velmi obtížná a nebyla dosud popsána. SI se od RI obvykle liší integrázou, která má však stejnou funkci jako u RI [14]. U SI Vibrio cholerae se vyskytují opakující se sekvence podobné 59-be, jež jsou označovány VCRs (V. cholerae Repetitive DNA Sequences) [3, 14].
Do současnosti bylo popsáno pět tříd integronů RI. Tyto integrony se opět liší strukturou integrázy a strukturou 3’ konzervativní sekvence (3’CS) [34]. Nejčastější jsou integrony třídy 1. Podskupinu těchto integronů tvoří sul1 komplexní integrony, obsahující v 3’CS regionu gen determinující rezistenci k sulfonamidům a inserční sekvenci ISCR1 [37]. Integrony třídy 2 byly popsány jako součást transpozonu skupiny Tn7 [34, 64]. Tyto integrony se vyznačují nefunkční integrázou. Pravděpodobně z tohoto důvodu bylo popsáno pouze šest genových kazet nesených na integronech této třídy. Nedávno však byl popsán nález integronu této třídy s funkční integrázou [33]. Integrony třídy 3 rovněž nejsou běžné a byly nalezeny například v souvislosti s lokalizací genové kazety pro metalo-β-laktamázy IMP a karbapenemázu GES [11]. Dosud byl popsán pouze jediný integron třídy 4 u Vibrio cholerae a třídy 5 u V. salmonicida [34].
Ač se aminokyselinová struktura integráz a vazebných míst attI těchto tříd integronů liší, byly na různých integronech prokázány stejné genové kazety. To ukazuje na možnost vzájemného toku genových kazet mezi integrony různých tříd [34].
Internetovou databázi integronů lze nalézt na stránce http://integrall.bio.ua.pt [36]. V této databázi je možné zobrazit strukturu jednotlivých integronů, přístupový kód databáze GenBank, druh, u něhož byl daný integron nalezen, a typ integrázy. V době přípravy tohoto textu čítala databáze 5 313 položek integronů izolovaných ze 173 různých bakteriálních druhů z 93 rodů. U těchto integronů bylo identifikováno 6 711 genových kazet.
4.2. Evoluce integronů a genových kazet
O evoluci integronů a genových kazet je dosud známo poměrně málo. Některé teorie byly vysloveny, avšak nejsou obecně přijímány [34, 55]. Na základě nízké homologie jednotlivých integráz se předpokládá, že se vyskytují déle než 60 let, tedy doby používání antibiotik. K jejich rozšíření však selekční tlak antibiotik může výrazně přispívat.
Vzhledem k tomu, že jsou SI součástí chromozomů některých bakteriálních druhů již velmi dávno (předpokládá se až stovky milionů let) a některé genové kazety objevené na SI byly současně nalezeny na RI, lze se domnívat, že původ RI je v superintegronech [14, 15, 59]. Pro tuto hypotézu svědčí zároveň fakt, že integrony třídy 1 byly nalezeny i u druhu Vibrio cholerae [14]. Z genových kazet zodpovědných za rezistenci, které byly nalezeny současně na superintegronech i RI lze jmenovat např. qnr – gen kódující protein zodpovědný rezistenci k fluorochinolonům [15], případně se jednalo o kazety velmi podobné [14, 67].
4.3. Mobilizace integronů
Všechny RI byly popsány jako součást mobilních elementů [34]. Proto byly zpočátku samotné integrony považovány za mobilní elementy [34]. Integrony tříd 1 až 3 bývají součástí transpozonů (Tn21 – integron třídy 1, Tn7 – integron třídy 2). Velmi zajímavou strukturu tvoří integron třídy 4, který byl nalezen jako součást konjugativního transpozonu označovaného SXT element [34]. Integron třídy 5 je součástí kompozitního transpozonu neseného na plazmidu [34].
Nedávno byly popsány struktury nazývané jednotky mobilizující integron (IMU – Integron Mobilization Unit) v souvislosti s genem pro β-laktamázou GES (ESBLCARBA-A [18]) [48]. Gen blaGES-5, lokalizovaný na defektním integronu třídy 1, byl ohraničen dvěma identickými elementy dlouhými 288 bp v opačné orientaci (viz obr. 9). Tato struktura byla podobná kompozitnímu transpozonu, byly zde identifikovány invertované repetice typické pro inserční sekvence, avšak element nekódoval žádný otevřený čtecí rámec (ORF), tedy žádnou transposázu. Analýzou invertovaných repetic byla zjištěna podobnost s ISSod9 a zároveň experimentálně prokázáno, že transposáza této IS (kódovaná v jiné oblasti) umožňuje transpozici celé této struktury.
9. Jednotky mobilizující integron (IMU) v souvislosti s genem β-laktamázy GES-5. Podle [48]. <b>Fig. 9. </b>Integron mobilization unit (IMU) as a source of mobility of the beta-lactamase gene GES-5. According to [48]. ![Jednotky mobilizující integron (IMU) v souvislosti
s genem β-laktamázy GES-5. Podle [48].
<b>Fig. 9. </b>Integron mobilization unit (IMU) as a source of mobility
of the beta-lactamase gene GES-5. According to [48].](https://www.prolekare.cz/media/cache/resolve/media_object_image_small/media/image/411c5505efea7096ad13ebfc3a9902ae.jpeg)
4.4. Integrony a sdružená rezistence
Integrony jsou nalézány u celé řady gramnegativních bakterií, avšak jejich význam narůstá s šířením metalo-β-laktamáz a dalších získaných karbapenemáz, jejichž geny (resp. genové kazety) jsou většinou na integronech kódovány. S integrony jsou spjaty i jiné determinanty antibiotické rezistence, jejichž přehled lze nalézt v přehledové práci [14], jako např. β-laktamázy GES, IMP, VIM, OXA atp., acetyl - a adenyltransferázy zodpovědné za rezistenci k aminoglykosidům, proteiny Qnr chránící gyrázu/topoizomerázu před účinkem fluorochinolonů a další [14, 47].
Exprese genových kazet sdružených na integronu může být různá. Většinou je nejintenzivněji exprimována první genová kazeta a exprese dalších genových kazet bývá nižší [27]. Přesto je funkce integronů jako expresních jednotek v epidemiologii antibiotické rezistence velmi významná, neboť ideálně umožňuje současnou expresi všech integrovaných genových kazet najednou [27]. Důsledkem toho je sdružená selekce rezistence ke všem antibiotikům, jejichž genové kazety jsou integrovány v integronu. Detailní popis integronů je významným prediktorem ko-selektujících antibiotik.
Příkladem může být komplexní sul1 integron popsaný u kmenů Klebsiella pneumoniae produkujících AmpC β-laktamázu DHA-1 [70, 71], který byl detekován i u těchto producentů izolovaných v České republice [Hrabák et al., nepublikovaná data]. Tento velmi atypický integron kóduje další determinanty rezistence, jako například aac(6’)-Ib – acetyltransferázu inaktivující některé aminoglykosidy, OXA-1 (v práci [70] je OXA-1 popisována synonymem OXA-30) – β-laktamázu hydrolyzující peniciliny a aminopeniciliny včetně kombinací s inhibitory a ADP-ribosyltransferázu (kazeta arr3) způsobující rezistenci k rifampicinu. Při infekcích vyvolaných těmito producenty vede podání kteréhokoliv ze zmíněných antibiotik k selekci rezistence k ostatním skupinám, jejichž determinanty jsou sdruženy na integronu.
5. Plazmidy
Plazmidy jsou molekuly dvouvláknové DNA uzavřené do kruhu a schopné samostatné replikace v cytoplazmě bakterií. Geny nesené na plazmidech nejsou pro bakterii esenciální, tedy i plazmidy jsou pro základní funkce bakterie postradatelné [6, 62].
Přirozeně se vyskytující plazmidy mají velký rozptyl vlastností jak do velikosti, tak i počtu kopií. Molekula může mít velikost až 400 kb a podobně i počet kopií plazmidu v buňce sahá od jediného exempláře až po stovky kopií. Počet kopií je stálou charakteristikou určitého plazmidu za konstantních podmínek, kdy další jeho replikace je omezená na plazmidu kódovaným systémem [41, 62].
Plazmidy obsahují nejen geny nutné pro zahájení a kontrolu vlastní replikace (obvykle negativní zpětná vazba), ale také geny zajišťující trvalou přítomnost plazmidů u daného kmene – například gen synchronizující replikaci plazmidu s buněčným dělením nebo geny umožňující konjugaci [6].
Plazmidy lze klasifikovat mnoha různými způsoby. Jednou z možností je klasifikace podle funkce v buňce. Rozeznáváme 5 hlavních funkčních skupin plazmidů: fertilní plazmidy (F-plazmidy), rezistence (R), Col-plazmidy obsahující geny pro koliciny – substance smrtící jiné bakterie, degradativní plazmidy umožňující pohlcování nezvyklých látek a virulentní plazmidy, kódující faktory patogenity bakterií. Jeden plazmid může náležet zároveň do několika různých funkčních skupin [62].
5.1. Inkompatibilita plazmidů
Dalším systémem je klasifikace podle inkompatibility, což je neschopnost dvou plazmidů být trvale přítomný u stejné buněčné linie. Je to důsledek příbuznosti - sdílení replikace, replikačních kontrolních mechanismů, nebo dělících elementů [41, 54].
Inkompatibilita může být symetrická, neboli oba plazmidy jsou ztráceny se stejnou pravděpodobností, nebo vektorová, kdy jeden z plazmidů je ztrácen s větší pravděpodobností než druhý, či zcela exkluzivně. Předpokládá se, že mechanismy podílející se na těchto dvou typech inkompatibility jsou odlišné. U symetrického typu se zřejmě jedná o neschopnost vyrovnat kolísání v počtu vznikajících kopií různých plazmidů v důsledku náhodného výběru jednotlivých entit pro replikaci a dělení. Při vektorové inkompatibilitě většinou interferují s replikací fragmenty klonovaného plazmidu obsahující geny pro kontrolu replikace nebo mutace v systému kontrolujícím replikaci u přímo regulovaných plazmidů [41, 54].
Na základě inkompatibility byly definovány 4 základní skupiny:
- skupina IncF, zahrnující: IncF, IncS, IncC, IncD, IncJ;
- skupina IncP, zahrnující: IncP, IncU, IncM, IncW;
- skupina Ti plazmidů, kam jsou řazeny IncX, IncH, IncN, IncT;
- skupina IncI, k níž náleží IncI, IncB a IncK [6].
5.2. Horizontální přenos plazmidů
Proces horizontálního přenosu plazmidů je nazýván konjugací. Jedná se o přenos kontaktní a na energii závislý, tedy aktivní. Konjugace plazmidu je nejdůležitějším způsobem výměny genetické informace u bakterií, v určitém ohledu srovnatelná se sexuálním rozmnožováním u eukaryot, ačkoli zde nesplývají gamety, nevzniká zygota, přesto dochází k výměně genetické informace mezi dvěma individui, přičemž se nemusí jednat o jedince stejného druhu. U rodů Agrobacterium a Rhizobium byl dokonce zaznamenán transfer informace z konjugativního plazmidu do genomu eukaryotních rostlinných buněk [6, 61].
Jako konjugativní plazmid je označován takový plazmid, na němž je kódován celý bílkovinný aparát nutný pro transfer z jedné buňky do druhé při konjugaci. Plazmidy schopné konjugace pouze za účasti pomocného konjugativního plazmidu se nazývají mobilizovatelné. Zatímco plazmidy konjugativní bývají obvykle velké (> 30 kb) s malým počtem kopií v cytoplazmě, mobilizovatelné jsou malé (pod < 15 kb) s vysokým počtem kopií [16].
Při konjugaci se nejprve rozštěpí transferovaná DNA v místě označovaném oriT enzymem relaxázou, která pak zůstává v kovalentní vazbě na oriT DNA. Vzniklý nukleoproteinový komplex je transportován do buňky recipienta bílkovinným exportním mechanismem T4SS (Type IV secretion system), vlastní DNA je pak aktivně vpravena do buňky proteinem T4CP (type IV coupling protein) [16].
Všechny plazmidy schopné transferu do recipientní buňky musí obsahovat MOB oblast nutnou k mobilizaci, přičemž konjugativní plazmidy disponují navíc proteinem T4SS, který umožňuje vytvoření a funkci “spojovacího“ kanálu [16].
Na základě sekvenční analýzy relaxáz je možné rozdělit konjugativní systémy plazmidů do šesti MOB rodin: MOBF, MOBH, MOBQ, MOBC, MOBP a MOBV. Sekvenace genů kódujících relaxázy jsou využívány k fylogenetickým studiím a jako jedna z možností klasifikace plazmidů [16].
5.3. Plazmidy nesoucí geny rezistence
Běžně používaná metoda typizace plazmidů pomocí PCR replikonového typování je relativně mladou metodou, proto epidemiologická data o typech plazmidů nesoucích geny rezistence jsou dosud neúplná. Přesto se zdá, že geny některých širokospektrých β-laktamáz (ESBL zde v obecném smyslu [18]), případně ostatních genů rezistence (např. aac(6’)-Ib-cr) jsou kódovány na plazmidech stejných typů – např. blaCMYna plazmidech IncA/C, IncI1, a IncN, blaVIMna plazmidech IncA/C a IncN [7]. Rovněž při komplexní analýze kmenů E. coli produkujících ESBLA typu CTX-M-15 izolovaných v různých zemích světa byly zjištěny plazmidy IncFII, u některých kmenů spolu s replikony FIA a FIB. Na těchto plazmidech byly zároveň neseny geny blaOXA-1 a aac(6’)-Ib-cr [10]. Stejně tak i v České republice byly ESBLA nalezeny především na plazmidech IncF, IncA/C a IncI1 [24].
6. SCC (Staphylococcal Cassette Chromosome)
Stafylokokové chromozomální kazety jsou nalézány u různých druhů stafylokoků (Staphylococcus aureus, S. hominis, S. haemolyticus, S. epidermidis, atp.) [25, 63]. Nejznámější je stafylokoková chromozomální kazeta mec (SCCmec), která kóduje gen mecA zodpovědný za expresi alternativního typu PBP – PBP2a, který má sníženou afinitu k meticilinu a způsobuje tak rezistenci k tomuto antibiotiku (MRSA – meticilin rezistentní Staphylococcus aureus) [25, 42].
SCCmec je složena ze tří podjednotek – mec genový komplex, který sdružuje samotný mecA gen, a geny zodpovědné za regulaci jeho exprese. Ty bývají v některých případech přerušeny inserčními sekvencemi IS431 a IS1272 [26, 29]. Dalším komplexem je ccr (Cassette Chromosome Recombinase) genový komplex, na němž je kódován gen, případně geny pro místně specifickou rekombinázu. Třetí podjednotkou je tzv. J-komplex (Junkyard), který sdružuje další geny, např. geny zodpovědné za rezistenci k ostatním antibiotikům [26, 42, 43, 69].
Rekombináza (rekombinázy) kódované na SCCmec rozpoznávají na tomto elementu místo označované jako attB. Místo, kde dochází na recipientní molekule DNA k integraci kazety se nazývá ISS (Integration Site Sequence). U druhu Staphylococcus aureus je toto místo přítomné u 3’ konce otevřeného čtecího rámce orfX. Teoretická sekvence ISS je úsek DNA dlouhý 18 bp – jedná se tudíž o poměrně specifickou rekombinaci [28, 42].
7. Význam analýzy mobilních elementů v epidemiologii antibiotické rezistence
Analýza mobilních elementů DNA má v epidemiologii antibiotické rezistence nezastupitelné místo. Základním důsledkem lokalizace různých genů zodpovědných za rezistenci k odlišným skupinám antibiotik na stejném mobilním elementu (resp. integronu) je sdružená rezistence ke všem těmto antibiotikům. Rovněž podávání antibiotika z jedné skupiny vede v takovém případě ke sdružené selekci rezistence k ostatním skupinám. Proto tyto analýzy umožňují efektivně optimalizovat antibiotickou politiku. Podobnou analýzu nelze relevantně provést žádným statistickým modelem.
Druhým důsledkem je evoluční analýza rezistence v bakteriálních populacích. Podrobnou analýzou mechanismů mobilizace lze přesně stopovat původ rezistence, cesty přenosu, klonální příbuznost, geografické aspekty šíření rezistence, atd. Jako příklad lze uvést dva případy VRSA ve Spojených státech (Michiganu a Pensylvanii), u nichž došlo k přenosu transpozonu Tn1546 nesoucího vanA gen a ostatní geny van z enterokoků. Analýzou PFGE bylo zjištěno, že se jedná o tentýž klon, avšak analýza zmíněných mobilních elementů dokázala, že se jedná o dva zcela nepříbuzné izoláty [8]. Obdobným příkladem jsou i enterokoky, kde metoda PFGE, ve vyspělých státech běžně používaná jako součást epidemiologického šetření, nemá dostatečnou diskriminační schopnost a teprve analýza mobilních elementů nesoucích příslušné geny rezistence umožňuje získat validní data [12, 21].
8. Závěr
Šíření determinant rezistence na mobilních elementech DNA, případně jejich sdružování na integronech je významnější než rezistence vzniklá mutacemi. Pochopení této problematiky je rovněž nezbytné pro efektivní prevenci vzniku a šíření rezistence k antibiotikům. Přestože tento souhrnný článek poskytuje recentní informace o této problematice, lze očekávat, že ke zpřesňování našich znalostí uvedených principů, případně objevování nových mechanismů mobilizace bude docházet neustále. Informace zde uvedené proto nelze považovat za neměnné dogma.
Poděkování:
Autoři děkují RNDr. Pavle Urbáškové, CSc., MUDr. Heleně Žemličkové, Ph.D. a prim. MUDr. Tamaře Bergerové za kritické přečtení a cenné připomínky k rukopisu.Práce byla podpořena granty MŠMT č. 2E08003 a IgAMZ NS 9717-41/2008.
Ing. Jaroslav Hrabák, Ph. D.
Ústav mikrobiologie LF UK a FN v Plzni
Dr. E. Beneše 13
305 99 Plzeň
e-mail: Jaroslav. Hrabak@lfp.cuni.cz
Sources
1. Bennett, P. M. Integrons and gene cassettes: a genetic construction kit for bacteria, J Antimicrob Chemother, 1999, 43, 1-4.
2. Bennett, P. M., Livesey, C. T., Nathwani, D., Reeves, D. S. at al. An assessment of the risks associated with the use of antibiotic resistance genes in genetically modified plants: report of the Working Party of the British Society for Antimicrobial Chemotherapy, J Antimicrob Chemother, 2004, 53, 418-431.
3. Biskri, L., Biuvier, M., Guérount, A-M., Boisnard, S., Mazel, D. Comparative study of class 1 integron and Vibrio cholerae superintegron integrase activities, J Bacteriol, 2005, 187, 1740-1750.
4. Brookfield, J. F. Evolutionary Genetics: Mobile DNAs as sources of adaptive change? Current Biol, 2004, 14, 344-345.
5. Carattoli, A. Importance of integrons in the diffusion of resistance, Vet Res, 2001, 32, 243-259.
6. Carattoli, A. Plasmid mediated Antimicrobial Resistence in Salmonella enterica . Curr Issues Mol Biol, 2003, 5, 113-122.
7. Carattoli, A., Miriagou, V., Bertini, A., Loli, A. et al. Replicon typing of plasmids encoding resistance to newer β-lactams. , 2006, 12, 1145-1148.
8. Clark, N. C., Weigel, L. M., Patel, J .B., Tenover, F. C. Comparison of TN1546-like elements in vancomycin-resistant Staphylococcus aureus isolates from Michigan and Pensylvania. Antimicrob Agents Chemother, 2005, 49, 470-472.
9. Collis, Ch. M., Recchia, G. D., Kim, M-J., Stokes, H. W., Hall, R. Efficiency of recombination reactions catalyzed by class 1 integron integrase IntI1, J Bacteriol, 2001, 183, 2535-2542.
10. Coque, T. M., Novais, A., Carattoli, A., Poirel, L. et al. Dissemination of clonally related Escherichia coli strains expressing extended-spectrum β-lactamase CTX-M-15. Emerg Infect Dis 2008, 14, 195-200.
11. Correia, M., Boavida, F., Grosso, F., Salgado, M. J. at al. Molecular characterization of a new class 3 integron in Klebsiella pneumoniae, Antimicrob Agents Chemother, 2003, 47, 2838-2843.
12. Darini, A. L., Palepou, M.-F., Woodford N. Effect of the movement of insertion sequence on the structure of vanA glycopeptide resistance elements in Enterococcus faecium, Antimicrob Agents Chemother, 2000, 44, 1362 – 1364.
13. Feizabadi, M. M., Shokrzadeh, L., Sayady, S., Asadi S. Transposon Tn5281 is the main distributor of the aminoglycoside modifying enzyme gene among isolates of Enterococcus faecalis in Tehran hospitals, Can J Microbiol, 2008, 54, 887-890.
14. Fluit, A. C., Schmitz, F.-J. Resistance integrons and super-integrons, Clin Microb Infect Dis, 2004, 10, 272-288.
15. Fonseca, E. L., Freitas, F.d S.,Vieira, V. V., Vicente, A. C. P. New qnr gene cassettes associated with superintegron repeats in Vibrio cholerae O1, Emerg Infect Dis, 2008, 14, 1129-1131.
16. Garcillán-Barcia, M. P., Francia, M. V., de la Cruz, F. The diversity of conjugative relaxases and its application in plasmid classification. FEMS Microbiol Rev, 2009, 33, 657-687.
17. Garza-Ramos, U., Davila, G., Gonzalez, V., Alpuche-Aranda, C. et al. The blaSHV-5 gene is encoded in a compound transposon duplicated in tandem in Enterobacter cloacae, Clin Microb Infect Dis, 2009, Epub ahead of print.
18. Giske C. G., Sundsfjord A.S., Kahlmeter G, Woodford N, Nordmann P. et al. Redefining extended-spectrum β-lactamases: balancing science and clinical need. J Antimicrob Chemother, 2009, 63, 1-4.
19. delGrosso, M., Camilli, R., Ianelli, F., Pozzi, G. The mef(E)-carrying genetic element (mega) of Streptococcus pneumoniae: insertion sites and association with other genetic elements, Antimicrob Agents Chemother, 2006, 50, 3361 – 3366.
20. Haniford, D. B. Transposome dynamics and regulation in Tn10 transposition, Crit Rev Biochem Mol Biol, 2006, 41, 407-424.
21. Handwerger, S., Skoble, J. Identification of chromosomal mobile element confering high-level vancomycin resistance in Enterococcus faecium, Antimicrob Agents Chemother, 1995, 39, 2446 – 2453.
22. Haroche, J., Allignet J., El Solh, N. Tn5406, a new Staphylococcal transposon conferring resistance to streptogramin A and related compounds including dalfopristin, Antimicrob Agents Chemother, 2002, 46, 2337-2343.
23. Honl J, Bukovský J. Zentralblatt für Bakteriologie, Parasitenkunde, Infektionskrankheiten und Hygiene Abteilung 1899, 126, 305.
24. Hrabák, J., Empel, J., Bergerová, T., Fajfrlík, K. et al. International clones of Klebsiella pneumoniae and Escherichia coli with extended-spectrum β-lactamases (ESBLs) in a Czech hospital, J Clin Microb, 2009, 47, 3353-3359.
25. Ito, T., Katayama, Y., Hiramatsu K. Cloning and nucleotide sequence determination of the entire mec DNA of pre-methicillin-resistant Staphylococcus aureus N315, Antimicrob Agents Chemother, 1999, 43, 1449-1458.
26. Ito, T., Ma, X. X., Takeuchi, F., Okuma, K. et al. Novel type V Staphylococcal cassette chromosome mec driven by a novel cassette chromosome recombinase, cvrC, Antimicrob Agents Chemother, 2004, 48, 2637-2651.
27. Jacquier, H., Zaoui, C., Sanson-le Pors, M-J., Mazel, D. Translation regulation of integrons gene cassette expression by the attC sites, Mol Microbiol, 2009, 72, 1475-1486.
28. Jansen, W. T. M., Beitsma, M. M., Koeman, C. J., van Wamel, W. J. B. et al. Novel mobile variants of staphylococcal cassette chromosome mec in Staphylococcus aureus, Antimicrob Agents Chemother, 2006, 50, 2072-2078.
29. Kobayaschi, N., Urasawa, S., Uehara N., Watanabe, N. Distribution of insertion sequence-like element IS1272 and its position relative to methicillin resistance genes in clinically important staphylococci, Antimicrob Agents Chemother, 1999, 43, 2780-2782.
30. Lartigue, M-F., Poirel, L., Aubert, D., Nordmann, P. In vitro analysis of ISEcp1B-mediated mobilization of naturally occurring β-lactamase gene blaCTX-M of Kluyvera ascorbata, Antimicrob Agents Chemother, 2006, 50, 1282-1286.
31. Liebert, C.A., Hall, R. M, Summers A. O.Transposon Tn21, flagship of the floating genome, Microbiol Mo. Biol Rev, 1999, 63, 507-522 .
32. Mahillon, J., Chanpodler, M. Insertion sequences, Microbiol Mol Biol Rev 1998, 62, 725-774.
33. Márquez, C., Labbate, M., Ingold, A. J., Chowdhury, P. R. at al. Recovery of a functional class 2 integron from an Escherichia coli strain madiating urinary tract infection, Antimicrob Agents Chemother, 2008, 52, 4153-4154.
34. Mazel, D. Integrons: agents of bacterial evolution, Nature Rev Microbiol, 2006, 4, 608-620.
35. Mugnier, P. D., Poirel, L., Nordmann, P. Functional analysis of insertion sequence ISAba1, responsible for genomic plasticity of Acinetobacter baumannii, J.Bacteriol, 2009, 191, 2414-2418.
36. Moura, A., Soares, M., Pereira, C., Leitčo, N. et al. INTEGRALL: a database and search engine for integrons, integrases and gene cassettes. Bioinformatics, 2009, 25, 1096–1098.
37. Naas, T., Aubert, D., Lambert, T., Nordmann, P. Complex genetic structures with repeated elements, a sul-type class 1 integron, and the blaVEB extended-spectrum β-lactamase gene, Antimicrob Agents Chemother, 2006, 50, 1745-1452.
38. Naas, T., Mikami, Y., Imai, T., Poirel, L. et at. Characterization of In53, a class 1 plasmid - and composite transposon-located integron of Escherichia coli which carries an unusual array of gene cassettes, J Bacteriol, 2001, 183, 235-249.
39. Nordmann, P., Poirel, L. Emergence of plasmid-mediated resistance to quinolones in Enterobacteriaceae, J Antimicrob Chemother 2005, 56, 463-469.
40. Novais, A., Cantón, R., Valverde, A., Machado, E. at al. Dissemination and persistence of blaCTX-M-9 are linked to class 1 integrons containing CR1 associated with defective transposon derivatives from Tn402 located in early antibiotic resistance plasmids of IncHI2, IncP1-@, and IncFI groups, Antimicrob Agents Chemother, 2006, 50, 2741-2750.
41. Novick, R. P. Plasmid incompatibility. Microbiol Rev, 1987, 51, 381-395.
42. Oliveira, D. C., Milheirićo, C., de Lencastre, H. Redefining a structural variant of staphylococcal cassette chromosome mec, SCCmec Type VI, Antimicrob Agents Chemother 2006, 50, 3457-3459.
43. Oliveira, D. C., Wu, S. W., de Lencastre H. Genetic organization of the downstream of the mecA element in methicillin-resistant Staphylococcus aureus isolates carrying different polymorphisms of this region, Antimicrob Agents Chemother, 2000, 44, 1906-1910.
44. Partridge, S. R., Brown, H. J., Stokes, H. W., Hall, R. M. Transposons Tn1696 and Tn21 and their integrons In4 and In2 have independent origins, Antimicrob Agents Chemother, 2001, 45, 1263-1270.
45. Partridge, S. R., Recchia, G. D., Scaramuzzi, C., Collis, CH. M. at al. Definition of the attl1 site of class 1 integrons, Microbiol, 2000, 146, 2855-2864.
46. Partridge, S. R., Recchia, G. D., Stokers, H. W., Hall, R. M. Family of class 1 integrons related to In4 from Tn1696, Antimicrob Agents Chemother, 2001, 45, 3014-3020.
47. Peters, E. D. J., Leverstein-van Hall, M. A., Box, A. T. A., Verhoef, J., Fluit, A. C. Novel gene cassettes and integrons, Antimicrob Agents Chemother, 2001, 45, 2961-2964.
48. Poirel, L., CarrĎr, A., Pitout, D., Nordmann, P. Integron mobilization unit as a source of mobility of antibiotic resistance genes, Antimicrob Agents Chemother, 2009, 53, 2492-2498.
49. Poirel, L., Decousser, J-W., Nordmann, P. Insertion sequence ISEcp1B is involved in expression and mobilization of a blaCTX-M β-lactamase gene, Antimicrob Agents Chemother, 2003, 47, 2938-2945.
50. Poirel, L., Lartigue, M-F., Decousser, J-W., Nordmann, P. ISEcp1B-mediated transposition of blaCTX-M in Escherichia coli, Antimicrob Agents Chemother, 2005, 49, 447-550.
51. Poirel, L., Nordmann, P. Carbapenem resistance in Acinetobacter baumannii: mechanisms and epidemiology, 2006, Clin Microb Infect Dis, 12, 826-836.
52. Post, V., Recchia, G. D., Hall, R. M. Detection of gene cassettes in Tn402-like class 1 integrons, Antimicrob Agents Chemother, 2007, 51, 3467-3468.
53. Quiroga, M. P., Andres, P., Petroni, A., Soler Bistué, A. J. C. et al. Complex class 1 integrons with diverse variable regions, including aac(6’)-Ib-cr, and a novel allele, qnrB10, associated with ISCR1 in clinical enterobacterial isolates from Argentina, Antimicrob Agents Chemother, 2007, 51, 4466-4470.
54. Rawlings, D. E., Tietze E. Comparative biology of IncQ and IncQ-like plasmids, Microb Mol Biol Rev 2001, 65, 481-496.
55. Recchia, G. D., Hall, R. M. Origins of the mobile gene cessettes found in integrons, Trends Microbiol, 1997, 5, 389-394.
56. Rice, L.B., Carias L. L., Marshall, S. H. Tn5384, a composite enterococcal mobile element conferring resistance to erythromycin and gentamicin whose end are directly repeated copies of IS256, Antimicrob Agents Chemother, 1995, 39, 1147-1153.
57. Roberts, A. P., Chanpodler, M., Courvalin, P., Guédon, G. et al. Revised nomenclature for transposable genetic elements. Plasmid, 2008, 60, 167-173.
58. Rowe-Magnus, D. A., Guérount, A-M, Mazel, D. Super-integrons, Res Microbiol, 1999, 150, 641-651.
59. Rowe-Magnus, D. A., Guérount, A-M, Mazel D. Bacterial resistance evolution by recruitment of super-integron gene cassettes, Mol Micobiol, 2002, 43, 1657-1669.
60. Salyers, A. A., Shoemaker, N. B., Stevens, A. M., Li, L-Y. Conjugative transposons: an unusual and diverse set of integrated gene transfer elements, Microbiol Rev, 1995, 59, 579-590.
61. Segal, H., Francia, M. V., Lobo, G. J. M., Elisha, G. Reconstuction of an active integron recombination site after integration of gene cassette at a secondary site, Antimicrob Agents Chemother, 1999, 43, 2538-2541.
62. Seifert, J. L. Defining the mobilome, In: Gogarten, M.B., Gogarten, J.P., Olendzenski, L. (Ed.), Horizontal gene transfer: genomes in flux, Humana press, New York, 2009.
63. Shore, A. C., Rossney, A. S., O’Connell, B., Herra, C. M. et al. Detection of Staphylococcal cassette chromosome mec-associated DNA segments in multiresistant methicillin-susceptible Staphylococcus aureus (MSSA) and identification of Staphylococcus epidermidis ccrAB4 in both methicillin-resistant S. aureus and MSSA, Antimicrob Agents Chemother, 2008, 52, 4407-4419.
64. Solberg, O. D., Ajiboye, R. M., Riley, L. W. Origin of class 1 and 2 integrons and gene cassettes in a population-based sample of uropathogenic Escherichia coli, J Clin Microb 2006, 44, 1347-1351.
65. Steiniger-White, M., Rayment, I., Reznikoff, W. S. Structure/function insights into Tn5 transposition, Curr Opin Struct Biol 2004, 14, 50-57.
66. Stokes, H. W., Elbourne, L. D. H., Hall, R. M. Tn1403, a multiple-antibiotic resistance transposon made up of three distinct transposons, Antimicrob Agents Chemother, 2007, 51, 1827-1829.
67. Sunde, M. Class I Integron with a group II intron detected in an Escherichia coli strain from a free-range reindeer, Antimicrob Agents Chemother, 2005, 49, 2512-2514.
68. Toleman, M. A., Bennett, P. M., Walsh T. R. ISCR elements: novel gene-capturing systems of the 21st century?, Microbiol Mol Biol Rev 2006, 70, 296-316.
69. Vaudaux, P. E., Monzillo, V., Francois, P., Lew, D. P. at al. Introduction of the mec element (methicillin resistence) into Staphylococcus aureus alters in vitro functional activities of fibrinogen and fibronectin Adhesins, Antimicrob Agents Chemothe,1998, 42, 564-570.
70. Verdet, Ch., Benzerara, Y., Gautier, V., Adam, O. et al. Emergence of DHA-1-producing Klebsiella spp. in the Parisian region: genetic organization of the ampC and ampR genes originating from Morganella morganii. Antimicrob Agents Chemother, 2006, 50, 607-617.
71. Verdet, Ch., Arlet, G., Barnaud, G., Lagrange, P.H. et al. A novel integron in Salmonella enterica serovar Enteritidis, carrying, the blaDHA-1 gene and its regulator gene ampR, originated from Morganella morganii. Antimicrob Agents Chemother, 2000, 44, 222-225.
Labels
Hygiene and epidemiology Medical virology Clinical microbiology
Article was published inEpidemiology, Microbiology, Immunology

2010 Issue 2-
All articles in this issue
- Mobile Genetic Elements in the Epidemiology of Bacterial Resistance to Antibiotics
- Opportunistic Pathogen Candida glabrata and the Mechanisms of its Resistance to Antifungal Drugs
- Susceptibility of Staphylococcus aureus Biofilms to Vancomycin, Gentamicin and Rifampin
- Antibiotic Resistance and Biofilm Formation in Staphylococcus saprophyticus Strains Isolated from Urine
- Genotyping of Viral Glycoprotein B (gB) in Hematopoietic Stem Cell Transplant Recipients with Active Cytomegalovirus Infection: Analysis of the Impact of gB Genotypes on the Patients’ Outcome
- Epidemiology, Microbiology, Immunology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Antibiotic Resistance and Biofilm Formation in Staphylococcus saprophyticus Strains Isolated from Urine
- Opportunistic Pathogen Candida glabrata and the Mechanisms of its Resistance to Antifungal Drugs
- Mobile Genetic Elements in the Epidemiology of Bacterial Resistance to Antibiotics
- Susceptibility of Staphylococcus aureus Biofilms to Vancomycin, Gentamicin and Rifampin
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

