-
Medical journals
- Career
Identifikace dermatofytů pomocí MALDI-TOF hmotnostní spektrometrie
: R. Walková 1,2; H. Janouškovcová 1,2; M. Šnajdrová 1; V. Hubka 3,4; A. Čmoková 3,4; J. Hrabák 1,2
: Ústav mikrobiologie, Fakultní nemocnice Plzeň, přednosta RNDr. Karel Fajfrlík, Ph. D. 1; Laboratoř antibiotické rezistence a aplikací hmotnostní spektrometrie v mikrobiologii, Biomedicínské centrum, Lékařská fakulta v Plzni, Univerzita Karlova, Plzeň, vedoucí laboratoře Costas C. Papagiannitsis, Ph. D. 2; Katedra botaniky, Přírodovědecká fakulta, Univerzita Karlova, Praha, vedoucí katedry doc. RNDr. Yvonne Němcová, Ph. D. 3; Laboratoř genetiky a metabolismu hub, Mikrobiologický ústav, Akademie věd České republiky, v. v. i., Praha, vedoucí laboratoře Mgr. Miroslav Kolařík, Ph. D. 4
: Čes-slov Derm, 93, 2018, No. 6, p. 250-256
: Clinical and laboratory Research
Dermatofyty jsou mikroskopické vláknité houby způsobující časté infekce kůže a kožních adnex. Jejich identifikace byla do nedávné doby prováděna pouze podle morfologických charakteristik. Ta je však velmi zdlouhavá a vyžaduje značné zkušenosti. V posledních letech se v identifikaci dermatofytů začíná uplatňovat MALDI-TOF (matrix-assisted laser desorption/ionization time-of-flight) hmotnostní spektrometrie (MS). U vláknitých hub je nutné, kvůli jejich komplexní morfologii a požadavkům na kultivaci, použít odlišné postupy zpracování kultury. Tato práce se zabývá srovnáním MALDI-TOF MS v identifikaci dermatofytů v porovnání s metodou sekvenace oblasti ITS (Internal Transcribed Spacer) rDNA a konvenčními metodami. Do studie bylo zahrnuto 81 izolátů dermatofytů získaných z klinických vzorků. Výsledky MALDI-TOF MS analýzy se shodovaly s výsledky molekulárně genetické identifikace u 75 % izolátů. Metodami klasické identifikace se podařilo identifikovat pouze 47 % izolátů. MALDI-TOF MS lze tedy považovat za účinný nástroj rychlé a jednoduché identifikace dermatofytů v rutinní diagnostické laboratoři.
Klíčová slova:
MALDI Biotyper – Nannizzia – Trichophyton
ÚVOD
Dermatofyty jsou keratinofilní vláknité houby, způsobující infekce keratinizovaných tkání, tj. kůže a kožních adnex (nehtů, vlasů a chlupů). Taxonomie dermatofytů je vzhledem k jejich složitým životním cyklům, v nichž se střídají pohlavní a nepohlavní stadia, poměrně nepřehledná. Pro dermatofyty bylo v minulosti navrženo několik stovek jmen, která byla postupně synonymizována nebo zcela opuštěna. Navíc názvosloví hub bylo do nedávné doby duální, tzn. že pro pohlavní a nepohlavní stadium jednoho organismu byla používána dvě různá jména. V souvislosti se zavedením molekulárně genetických metod prošla taxonomie dermatofytů řadou změn. V současnosti je uznáváno asi 60 druhů dermatofytů náležejících do rodů Trichophyton (16 druhů), Epidermophyton (1 druh), Nannizzia (9 druhů), Microsporum (3 druhy), Lophophyton (1 druh), Arthroderma (21 druhů) a Ctenomyces (1 druh). Pro každý druh bylo vybráno jedno jméno podle pravidel priority [8].
Infekce způsobené dermatofyty patří mezi nejčastější lidské infekce s celosvětovým rozšířením. Prevalence těchto onemocnění závisí především na klimatických a socioekonomických podmínkách. Udává se, že minimálně jednu epizodu onemocnění prodělá během svého života asi 30 % obyvatelstva v ekonomicky vyspělých zemích, v rozvojových zemích až 70 % [13].
Z výše uvedeného je zřejmé, že dermatovenerologové a kliničtí mikrobiologové se ve své praxi s dermatofyty setkávají poměrně často. Práce s dermatofyty s sebou však přináší úskalí spočívající v obtížnosti jejich identifikace. Správná identifikace je přitom důležitá jak z hlediska klinického (volba účinného antimykotika k léčbě), tak zejména z hlediska epidemiologického. Znalost původce umožňuje vyhledání zdroje infekce a zavedení epidemiologických opatření bránících šíření infekce [15].
Identifikace dermatofytů se do nedávné doby zakládala pouze na hodnocení morfologických znaků, případně na omezeném spektru biochemických testů. Tento způsob identifikace může být v některých případech časově zdlouhavý a vyžaduje značné zkušenosti klinického mikrobiologa. Navíc není tento koncept aplikovatelný u atypických izolátů, se kterými se u dermatofytů lze běžně setkat. Tyto kmeny není možné podle morfologických kritérií přesně klasifikovat, a tak jsou často identifikovány pouze do rodu [14]. Nepřesnost klasické identifikace dokazují i některé studie. Například v externím hodnocení kvality, kterého se v letech 1991–2002 v Nizozemsku zúčastnilo 50 laboratoří, bylo správně identifikováno pouze 56 % z 1 171 testovaných izolátů dermatofytů [1]. Jednoznačné a spolehlivé výsledky identifikace poskytují molekulárně genetické metody, které však pro svou technickou a stále do jisté míry i ekonomickou náročnost nejsou v rutinní laboratorní praxi běžně dostupné. Jako významný pokrok v identifikaci dermatofytů (i ostatních vláknitých hub) se proto jeví možnost využití MALDI--TOF (matrix-assisted laser desorption/ionization-time of flight) hmotnostní spektrometrie (MS).
MALDI-TOF MS je poměrně nová metoda využívaná v klinické mikrobiologii k rychlé a jednoduché identifikaci mikroorganismů na základě analýzy molekulové hmotnosti dominantních a snadno ionizovatelných proteinů (především se jedná o ribosomální proteiny).
V případě bakterií a kvasinek je kultura nanesena na kovový terčík a po zaschnutí převrstvena roztokem matrice. U organismů s pevnou buněčnou stěnou lze extrakci buněčného obsahu napomoci překrytím organickou kyselinou (např. kyselina mravenčí) nebo provedením extrakce ve zkumavce. Matrice je malá molekula organické kyseliny se silnou optickou absorbancí, která musí korelovat s vlnovou délkou laseru použitou k ionizaci částic. Poté co laser hmotnostního spektrometru ozáří směs matrice se vzorkem, absorbuje matrice energii z laseru a uvolní protony, které přenese na extrahované bílkoviny. Tím dojde k šetrné desorpci, ionizaci a odpaření analyzovaných částic. Ionizované molekuly jsou elektrostaticky urychleny a proletí trubicí přístroje rychlostí, která je úměrná jejich hmotnosti a velikosti náboje. V celé soustavě hmotnostního spektrometru je vysoké vakuum. Stanovením doby letu částic pomocí detektoru na konci trubice lze přesně změřit rychlost jednotlivých iontů a z té pak vypočítat jejich molekulární hmotnost. Molekulární hmotnosti všech analyzovaných iontů vytváří hmotové spektrum, které je druhově specifické. Identifikace je provedena srovnáním hmotového spektra se spektry z referenční databáze [6].
Identifikace pomocí MALDI-TOF MS se již několik let s velkým úspěchem používá v mnoha laboratořích k rutinní identifikaci bakterií a kvasinek [19]. V posledních letech jsou vyvíjeny aplikace pro identifikaci virů [3, 4, 5]. Dále lze tuto techniku použít k detekci různých mechanismů antibiotické rezistence [12].
Aplikace MALDI-TOF MS v identifikaci vláknitých hub dlouho narážela na obtíže vyplývající z jejich složitější morfologie. Vláknité houby mají silnou buněčnou stěnu tvořenou z 80–90 % polysacharidy, zejména chitinem. Tato buněčná stěna je obtížně rozrušitelná. V prvních studiích s vláknitými houbami uskutečněných kolem roku 2000 proto získávali autoři většinou spektra sacharidová, nikoliv bílkovinná. Bylo proto nutné vyvinout metodu účinnou pro extrakci obsažených bílkovin [21]. Řada plísní navíc produkuje pigment melanin, který může inhibovat ionizaci. Dalším komplikujícím faktorem je skutečnost, že vláknité houby vytváří rozdílná spektra v závislosti na podmínkách růstu, stáří kolonie a přítomnosti či nepřítomnosti konidií. Další rozmanitost spekter pak může vzniknout v důsledku zvolené extrakční metody a použité matrice [2]. Z těchto důvodů se v identifikaci vláknitých hub pomocí MALDI-TOF MS uplatňují dva rozdílné přístupy. Buď je nutné standardizovat podmínky růstu a veškeré kroky preanalytické přípravy vzorku, aby získaná spektra jednotlivých izolátů stejného biologického druhu byla co nejvíce uniformní, nebo je třeba konstruovat referenční databázi tak, aby pro každý druh plísně bylo zahrnuto co nejvíce spekter a jejich heterogenita pokrývala většinu výše popsaných faktorů [2].
V současnosti nabízí vlastní komerční databáze vláknitých hub (zahrnujících i dermatofyty) řada renomovaných výrobců hmotnostních spektrometrů. Nejpoužívanější systémy jsou Andromas – Andromas SAS (Francie), SARAMIS/VITEK MS – bioMérieux (Francie) a Biotyper – Bruker Daltonik (Německo) [11].
Metoda použitá v této studii byla vyvinuta firmou Bruker Daltonik v roce 2012 ve spolupráci s Mykologickou laboratoří Limbach v Heidelbergu. Je založena na standardizované kultivaci plísní v tekutém médiu, která eliminuje negativní vliv rozdílných růstových podmínek, podporuje tvorbu uniformního mycelia a brání tvorbě konidií. Současná databáze vláknitých hub obsahuje spektra 14 druhů dermatofytů (tab. 1). Výstupem identifikace je tzv. identifikační index (nebo také skóre vzorku). Ten udává míru shody změřeného spektra s některým z referenčních spekter uložených v databázi. V rámci srovnávacího algoritmu je nejprve stanoven počet signálů v referenčním spektru, které se srovnávanému neznámému spektru podobají nejvíce, poté je stanoven počet signálů neznámého spektra, které se nejvíce přibližují referenčnímu spektru a na závěr je propočtena symetrie srovnávaných signálních párů. Každé z těchto tří charakteristik je přiřazena číselná hodnota od 0 (žádná shoda) do 1 (úplná shoda). Uvedené tři hodnoty jsou společně vynásobeny a výsledek je normalizován na 1 000. Hodnota vypočteného skóre vzorku je dekadickým logaritmem tohoto výsledku. Maximálně dosažitelná hodnota skóre je rovna 3 (= log 1 000). Čím je dosažená hodnota skóre vyšší, tím je vyšší pravděpodobnost, že neznámý mikroorganismus byl identifikován správně [3] (tab. 2).
1. Seznam druhů dermatofytů, jejichž spektra jsou zastoupena v databázi MALDI Biotyper Filamentou fungi library 1.0. 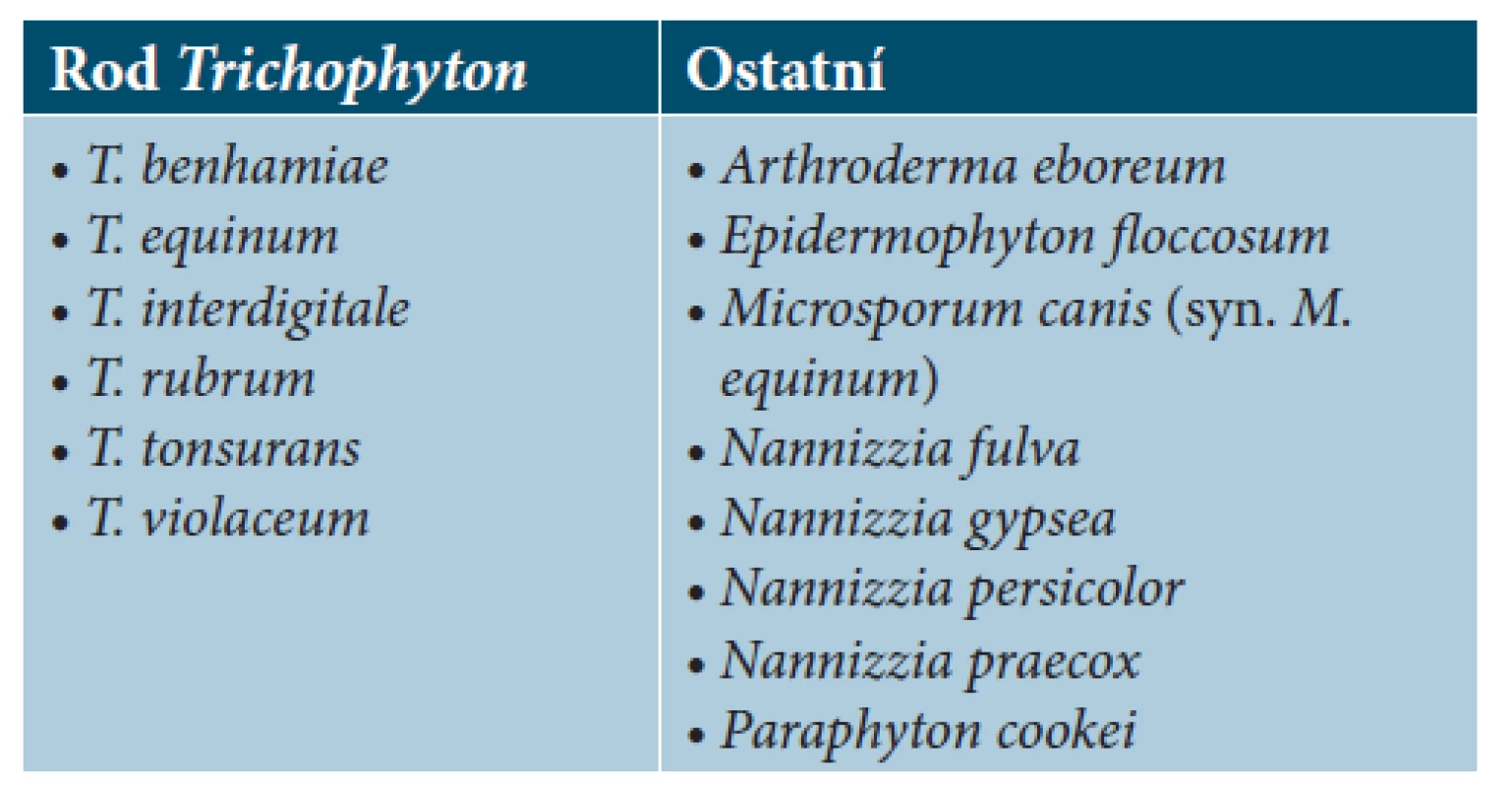
2. Skóre vzorku určující spolehlivost MALDI-TOF MS identifikace 
Cílem práce bylo srovnat MALDI-TOF MS identifikaci klinických izolátů dermatofytů s konvenční identifikační metodou a identifikací pomocí molekulárně genetických metod.
MATERIÁL A METODY
Soubor izolátů
Soubor izolátů dermatofytů byl získán kultivací kožních šupin (64 kmenů) a podnehtové drtě (17 kmenů) od pacientů ošetřených na Dermatovenerologické klinice FN Plzeň v období od 1. 5. 2013 do 30. 9. 2017. Všechny izoláty byly identifikovány konvenčními morfologickými metodami [9, 16], pomocí MALDI-TOF MS a sekvenací ITS (Internal Transcribed Spacer) oblasti rDNA [15].
Kultivační podmínky
Vzorky klinického materiálu byly kultivovány po dobu 3 týdnů při teplotě 27 °C paralelně na třech kutlivačních půdách – Sabouraudově agaru bez přídavku antibiotik (Bio–Rad), na Sabouraudově agaru s chloramfenikolem (Fagron) a na Sabouraudově agaru s chloramfenikolem a cykloheximidem (Serva Electrophoresis). Všechny půdy byly připravovány v laboratoři krátce před použitím.
K vlastní MALDI-TOF MS analýze byly použity kultury dermatofytů získané standardizovanou kultivací v tekutém Sabouraudově médiu (BBL™ Sabouraud Liquid Broth, Modified, Becton Dickinson). Stáří kultur bylo voleno tak, aby byly v médiu patrny mycelia tvaru vloček (obvykle dvou - až třídenní kultura). Vlastní postup kultivace v tekutém médiu probíhal následovně: z kultury narostlé na pevné kultivační půdě byla sterilní očkovací jehlou odebrána část mycelia a přenesena do zkumavky s tekutým médiem. Naočkovaná zkumavka byla umístěna do rotátoru, případně do třepačky a za stálého pohybu kultivována při teplotě 27 °C. Délka kultivace byla závislá na rychlosti růstu houby tak, aby bylo získáno dostatečné množství materiálu. U dermatofytů se obvykle jednalo o 3 dny. Kultivace byla ukončena tehdy, když byl v médiu viditelný nárůst houby ve formě vloček se smáčivým povrchem.
Příprava vzorku pro MALDI-TOF MS analýzu
Po vyjmutí z třepačky byla zkumavka s narostlou kulturou (postup kultivace viz výše) ponechána cca 10 minut odstát, aby vločky sedimentovaly na dno zkumavky. Následně bylo 1,5 ml sedimentu odsáto do čisté zkumavky a centrifugováno 2 minuty při 14 000 otáčkách/min. Po centrifugaci byl supernatant odstraněn a k peletě přidán 1 ml sterilní destilované vody. Směs byla promíchána krátkým vortexováním a poté centrifugována po dobu 2 minut při 14 000 otáčkách/min. Promytí, vortexování a centrifugace bylo následně zopakováno a supernatantant odstraněn. K peletě bylo přidáno 300 μl deionizované vody (Sigma-Aldrich) a 900 μl etanolu (Sigma-Aldrich), směs byla vortexována a centrifugována, supernatant odstraněn. Peleta byla následně vysušena při pokojové teplotě s cílem odstranit zbytkový ethanol. Sušení probíhalo při 37 °C po dobu cca 60–90 min. Následně bylo k peletě přidáno 25–50 μl (podle velikosti pelety) 70% kyseliny mravenčí (Sigma-Aldrich) a stejné množství acetonitrilu (Sigma-Aldrich), směs byla vortexována a centrifugována. 1 μl supernatantu byl nanesen na MALDI terčík (Microscout MSP 96 target, Bruker Daltonik) a po zaschnutí převrstven 1 μl matrice (kyselina skořicová [4-cyano-4-hydroxy-cinnamic acid, HCCA] [Bruker Daltonik] rozpuštěná v roztoku deionizované vody, acetonitrilu a kyseliny trifluoroctové [Sigma-Aldrich]). Po zaschnutí bylo provedeno vlastní měření.
MALDI-TOF MS analýza
Měření vzorků probíhalo na hmotnostním spektrometru MicroFlex LT (Bruker Daltonik) s iontovým zdrojem a analyzátorem doby letu ve vertikálním uspořádání s využitím automatického nastavení. Získaná hmotová spektra byla analyzována pomocí softwaru MALDI Biotyper 3.0 (Bruker Daltonik) a srovnána s referenční databází spekter Filamentous fungi library 1.0 (BrukerDaltonik).
VÝSLEDKY
Molekulárně genetickými metodami (sekvenace ITS rDNA) bylo v našem souboru identifikováno 31 izolátů druhu Trichophyton benhamiae, 17 izolátů druhu T. interdigitale, 13 izolátů druhu Microsporum canis, 11 izolátů druhu T. rubrum, 4 izoláty druhu T. tonsurans, 3 izoláty druhu Nannizzia persicolor, 1 izolát druhu N. gypsea a 1 izolát druhu T. verrucosum. Trichophyton rubrum, který je nejčastějším dermatofytem nalézaným v klinickém materiálu od lidí, má v našem souboru poměrně nízké zastoupení z toho důvodu, že jeho morfologická identifikace obvykle nečiní potíže a většina izolátů tohoto druhu nebyla identifikována pomocí MALDI-TOF MS.
Výsledky identifikace získané metodou MALDI-TOF MS se shodovaly s výsledky identifikace pomocí molekulárně genetických metod u 61 izolátů (75,3 %). U zbylých 20 izolátů (24,7 %) byla druhová identifikace metodou MALDI-TOF MS chybná.
Spolehlivost každé identifikace získané MALDI-TOF MS analýzou byla orientačně posouzena podle identifikačního indexu. V našem případě bylo u 35 izolátů (43,2 %) dosaženo identifikačního skóre nad 2,00 predikujícího vysokou pravděpodobnost správné druhové identifikace. Podle srovnání s výsledky molekulárně-genetických metod bylo 30 z těchto izolátů (85,7 %) identifikováno správně. Všechny chybné identifikace (5 izolátů) s indexem vyšším než 2,00 byly způsobeny vzájemnou záměnou druhů T. interdigitale a T. tonsurans. Jedná se fylogeneticky příbuzné druhy, které jsou řazeny do společného komplexu Arthroderma vanbreuseghemii [15] a podle našich výsledků je nelze pomocí MALDI-TOF MS s využitím systému Biotyper (Bruker Daltonik) spolehlivě odlišit. Třicet čtyři izolátů našeho souboru (42,0 %) bylo metodu MALDI-TOF MS identifikováno se skóre v rozmezí 1,70–1,99. Tyto hodnoty identifikačního skóre udávají vysokou pravděpodobnost správné identifikace již jen na rodové úrovni, identifikace druhu je nejistá. Přesto bylo správně určeno 28 izolátů (82,4 %). Dva izoláty T. interdigitale byly opět chybně určeny jako T. tonsurans a ve 4 případech došlo k záměně druhů T. interdigitale a T. benhamiae. S nižším skóre než 1,69 bylo analyzováno 12 izolátů (14,8 %). V této oblasti identifikačního skóre byly správně určeny pouze 3 izoláty (25,0 %). Identifikace s indexem nižším než 1,69 je tedy nespolehlivá a musí být opakována nebo podpořena výsledkem jiné metody identifikace.
Úspěšnost identifikace jednotlivých druhů dermatofytů MALDI-TOF MS je znázorněna v tabulce 3. Nejvíce byl v souboru zastoupen druh T. benhamiae (pouze žlutý fenotyp), následovaný druhy T. interdigitale, M. canis a T. rubrum. Ostatní druhy byly zastoupeny v malých počtech. Z tohoto důvodu je výpočet přesnosti obtížně hodnotitelný.
3. Srovnání výsledků molekulárně genetické identifikace s výsledky MALDI-TOF MS identifikace podle jednotlivých druhů dermatofytů 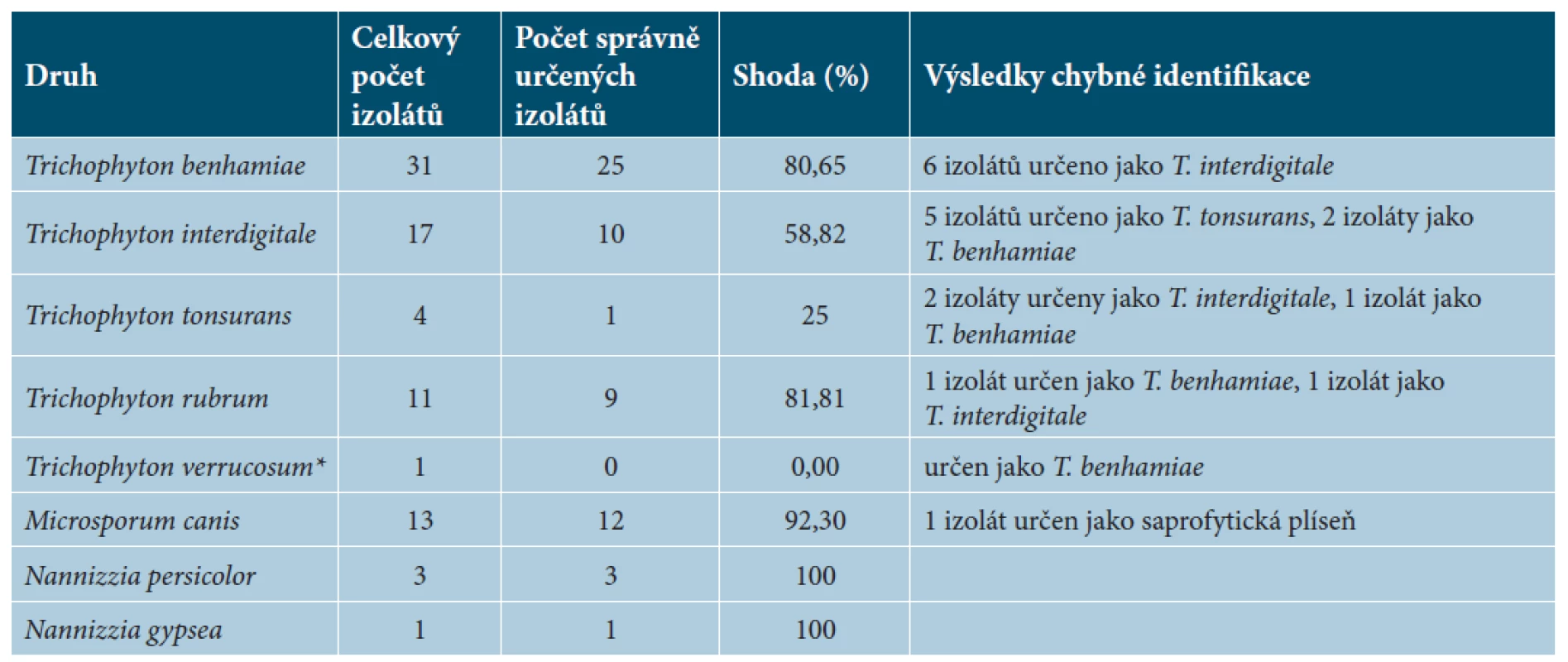
*Jedná se o druh, který není zastoupen v databázi. Z testovaného souboru 81 izolátů se klasickou metodou identifikace nepodařilo druhové určení u 34 izolátů. Ty byly identifikovány pouze do rodu jako Trichophyton spp. (33 izolátů) a Nannizzia spp. (1 izolát). Ze zbylých 47 izolátů, které byly klasickou metodou identifikovány do druhu, byla tato identifikace následně potvrzena molekulárně genetickými metodami v 38 případech, 9 izolátů bylo identifikováno chybně. Celková shoda klasické metody s molekulárně genetickými metodami na úrovni druhové identifikace tak v našem případě činila 46,9 %.
DISKUSE
Celkovým srovnáním výsledků MALDI-TOF MS identifikace s výsledky molekulárně genetických metod jsme prokázali shodu v druhové identifikaci u 75,3 % izolátů. O spolehlivosti MALDI-TOF MS identifikace značně vypovídá identifikační skóre vzorku, které vyjadřuje míru shody změřeného neznámého spektra s některým ze spekter referenční databáze. Důvody nízkého identifikačního skóre, vyjadřujícího malou shodu změřeného spektra s referenční databází, mohou spočívat na straně vzorku i na straně referenční databáze spekter. Nekvalitní hmotové spektrum vzorku může být způsobeno stářím testované kultury, kultivačními podmínkami, chybným krokem během preanalytické přípravy vzorku (chemická extrakce) nebo nanesením nehomogenní vrstvy materiálu na MALDI terčík. Zásadním faktorem ovlivňujícím kvalitu a úspěšnost MALDI-TOF MS identifikace je počet druhů dermatofytů zastoupených v referenční databázi. Námi používaná databáze Filamentous fungi library 1.0 (Bruker Daltonik) obsahuje spektra 14 druhů dermatofytů (viz tabulka 1). V epidemiologické studii mapující spektrum původců dermatofytóz na území ČR (15) bylo zachyceno 5 druhů dermatofytů, které v této databázi nemají zastoupení. Jedná se o druhy T. erinacei, T. verrucosum, Arthroderma onychocola, Nannizzia aenigmatica a A. quadrifidum. V našem souboru byl přítomen 1 izolát druhu T. verrucosum, který byl určen metodou MALDI-TOF MS jako T. benhamiae s velmi nízkým identifikačním skóre 1,67. Dalším limitujícím faktorem může být i omezené množství referenčních spekter pro jednotlivé druhy dermatofytů. Jak bylo popsáno v úvodu různé izoláty stejného biologického druhu, vytváří velice různorodá spektra v závislosti na množství různých faktorů. I přes standardizovanou metodu kultivace a přípravy vzorku není možné stoprocentně eliminovat drobné odchylky a zajistit, aby hmotová spektra izolátů stejného druhu byla vždy zcela uniformní. Úspěšnost identifikace pomocí MALDI-TOF MS tak výrazně závisí na počtu a heterogenitě zastoupených referenčních spekter pro jednotlivé druhy dermatofytů.
Aplikace MALDI-TOF MS v identifikaci vláknitých hub je oproti použití u bakterií a kvasinek poměrně nová metoda, která se stále ještě rozvíjí. První novodobou studii týkající se identifikace dermatofytů pomocí MALDI-TOF MS publikovali v roce 2008 Erhard et al. [10]. Studie byla vypracována na omezeném počtu 20 izolátů 5 druhů dermatofytů za použití systému SARAMIS (bioMérieux). Vzorky byly analyzovány přímou metodou bez chemické extrakce s využitím DHB (2,5-Dihydroxy benzoic acid) matrice. Autoři dosáhli úspěšné druhové identifikace u všech testovaných izolátů. Ve studii Theel et al. [22] z roku 2011 probíhalo měření na hmotnostním spektrometru MicroFlex LT (Bruker Daltonik) a ke srovnání spekter byla použita základní databáze MALDI Biotyper library 3.0 v kombinaci s doplněnou databází obsahující 20 přídatných spekter získaných z klinických izolátů dermatofytů a identifikovaných sekvenací oblasti 28S rDNA. Kultivace vzorků probíhala na pevných kultivačních půdách. Před vlastní analýzou byly vzorky podrobeny chemické extrakci za použití etanolu, kyseliny mravenčí a acetonitrilu jako v našem případě. Bylo analyzováno 171 izolátů náležejících do 7 druhů dermatofytů. Při použití rozšířené databáze doplněné o vlastní spektra bylo dosaženo úspěšné druhové identifikace u 60,0 % izolátů. Při použití základní databáze samotné bylo dosaženo úspěšné identifikace pouze u 20,5 % izolátů. Alshawa et al. [1] publikovali v roce 2012 studii zabývající se analýzou rozsáhlého souboru 360 izolátů 7 druhů dermatofytů pomocí systému Andromas (Andromas SAS) a vlastní databáze vytvořené ze spekter 50 referenčních kmenů náležejících do 12 druhů dermatofytů. K analýze použili třítýdenní kultury, které byly podrobeny chemické extrakci samotnou kyselinou mravenčí. Úspěšné identifikace dosáhli u 92,0 % izolátů. V roce 2013 byla publikována studie od autorů Nenoff et al. [20], v níž se autoři zabývali identifikací dermatofytů s využitím stejného identifikačního systému a stejné metody přímého stanovení jako v první studii z roku 2008 [10]. Soubor testovaných kmenů byl rozšířen na 285 izolátů náležejících do 21 druhů dermatofytů. Úspěšná identifikace proběhla v 99,0 % případů. Studie autorů De Respinis et al. [7] publikovaná v červenci 2013 byla vypracována za použití systému SARAMIS a vlastní databáze vytvořené ze spekter 108 referenčních kmenů 18 druhů dermatofytů. K analýze autoři použili třídenní kultury, které podrobili chemické extrakci kyselinou mravenčí a také proceduře odstranění lipidů za použití metanolu a chloroformu. Ze 141 izolátů 9 druhů dermatofytů bylo správně určeno 96,0 %. Ve studii L‘Ollivier et al. [17] z října 2013 se autoři se zabývali MALDI-TOF MS identifikací dermatofytů s využitím hmotnostního spektrometru UltraFlex (Bruker Daltonik). K analýze byly použity kultury dermatofytů staré 3 a 15 dnů, následná analýza neprokázala zásadní rozdíl ve výsledku identifikace podle stáří kultury. Kultivace probíhala na pevných kultivačních půdách a před analýzou byla provedena chemická extrakce etanolem, kyselinou mravenčí a acetonitrilem. Ke srovnání spekter autoři použili vlastní databázi obsahující 384 spekter získaných ze 48 izolátů 17 druhů dermatofytů. S takto vybavenou databází dosáhli úspěšné identifikace MALDI-TOF MS analýzou u 98,0 % ze 133 testovaných izolátů.
Tento soubor publikovaných studií potvrzuje, že úspěšnost MALDI-TOF MS identifikace zcela zásadně závisí na kvalitě referenční databáze. Autoři, kteří používali vlastní databáze obsahující velký počet heterogenních spekter, dosáhli bez ohledu na použitý identifikační systém úspěšné identifikace v rozmezí 92–98 %. Naproti tomu Theel et al. [22], kteří používali komerční databázi zahrnující omezený počet spekter, dosáhli úspěšné identifikace pouze v 20,5 %, při použití databáze rozšířené o vlastní spektra pak v 60,0 %. Právě tato studie se našemu provedení přibližuje nejvíce a v porovnání s ní je patrné, že použití nové databáze vláknitých hub Filamentous fungi library 1.0 značně zvyšuje úspěšnost MALDI-TOF MS identifikace (75% úspěšnost v naší studii).
ZÁVĚR
Ačkoliv úspěšnost identifikace metodou MALDI-TOF MS na našem pracovišti nedosahuje hodnot referovaných jinými autory, přesto značně převyšuje úspěšnost klasické metody identifikace. Kromě vyšší úspěšnosti a spolehlivosti přináší MALDI-TOF MS analýza ve srovnání s klasickou identifikací i četné další výhody. Klasická identifikace dermatofytů je levná metoda nevyžadující žádné speciální vybavení. Je však značně subjektivní a klade vysoké nároky na zkušenosti vyšetřujícího mikrobiologa. Její úspěšnost je závislá na tvorbě konidií, případně jiných charakteristických mikroskopických struktur, což trvá dlouhou dobu (obvykle týdny) a v řadě případů nedojde k vytvoření charakteristických identifikačních znaků vůbec. Nejspolehlivější výsledky identifikace poskytují molekulárně genetické metody, které však pro svou ekonomickou nákladnost a náročnost provedení nejsou ve většině běžných klinických laboratoří dostupné. Metoda MALDI-TOF MS vyžaduje vysokou vstupní investici, avšak další náklady na jednotlivá vyšetření jsou minimální. K identifikaci touto metodou je zapotřebí pouze malé množství materiálu. K identifikaci obvykle postačuje třídenní kultura, takže časová úspora proti klasické identifikaci činí v některých případech i týdny. Vlastní příprava vzorku k vyšetření je u vláknitých hub sice složitější než přímá analýza bakterií a kvasinek, přesto je stále dostatečně jednoduchá na to, aby nezatěžovala běžný chod laboratoře. Souhrnně řečeno MALDI-TOF MS je metoda levná, rychlá, jednoduchá a spolehlivá. Mezi omezenými možnostmi identifikace dermatofytů představuje významný přínos, stejně jako je tomu již řadu let v případě bakterií a kvasinek.
Poděkování
Práce byla podpořena grantem Agentury zdravotnického výzkumu České republiky (AZV 17-31269A), Národním programem udržitelnosti č. LO1503 a Grantovou agenturou Univerzity Karlovy (GAUK 600217).
Vít Hubka děkuje Československé mikroskopické společnosti za poskytnutou podporu (ČSMS stipendium 2016).
Do redakce došlo dne 5. 3. 2018.
Adresa pro korespondenci:
doc. Ing. Jaroslav Hrabák, Ph.D.
Biomedicínské centrum, LF Plzeň
Alej Svobody 76
323 00 Plzeň
email: Jaroslav.Hrabak@lfp.cuni.cz
Sources
1. ALSHAWA, K., BERETTI, JL., LACROIX, C., FEUILHADE, M. et al. Successful identification of clinical dermatophyte and Neoscytalidium species by Matrix-assisted laser desorption ionization–time of flight mass spectrometry. J Clin Microbiol, 2012, 50 (7), p. 2277–2281.
2. BADER, O. MALDI-TOF-MS-based species identification and typing approaches in medical mycology. Proteomics, 2013, 13, p. 788–799.
3. CALDERARO, A., ARCANGELETTI, M. C., RODIGHIERO, I., BUTTRINI, M. et al. Matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) mass spectrometry applied to virus identification. Sci Rep, 2014, 4, p. 6803.
4. CALDERARO, A., ARCANGELETTI, M. C., RODIGHIERO, I., BUTTRINI, M. et al. Identification of different respiratory viruses, after a cell culture step, by matrix assisted laser desorption/ionization time of flight mass spectrometry (MALDI-TOF MS). Sci Rep, 2016, 6, p. 36082.
5. COBO, F. Application of MALDI-TOF mass spectrometry in cinical virology: a review. Open Virol J, 2013, 7, p. 84–90.
6. CROXATTO, A., PROD´HOM, G., GREUB, G. Application of MALDI-TOF mass spectrometry in clinical diagnostic microbiology. FEMS Microbiol Rev, 2012, 36, p. 380–407.
7. DE RESPINIS, S., TONOLLA, M., PRANGHOFER, S., PETRINI, L. et al. Identification of dermatophyte by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Med Mycol, 2013, 51, p. 514–521.
8. DE HOOG, G. S., DUKIK, K., MONOD, M., PACKEU, A. et al. Toward a novel Multilocus Phylogenetic Taxonomy for the Dermatophytes. Mycopathologia, 2017, 182, p. 5–31.
9. DE HOOG, G. S., GUARRO, J., GENÉ, J., FIGUERAS, M. J. Atlas of clinical fungi. 2nd edition. Centraalbureau voor Schimmelcultures, 2000. ISBN 90-70351-43-9.
10. ERHARD, M., HIPLER, U. C., BURMESTER, A., BRAKHAGE, A. A., WÖSTEMEYER, J. Identification of dermatophyte species causing onychomycosis and tinea pedis by MALDI-TOF mass spectrometry. Exp Dermatol, 2008, 17, p. 356–361.
11. GRÄSER, Y. Species identification of dermatophytes by MALDI-TOF MS. Curr Fungal Infect Rep, 2014, 8, p. 193–197.
12. HRABÁK, J., CHUDÁČKOVÁ, E., WALKOVÁ, R. Matrix-assisted laser desorption ionization–time of flight (MALDI-TOF) mass spectrometry for detection of antibiotic resistance mechanisms: from research to routine diagnosis. Clin Microbiol Rev, 2013, 26(1), p. 103–114.
13. HUBKA, V., MALLÁTOVÁ, N. Vláknité houby z povrchu lidského těla. Živa, 2012, 3, p. 107–110.
14. HUBKA, V., ČMOKOVÁ, A., SKOŘEPOVÁ, M., MALLÁTOVÁ, N. et al. Současný vývoj v taxonomii dermatofytů a doporučení pro pojmenovávání klinicky významných druhů. Čes-slov Dermatol, 2014, 89(4), p.151–165.
15. HUBKA, V., VĚTROVSKÝ, T., DOBIÁŠOVÁ, S., SKOŘEPOVÁ, M. et al. Molekulární epidemiologie dermatofytóz v České republice – výsledky dvouleté studie. Čes–slov Dermatol, 2014, 89(4), p. 167–174.
16. LARONE, D. H. Medically important fungi: a guide to identification. 3rd edition. Washington D.C.: American Society for Microbiology, 1995. ISBN 1-55581-091-8.
17. L‘OLLIVIER, C., CASSAGNE, C., NORMAND, A. C., BOUCHARA, JP. et al. A MALDI-TOF MS procedure for clinical dermatophyte species identification in the routine laboratory. Med Mycol, 2013, 51, p. 713–720.
18. MALDI Biotyper 3.0 uživatelská příručka. 2. revize. Bruker Daltonik GmbH. 2011.
19. MURRAY, P. R. What is new in clinical microbiology – Microbial identification by MALDI-TOF mass spectrometry. J Mol Diag, 2012, 14(5), p. 419–423.
20. NENOFF, P., EEHARD, M., SIMON, J. C., MUYLOWA, G. K. et al. MALDI-TOF mass spectrometry – a rapid method for the identification of dermatophyte species. Med Mycol, 2013, 51, p. 17–24.
21. SANTOS, C., PATERSON, R. R. M., VENANCIO, A., LIMA, N. Filamentous fungal characterization by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. J Appl Microbiol, 2010, 108(2), p. 375–385.
22. THEEL, E. S., HALL, L., MANDREKAR, J., WENGENACK, N. L. Dermatophyte identification using Matrix-assisted laser desorption ionization–time
of flight mass spectrometry. J Clin Microbiol, 2011, 49(12), p. 4067–4071.
Labels
Dermatology & STDs Paediatric dermatology & STDs
Article was published inCzech-Slovak Dermatology

2018 Issue 6-
All articles in this issue
- Effective Therapeutical Modulation of the Inflammation in Patients with Psoriasis Using Guselkumab Targeting the Specific Subunit p19 IL-23 of the Regulatory Axis of IL-23/Th17
- Identification of Dermatophytes Using MALDI-TOF Mass Spectrometry
- Use of PCR-HRMA for Direct Detection and Identification of Dermatophytosis Agents from Clinical Samples
- Therapy of Onychomycosis Using the Non-thermal Plasma
- Zoonotic Dermatophytoses: Clinical Manifestation, Diagnosis, Etiology, Treatment, Epidemiological Situation in the Czech Republic
- Five Cases of Dermatophytosis in Man Caused by Zoophilic Species Trichophyton erinacei Transmitted from Hedgehogs
- Czech-Slovak Dermatology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Zoonotic Dermatophytoses: Clinical Manifestation, Diagnosis, Etiology, Treatment, Epidemiological Situation in the Czech Republic
- Five Cases of Dermatophytosis in Man Caused by Zoophilic Species Trichophyton erinacei Transmitted from Hedgehogs
- Identification of Dermatophytes Using MALDI-TOF Mass Spectrometry
- Therapy of Onychomycosis Using the Non-thermal Plasma
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

