-
Medical journals
- Career
Diferenciální exprese microRNA u systémových autoimunitních onemocnění
Authors: M. Škoda 1; P. Novota 1; L. Pleštilová 2; M. Remáková 1; M. Klein 2; J. Vencovský 2
Authors‘ workplace: Laboratoř molekulární biologie a imunogenetiky, 2Vědeckovýzkumné centrum, Revmatologický ústav Praha 1
Published in: Čes. Revmatol., 19, 2011, No. 4, p. 170-189.
Category: Rewiev
Overview
MicroRNA (miRNA) jsou malé, nekódující, evolučně konzervované RNA molekuly, posttranskripčně regulující genovou expresi. Epigenetickým mechanismem RNA interference (RNAi) ovlivňují stabilitu a translační účinnost cílových mRNA. Jsou nalézány u většiny organismů, tvoří 1-2% eukaryotických genomů a řídí expresi přibližně 1/2 protein kódujících genů. Podílejí se na regulaci klíčových biologických procesů (buněčný růst, diferenciace, proliferace, apoptóza). Recentní nálezy zdůrazňují zásadní význam miRNA ve vývoji, homeostáze a funkci vrozené i adaptivní imunity. Aberantní expresní vzory jsou součástí patogeneze široké škály chorob, včetně poruch autoimunity. Článek shrnuje diferenciální microRNAexpresi u revmatoidní artritidy, systémového lupusu erythematodes, Sjögrenova syndromu, systémové sklerodermie a idiopatických zánětlivých myopatií a podává charakteristiku microRNA jako biomarkerů a potenciálních terapeutických cílů.
Klíčová slova:
microRNA, systémová revmatická onemocnění, RA, SLE, Sjögrenův syndrom, systémová sklerodermie, IIM, epigenetikaÚvod
Etiologie systémových autoimunitních chorob zůstává neobjasněna. Příčiny vzniku jsou multifaktoriální, včetně polygenní predispozice. Podstatný podíl genetického rizika připadá na oblast HLA systému, asociaci s autoimunitními procesy však vykazují i polymorfismy dalších genů ležících mimo tento region (např. PTPN 22). Celogenomové asociační studie navrhly i řadu vazeb platných pouze pro menšinové skupiny pacientů.
Snahy o identifikaci asociovaných genů doplňují výzkumy dějů řídících genovou expresi. Zvláštní třídu regulačních pochodů představují epigenetické modifikace. Epigenetické modifikace jsou dědičné změny genové exprese (fenotypu), které nepostihují primární strukturu DNA (nukleotidová sekvence) (1). Podléhají vlivu prostředí (životní styl, věk, pohlaví) a mohou být reverzibilní (2). Jejich charakter je dynamický a potenciálně výlučný pro každou buňku organismu (3). K typickým epigenetickým procesům se značným dopadem na DNA funkci patří metylace DNA a posttranslační modifikace histonů (acetylace, metylace, fosforylace, ubiquitinace a sumoilace). Specifický epigenetický jev představuje RNA interference (RNAi). Nedokonale prozkoumáno je působení iontového prostředí na vlastnosti chromatinu, či vliv změn v terciální struktuře DNA (supervinutí) (4). Epigenetické modifikace vytvářejí komplexní aparát zapojený v poruchách imunitní tolerance a udržování autoimunity. Novou vrstvu řízení genové exprese s podílem v etiopatogenezi revmatických onemocnění reprezentují nekódující molekuly RNA (ncRNA). NcRNA nepodstupují překlad do proteinu, mnohé z nich jsou funkční a účastní se zpracování a regulace jiných RNA typů (mRNA, tRNA, rRNA) (5). Charakteristickou skupinu regulačních ncRNA tvoří krátké RNA molekuly (siRNA, miRNA, piRNA), které účinkují epigenetickým mechanismem RNA interference (6). RNAi je způsob sekvenčně specifického, posttranskripčního ovlivnění genové exprese. Zvláštní úseky krátkých ncRNA hybridizují ke kódující či promotorové oblasti mRNA a obvykle tlumí její translaci. Tři hlavní třídy krátkých ncRNA: siRNA (small interfering), miRNA (microRNA) a piRNA (piwi interacting) se liší strukturou, biogenezí a způsobem cílového účinku a kromě RNAi jsou zapojeny do dalších dějů (chromatinem zprostředkovaný genový silencing, přestavba DNA) (5). Vedle uvedených druhů existují ještě jiné ncRNA s epigenetickou úlohou (např. krátké XiRNA či dlouhé ncRNA, větší než 200nt) (7). Zmíněné ncRNA vytvářejí složitou infrastrukturu, která časově i prostorově reguluje genovou expresi.
Biogeneze a funkce molekul miRNA
Nejlépe prozkoumanou třídu regulačních ncRNA představují microRNA. MiRNA je rodina cca 22nt dlouhých, nekódujících, evolučně konzervovaných RNA molekul, které formou RNAi upravují stabilitu a translační účinnost cílových mRNA. Tvoří přibližně 1–2 % eukaryotických genomů a regulují nejméně 50% protein kódujících genů (8). Biogeneze je složitá a probíhá ve vícestupňovém procesu. V jádře jsou miRNA geny přepsány do primárního miRNA transkriptu, sestřiženy a multiproteinovým komplexem obsahujícím RNázu III Drosha štěpeny na tzv. prekurzorovou miRNA vlásenku. Ta je transportována do cytoplasmy, kde podstupuje štěpení endonukleázou DICER (RNáza III.). DICER odstraní vlásenkovou smyčku a ve spolupráci s proteiny vážícími RNA generuje nestabilní, asymetrický miRNA/miRNA* duplex. Vlákno s méně stabilním 5´koncem dozrává a je začleněno do ribonukleoproteinového komplexu RISC. RISC hraje zásadní roli v inhibici genové exprese. Většina miRNA interaguje s komplementární sekvencí v 3´UTR cílových mRNA. Míra komplementarity určí mechanismus posttranskripční regulace. Dokonalé párování ústí v přímou degradaci mRNA, neúplná komplementarita buď ovlivní stabilitu, nebo blokuje překlad cílového transkriptu (9). Kromě typické vazby na 3´UTR se miRNA mohou zaměřit i na 5´UTR a kódující mRNA sekvence (10). Popsány jsou i interakce s DNA a proteiny (11).
Regulace genové exprese miRNA molekulami je polyvalentní a redundantní: každá miRNA může interagovat s množstvím mRNA cílů, jedna mRNA je schopna vázat různé miRNA. Inhibiční účinek určitého miRNA typu na syntézu cílového proteinu je mírný až střední (12, 13). Expresní profily bývají tkáňově a buněčně specifické a podléhají přísné kontrole (14). Funkční studie ukazují, že některé miRNA využívají alternativní metody tlumení. (např. urychlenou deadenylaci polyA konce mRNA) (15). Jiné práce shledaly, že živočišné miRNA mohou za specifických podmínek zesilovat translaci (16). MiRNA se podílejí i na složitých mechanismech, které zahrnují zpětnou vazbu, samoregulaci a v některých případech i metylační a acetylační procesy (17). Rostoucí množství důkazů navíc potvrzuje, že geny pro miRNA jsou cílem výše zmíněných epigenetických modifikací (metylace, acetylace) (17).
MicroRNA ovlivňují řadu biologických dějů (embryogeneze, organogeneze, proliferace, apoptóza, stres). Přispívají k regulaci kmenových buněk (18–20), kožní morfogenezi (21), svalové diferenciaci (22), ontogenezi slinivky (23), srdce (24, 25) a nervové soustavy (26). Aberantní expresní vzory jsou asociovány s patogenezí malignit (27, 28), srdečních (29), neurodegenerativních (30) a neuropsychiatrických (31) chorob, autoimunitních zánětlivých onemocnění a infekcí (32, 33). Současné studie zdůrazňují zásadní význam miRNA ve vývoji, homeostáze a funkci vrozené i adaptivní imunity. U systémových autoimunitních onemocnění hrají klíčovou úlohu v regulaci hlavních, v patogenezi zapojených molekul (TNFα), centrálních signálních drah (IFN I. typu) a kritických imunoregulačních buněk (regulační T buňky) (34). Obecná role microRNA v imunitě a autoimunitních dějích byla shrnuta v předchozím přehledovém článku (35).
MicroRNA jako biomarkery a potenciální léčebné cíle
Značná očekávání vzbuzuje studium miRNA jako biomarkerů. Expresní profily odrážejí patofyziologické procesy, jsou patognomické a specifické pro jednotlivá stadia nemoci. Diagnostická či prognostická hodnota byla prokázána u řady neoplasií a několika nemaligních procesů (36–39). Onkologie dnes disponuje prvními, na microRNA diagnostice založenými testy (37, 38). Vzhledem k předběžným závěrům výzkumů autoimunitních chorob lze miRNA považovat za slibné kandidátní ukazatele diagnózy, prognózy, aktivity a závažnosti těchto onemocnění. MicroRNA jsou reprodukovatelně měřitelné a v porovnání s mRNA mají několik předností. Vzhledem k malé velikosti a vazbě s proteinovými komplexy vykazují dlouhou in vivo životnost, stabilitu a rezistenci k degradaci nukleázami. Detekci lze provádět z různých zdrojů: krve a krevních složek (sérum, plazma), ostatních biologických tekutin, tkání či formalinem fixovaných histologických řezů.
Specifickou skupinu miRNA biomarkerů představují cirkulující extracelulární microRNA. V plazmě, séru a ostatních nebuněčných tekutinách jsou miRNA vázány v mikropartikulích (exosomy, mikrovesikuly) tj. v intercelulárních mediátorech biologické informace odvozených z pevných tkání, leukocytů a trombocytů. Tato skutečnost významně zvyšuje jejich klinický potenciál (sliny, moč – neinvazivita vyšetření). Recentní práce navíc ukázaly, že v krevní plazmě je podstatná část miRNA molekul spojena i s jinými typy nosičů: proteiny typu Argonaut 2 či high density lipoproteiny (HDL) (40, 41). Potenciální funkce extracelulární miRNA je předmětem aktivního výzkumu, předpokládá se, že může sloužit jako prostředek mezibuněčné komunikace (42, 43) a k regulaci genové exprese recipientních buněk (40).
V případě funkční vazby s chorobou lze micro RNA využít terapeuticky. Léčba je založena na několika přístupech. Nadměrně exprimované miRNA mohou být přímo inhibovány protismyslnými, specificky upravenými anti-miRNA oligonukleotidy. Mezi tři v současnosti využívané třídy těchto blokátorů patří antagomiry, LNA (locked nucleic acid) a MOE (2´-O-metoxyethyl phosphorothioate). Antagomiry jsou cholesterolem konjugované syntetické nukleotidy používané např. k výzkumu léčby rakoviny (blokace onkomirů) (44). První úspěšný experiment provedli v r. 2004 Hautvágner et al., kteří vpravovali 2´-O - metyl nukleotid komplementární k let–7 do larvy háďátka (C. elegans) (45). Iniciální in vivo aplikaci u savců popsali Krützfeldt et al. (46). Opakované podávání intravenosní injekce antagomirů proti miR-16, miR-122, miR-192 a miR-194 dlouhodobě snížilo hladiny těchto microRNA u myší. V případě miR-16 byl efekt zaznamenán v mnoha tkáních s výjimkou mozku, kde mu bránila hematoencefalická bariéra. Překvapivý úspěch přinesla aplikace LNA nehumánním primátům (47). Nitrožilní podání LNA anti-miR-122 dlouhodobě umlčelo expresi zralé miR-122. Pokus byl bezpečný, autoři nezaznamenali toxicitu ani histopatologické změny. Klinické využití anti-miR terapie zkoumali též Landford et al. (48). MiR-122 je játry exprimovaný typ nezbytný pro replikaci viru hepatitidy C. Léčba chronicky infikovaných šimpanzů pomocí LNA anti-miR-122 potlačila virémii žloutenky C a vytvořila rezistenci k onemocnění.
Nepřímé inhibiční metody snižují množství microRNA zásahem do biogeneze. Tak mohou tetracyklinem indukovatelné shRNA (short hairpin) potlačit DICER či Droshu, klíčové enzymy v tvorbě microRNA. Tento způsob je neselektivní, ovlivňuje všechny miRNA typy a musí být přísně kontrolován (44).
Hladiny hypoexprimovaných microRNA lze zvyšovat podáním syntetických RNA duplexů, které jsou rozpoznány a zpracovány RISC komplexem. Strategie však vyžaduje odzkoušení in vivo (44). K obnovení microRNA exprese u buněk se využívají i krátké interferující vlásenky řízené promotory typu RNA Pol III. Genová dávka musí být kontrolována, protože trvale vysoká exprese shRNA v játrech myši byla pro organismus fatální (49).
MiRNA a viry
Soubor microRNA lze nahlížet jako archaický imunitní systém, chránící organismy před virovou infekcí. Leccelier et al. prokázali, že miR-32 snižuje expresi nejméně pěti mRNA PFV1 (primate foamy virus) viru a zpomaluje tak jeho množení (50). Jiná studie shledala, že stimulace hepatocytů IFNgama aktivuje osm microRNA, které potlačují replikaci viru lidské hepatitidy C (51).
Některé viry však kódují vlastní microRNA inhibující genovou expresi hostitele. Geneticky výhodným mechanismem tak zvyšují replikaci a dobu přežití. Příkladem jsou EBV (virus Epsteina a Baarové), HCMV (lidský cytomegalovirus), HSV-1 (herpes simplex virus), HIV-1 (virus lidské imunodeficience) či KSHV (s Kaposiho sarkomem asociovaný herpes virus), z nichž EBV a HCMV jsou spojované s patogenezí revmatických onemocnění. Např. miRUL-122 lidského cytomegaloviru snižuje expresi MHC povrchových receptorů a zabraňuje buňkám v prezentaci svých antigenů (vazba na MICB (MHC class I polypeptide-related sequence B) sekvenci, nezbytnou pro identifikaci infekce NK buňkami) (52). MiR-K12-11 kódovaná KSHV je ortologní s buněčnou miR-155 a zaměřením stejných cílů může přispívat k zvýšené incidenci B buněčných lymfomů u pacientů trpících touto infekcí (53). Je proto pravděpodobné, že se některé virové microRNA podílejí i na patogenezi systémových autoimunitních onemocnění. EB viróza indukovala expresi miR-146a a miR-155 způsobem závislým na NFκB (54–56). Přestože množství miR-146a v PBMC SLE pacientů bylo sníženo (57,58), hladiny miR-146 a miR-155 ve vzorcích RA pacientů (CD4+T lymfocyty, PBMC, RASF) byly elevované (59–63). Uvedené údaje naznačují možnou úlohu těchto dvou miRNA při vzniku onemocnění zprostředkovaných EBV.
Diferenciální miRNA exprese u systémových autoimunitních onemocnění
Deregulace exprese miRNA ovlivňuje vývoj a udržování autoimunity. Aberantní expresní vzory byly zjištěny u revmatoidní artritidy, systémového lupusu erytematodes, Sjögrenova syndromu, systémové sklerodermie a idiopatických zánětlivých myopatií. Získaná data shrnuje tabulka 1. Dosavadní nálezy osvětlily funkci některých microRNA a naznačily možnost jejich využití jako diagnostických markerů a léčebných cílů. Přesná patogenetická úloha u revmatických onemocnění však zůstává nejasná. K tomuto faktu přispívá i značná diverzita dosud provedených studií.
Table 1. miRNA u systémových autoimunitních onemocnění. 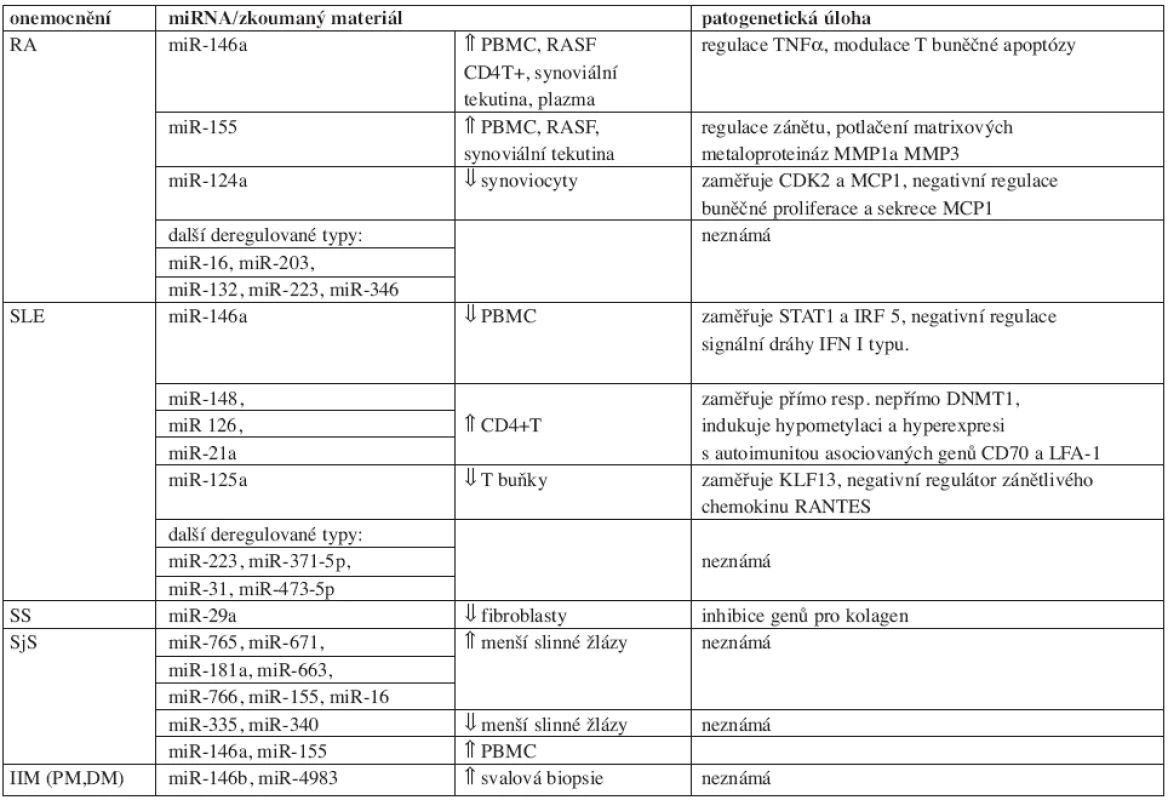
Revmatoidní artritida (RA)
Alteraci miRNA exprese u pacientů s RA popisuje 15 prací (59–73). Analýzy synoviální tkáně, synoviálních fibroblastů (RASF), PBMC, plazmy, séra, mononukleárních buněk synoviální tekutiny, naivních CD4+ T lymfocytů a T buněk produkujících IL-17 identifikovaly množství deregulovaných miRNA typů. Přehled podává tabulka 2. Některé výzkumy odhalily zapojení miRNA v patogenezi choroby, žádná z abnormálně exprimovaných miRNA však není pro nemoc specifická. MiR-124a reguluje proliferaci RASF (66, 67), miR-155 potlačuje matrixové metaloproteinázy a inhibitor zánětu SHIP (60, 72). MiR-146a, miR-155, miR-203 a miR-346 ovlivňují hladiny prozánětlivých cytokinů (59, 62, 65, 72, 73). O úspěchu miRNA terapie u myší referují dvě studie (74, 75).
Podstatné informace o miRNA funkci, roli jako biomarkeru a terapeutickém potenciálu u RA lze shrnout následovně:
miR-15a/16-1 a miR-15b/16-2
Obecné poznámky:
MiR-15a/16-1 a miR-15b/16-2 představují vysoce konzervované klastry (76) s významnou úlohou v regulaci buněčné proliferace a apoptózy (zaměření proteinů buněčného cyklu a antiapoptotických genů) (77, 78). Četné studie popsaly úlohu v indukci programované buněčné smrti a hypoexpresi u různých typů malignit (79–82). Vazbu miR-16 na 3´UTR TNFα a její roli v procesu ARE zprostředkované degradace odhalili Jing et al. (83).
miR-15a jako terapeutikum:
Nagata et al. vpravovali dvouvláknovou miR-15a do kloubů myší s artritidou indukovanou autoprotilátkami (74). Exprese miR-15a je v synoviální tkáni myšího modelu snížena, množství antiapoptotického proteinu Bcl zvýšeno. Tento vztah podporuje teorii o zesílené rezistenci revmatoidních fibroblastů vůči apoptóze. Experiment měl zjistit, zda dojde k přijetí miR-15a do buněk, následnému potlačení Bcl-2 a navození programované buněčné smrti. Intraartikulární infiltrace způsobila hyperexpresi miR-15a, sníženou tvorbu Bcl-2 a snížení hladin kaspázy 3 u exponované skupiny. Terapeutické podání miR-15a tak indukovalo apoptózu buněk hyperplastické kloubní tkáně.
miR-16 jako biomarker:
Pauley et al. (59) shledali 1,9násobný vzrůst exprese miR-16 (p < 0,05) v RA PBMC ve srovnání s buňkami zdravých dárců. Výše hladin se vztahovala k aktivitě onemocnění.
Jejich závěry potvrdili na kohortě čínských pacientů Feng et al. Exprese miR-16 korelovala s hodnotami ESR, CRP a DAS 28, nikoli však revmatoidního faktoru (RF) (71).
Rovněž Murata et al. informují o korelaci plazmatické miR-16 s DAS 28 a počtem bolestivých kloubů (TJC). Množství miR-16 v synoviální tekutině OA a RA nemocných bylo výrazně vyšší než v plazmě. Pacienti trpící OA vykázali signifikantně nižší hodnoty miR-16 než zdraví dárci. Získaná data naznačují potenciální využití mir-16a jako biomarkeru aktivity onemocnění (63).
miR-124a
Obecné poznámky:
Přednostně exprimována v nervové tkáni (84), slinivce (85), zapojena v karcinogenezi (86, 87), kde působí, v závislosti na typu malignity, jako tumor supresorová či s metastatickým procesem asociovaná miRNA (88). Úlohu v posttranskripční represi IκBζ mRNA a v modulaci genové exprese zprostředkované NFκB zmiňují Lindelblatt et al. (89)
Funkce u RA:
Expresní profilování RA a OA synoviocytů ukázalo snížené hladiny miR-124a v RA fibroblastech podobných synoviocytům (FLS) ve srovnání OA FLS (66, 67). Nadměrná in vitro exprese miR-124a v RA FLS potlačila proliferaci a pozastavila buněčný cyklus v G1 fázi. MiR-124a zaměřuje 3’UTR CDK-2 a MCP-1 mRNA a snižuje jejich translaci (66). CDK 2 je katalytická podjednotka komplexu cyklin-dependentních kináz, jejichž činnost má zásadní význam v G1 / S přechodu. MCP-1 (Monocyte chemotactic protein) je CC chemokin, vysoce exprimovaný v synoviální tkáni RA pacientů. Patogeneticky působí podporou migrace a udržením mononukleárních fagocytů v kloubech. Vzhledem k zjištěným faktům lze MiR-124α považovat za významného regulátora proliferace RA FLS. Dalším cílem této miRNA se zdá být IαB, což svědčí o úloze v regulaci signální dráhy NFκB (89).
miR-132
Obecné poznámky:
Modulátor neuronálních a imunitních procesů. Zapojena v morfogenezi nervových buněk, deregulována u neuropatologií (Alzheimerova demence, Huntingtonova chorea, mozkové ischemie, schizofrenie) (90). Účast miR-132 v regulaci antivirové imunity popisují Lagos et al. (91). Vazbou na enzym acetylcholinesterázy posiluje miR-132 protizánětlivou cholinergní signalizaci (92).
miR-132 jako biomarker:
Zatímco RA PBMC vykazují zvýšené hladiny miR-132 (59), plazmatické koncentrace RA pacientů jsou ve srovnání se zdravými dárci sníženy (63). Diskrepanci vysvětluje Doroux-Richard odlišnou miRNA expresí v obou zdrojích: typy vyjádřené v krevních buňkách nebývají v séru a plazmě zpravidla nalézány (93).
Murata et al. měřili množství miR-132 v plazmě a synoviální tekutině pacientů s RA a OA. Plazmatická miR-132 byla detekována též u zdravých subjektů. Expresní analýza ukázala signifikantně nižší koncentrace miR-132 v synoviální tekutině oproti plazmě. Hodnoty plazmatické miR-132 byly u nemocných výrazně nižší, než u zdravých dárců. Synoviální tekutina RA pacientů měla vyšší koncentraci než punktáty OA nemocných. Vyšetření plazmy zřetelně odlišilo zdravé kontroly od skupin pacientů, vyšetření synoviální tekutiny diferencovalo RA a OA nemocné (63).
miR-146a
Obecné poznámky:
Reguluje vrozenou imunitu, inhibuje TLR zprostředkovanou NFαB signalizaci a brzdí tak zánětlivou odpověď. Taganov et al. pozorovali hyperexpresi miR-146a/b v lidských THP-1 monocytech stimulovaných lipopolysacharidem (LPS) (94). Exprese miR-146a je NFκB dependentní a omezuje nadprodukci prozánětlivých cytokinů (TNFα, IL-1β). Dva výzkumy identifikovaly TRAF6 a IRAK1, klíčové molekuly zapojené v signalizaci Toll-like a IL-1 receptorů jako cílové geny posttranskripční represe miR-146a/b (94, 95). Mir-146α potlačuje aktivitu NFκB (95), LPS indukovaný zánět (96) a ovlivňuje rozvoj tolerance k endotoxinům (97, 98). Lu et al. ukázali nezbytnost miR-146a pro udržení supresorických funkcí T regulačních lymfocytů (Treg). Myši s nedostatkem miR-146a v Treg vyvinuly poruchy imunologické tolerance ústící ve smrtelné, multiorgánové imunitní léze zprostředkované IFNγ (99). Úlohu v odpovědi na virovou infekci zkoumalo několik prací. Negativní regulaci antivirové dráhy RIG I. pomocí miR-146a popsali Hou et al. (96). EB virózou kódovaný LMP1 může indukovat expresi miR-146a skrze vazebné místo NFαB v jejím promotoru (54, 56). Alterovaná exprese miR-146a je součástí patogeneze mnoha chorob, včetně virových (54, 56), svalových (100) a onkologických onemocnění (101–106). Deregulaci vykazují též zánětlivé autoimunitní procesy: RA (59–64, 68, 70, 71), SLE (57, 58, 107), Sjögrenův syndrom (108) a psoriáza (109, 110).
Funkce u RA:
Zvýšenou expresi miR-146a v RASF (60), synoviální tkáni (61, 68), synoviální tekutině (63), PBMC (60, 68, 70, 71), CD4+T lymfocytech synovie a periferní krve (62), T buňkách produkujících IL-17 (68) a plazmě pacientů s RA (63) shledalo osm studií.
Tři z nich zjistily zvýšenou bazální expresi v RASF ve srovnání s OASF (60, 61, 68). RASF vykazují proti OASF vyšší senzitivitu k prozánětlivým stimulům: působení LPS, TNFα a IL-1β indukovalo produkci miR-146. In situ hybridizace odhalila, že miR-146a je primárně exprimována buňkami povrchové a podvýstelkové vrstvy, zejména pak CD 68+ makrofágy, ale též CD3+T buňky a CD79+ B lymfocyty (61).
Vyšetření periferní krve RA pacientů (PBMC, CD4+T buňky, IL-17 T lymfocyty, plazma) detekovala hyperexpresi této microRNA (59, 62, 63, 68, 71). In vitro studie ukázala, že potlačení TRAF a IRAK1, cílových molekul miR-146a v lidských THP1 (human acute monocytic leukemia cell line) monocytech vede k významnému snížení produkce TNFα (59). Autoři se proto domnívají, že miR-146 u RA nedostatečně reguluje TRAF/IRAK1, což vede k nadprodukci TNFα (59). Li et al., kteří analyzovali RA CD4+ T lymfocyty synovie a periferní krve nalezli pozitivní korelaci hladin miR-146 s hladinou TNFα. In vitro stimulace pomocí TNFα indukovala expresi miR-146a. Transfekcí zvýšená hladina miR-146a potlačila apoptózu Jurkat T buněk. Transkriptomová analýza T buněk navíc identifikovala Fas associated factor 1 (FAF1), zahrnutý v modulaci T buněčné apoptózy jako cílovou molekulu této miRNA (62).
Chatzykyriakidou et al. informují, že nosičství polymorfismu IRAK1 (rs3027898 A > C ), cílového genu miR-146a, je v řecké populaci asociováno s vnímavostí ke vzniku RA (111).
miR-146a jako biomarker a potenciální terapeutikum
Zvýšené koncentrace miR-146 v CD4+T buňkách korelují s DAS-28 (62). Stejný závěr týkající se PBMC učinily dvě studie (59,70). Plazmatické hladiny a poměr množství miR-146a v synoviální tekutině a plazmě vykázaly vztah k počtu bolestivých kloubů (TJC) (63). Hladiny miR-146a v CD4+ T lymfocytech navíc korelovaly s množstvím TNFα (62). Pozitivní korelaci miR-146a s TNFα a ESR v PBMC konstatují Abou-Zeid et al. (70).
Potenciální léčebné využití miR-146a zkoumali Nakasa et al. In vitro studie odhalila, že tento typ inhibuje osteoklastogenezi. Intravenózní podávání dvouvláknové miR-146a myšímu modelu s kolagenem indukovanou artritidou chránilo před vznikem kostních a chrupavčitých destrukcí. Terapie mírnila synoviální hyperplasii a produkci cytokinů (TNFα, IL-1β, IL-6), vzniku kloubního zánětu však zabránit nedokázala (75).
miR-155
Obecné poznámky:
Aberantní exprese je součástí patogeneze hematologických malignit (112–114), solidních tumorů (115–117) ale i systémových revmatických onemocnění: RA (59, 60, 63, 68, 72), SLE (118), SjS (119, 120), PM, DM (121). Četné studie popisují regulační úlohu miR-155 v několika typech imunocytů. (B lymfocyty, DC, Th17 lymfocyty, Treg) (122). Myši deficientní pro miR-155 manifestují poškozenou T a B buněčnou odpověď a dysfunkci DC (123, 124). Knockout (cílené vyřazení) této miRNA omezil počet germinálních center, snížil produkci cytokinů a narušil afinitní maturaci B buněk. Podíl miR-155 na aktivaci T lymfocytů je realizován vazbou na transkripční faktor c-Maf, aktivátor transkripce IL-4, který podporuje diferenciaci imunitní odpovědi do Th2 fenotypu (123, 125). Vyřazení miR-155 vede k redukci Th17 a defektní produkci IL-17A (126). Snížené hladiny IL-17 a IL-22 v T lymfocytech miR-155 -/ - myší pozorovali Blüml et al. (127). O zapojení miR-155 ve vývoji Treg buněk referovaly dva výzkumy (128, 129). Úlohu této miRNA jako negativního modulátoru IL-1 dráhy lidských DC zmiňují Ceppi et al. Stejní autoři vyslovují hypotézu, že miR-155 může participovat v negativní zpětnovazebné smyčce kontrolující IL-1β a další zánětlivé cytokiny produkované během LPS zprostředkované aktivace dendritických buněk (130). Lu et al. ukázali, že hyperexprese miR-155 indukuje apoptozu lidských DC, avšak též patogenní odpovědi DC skrze podporu sekrece specifických cytokinů (IL-12p70). Získaná data naznačují, že zvýšené hladiny miR-155 ve zralých DC ovlivňují produkci IFN-γ NK buňkami (131). O´Conell et al. zaznamenali signifikantní pokles exprese genů pro prozánětlivé cytokiny (IL-12, IL-6, IL-23p19, IL-23p40) v miR 155 -/ - dendritických buňkách. Vynucená exprese miR-155 zvýšila produkci TNFα (126). K potvrzeným cílům miR-155 patří transkripční faktor PU.1 – klíčový regulátor ontogeneze myeloidní linie, AID zapojený v afinitní maturaci a diverzifikaci imunoglobulinů, antionkogen SHIP1, SOCS1 – negativní regulátor cytokinové signalizace či transkripční faktor BACH - blokátor buněčné proliferace.
Funkce u RA:
Zvýšené hladiny miR-155 v RA synoviální tkáni, synoviální tekutině i fibroblastech ve srovnání s OA vzorky detekovaly 3 studie (60, 63, 72). RA synoviální monocyty (CD14+) vykázaly 4,4krát vyšší expresi než RA PBMC (60). Stimulace RASF a synoviální tkáně TNFα u RA a OA pacientů indukovala hyperexpresi miR-155 v RA vzorcích (60). Murata et al. (63) ukázali, že miR-155 je přítomna v synoviální tkáni a potvrdili její zvýšené koncentrace v synoviální tekutině pacientů s RA. Nadměrnou expresi miR-155 v makrofázích synoviální membrány a tekutiny konstatují též Kurowska-Stolarska et al. (72).
Srovnávací analýzy plazmy a PBMC RA pacientů a zdravých kontrol zjistily zvýšené hladiny miR-155 u RA (59, 53, 68, 72). Protikladná data uvádějí Stanczyk et al. (60), kteří rozdíl v expresi mezi RA PBMC a PBMC zdravých subjektů nenalezli. Niimoto et al. shledali, že množství miR-155 bylo zvýšeno během diferenciace T lymfocytů produkujících prozánětlivý IL-17 (68).
Funkční studie RASF demonstrovaly zapojení miR-155 v potlačování matrixových metaloproteináz. Vynucená exprese miR-155 snižuje množství MMP-3 a MMP-1 a jejich indukci TLR ligandy a cytokiny. MiR-155 tak pravděpodobně slouží k ochraně před tkáňovým poškozením (60).
Nadměrná exprese miR-155 v CD 14+ makrofázích synoviální tekutiny asociovala s redukcí hladin SHIP1 (Src homology 2-containing inositol phosphatase-1), cílového genu miR-155 , který inhibuje zánět. Exprese SHIP1 byla snížena též v RA CD68(+) buňkách výstelkové vrstvy synovie. Zvýšení exprese miR-155 v CD14+ buňkách periferní krve snížilo množství SHIP1 a vedlo k nadprodukci cytokinů a chemokinů asociovaných s revmatoidní artritidou (TNFα, IL-6). Inhibice miR-155 v RA synoviálních CD14+ makrofázích redukovala množství TNFα (72).
Dle stejné studie jsou miR-155 deficientní myši odolné proti kolagenem indukované artritidě (72). Publikované závěry souhlasí s výsledky Blümla et al. (127) a naznačují potenciální terapeutické využití této miRNA.
MiR-155 jako biomarker:
Zatímco plazmatické hladiny miR-155 inverzně korelovaly s počtem bolestivých kloubů (TJC), poměr této miRNA v plazmě a séru koreloval s TJC pozitivně (63).
miR-203
Obecné poznámky:
Typ specifický pro kůži a keratinocyty, nadměrně vyjádřen v epidermis pacientů s psoriázou (132). Deregulován u karcinomu prostaty (133, 134), GIT (135-138), ale i dalších onkologických procesů.
Funkce u RA:
Exprese miR-203 je v RA synoviálních fibroblastech zvýšena, regulace v RASF souvisí s metylačními procesy: přidání DNA demetylačního činidla 5-azacytydinu vedlo k hyperexpresi této miRNA. Zvýšené množství miR-203 zvyšuje hladiny MMP1 a IL-6. Bazální hladiny miR-203 korelovaly s bazálními hladinami IL-6. MiR-203 reguluje IL-6 skrze signální dráhu NFκB (65).
miR-223
Obecné poznámky:
Zapojena v hematopoéze (diferenciace myeloidní řady, granulopoeza), rozdílně vyjádřena u leukemií a dalších malignit (139–141). Sérová miR-223 může být potenciálním biomarkerem sepse (142).
Funkce u RA:
Fulciho studie detekovala miR-223 jako jedinou miRNA deregulovanou v periferních, zejména CD3 a CD4+ T lymfocytech RA pacientů (nadměrná exprese) (64). Tento nález lze považovat za aberantní asociaci s patologií, protože CD4+T lymfocyty zdravých dárců neexprimovaly miR-223 ani po TCR stimulaci. Expresní profilování RA a OA synoviocytů provedené Kawanem ukázalo významně zvýšené hladiny miR-223 v RASF ve srovnání OASF (67). Koncentrace miR-223 v synoviální tekutině jsou u RA i OA pacientů nižší než hladiny plazmatické (63). Exprese v RASF je slabá, množství miR-223 je díky infiltrujícím mononukleárům zvýšeno v punktátech RA nemocných (63).
miR-223 jako biomarker:
Vyšetření synoviální tekutiny odlišuje RA (zvýšená exprese) a OA nemocné. Množství miR-223 v plazmě inversně korelovalo s počtem bolestivých kloubů (63).
miR-346
Funkce u RA:
MiR-346 je nadměrně exprimována ve fibroblastech podobných synoviocytům (FLS) stimulovaných LPS. Nepřímo snižuje expresi IL-18 skrze inhibici produkce LPS indukované Brutonovy tyrosin kinázy, která stabilizuje mRNA některých cytokinů (včetně TNFα) (69).
Navazující studie naznačila, že v LPS aktivovaných RA FLS reguluje miR-346 syntézu TNFα skrze posttranskripční tlumení tristetraprolinu (TTP). TTP je RNA vážící protein, inhibitor syntézy TNFα (destabilizace mRNA). Lipopolysacharidem stimulované RA FLS exprimují pouze TNF-α mRNA, ne však zralý protein. Blokace miR-346 však produkci tohoto cytokinu obnovila (73).
Systémový lupus erythematodes (SLE)
Profilovací studie SLE identifikovaly početné množství diferenciálně vyjádřených microRNA. Mnoho z nich však bylo detekováno výhradně mikročipy (143, 144) a vyžaduje potvrzení kvantitativní RT-PCR či Northern blottingem. Několik prací se omezilo na vyšetření expresních signatur, aniž by zkoumalo funkci deregulovaných miRNA (107, 143, 144, 145, 146). Pět studií popsalo příspěvek diferenciálně vyjádřených typů k patogenezi SLE. MicroRNA ovlivňují signalizaci IFN I typu (57), metylaci DNA (147, 148) expresi RANTES (149) a regulují aberantní T buněčné odpovědi (150). Závěry některých experimentů se neshodují: snížené hladiny miR-146a v SLE PBMC pozorovali Tang et al. a Hai-yan et al. (57, 58), ne však ostatní výzkumníci (143, 145).
Úvodní pokus o stanovení expresního profilu v SLE PBMC podnikli v roce 2007 Dai et al. (143). S využitím mikročipové analýzy (microarray) odhalili deregulaci šestnácti microRNA ve srovnání se vzorky zdravých kontrol.
Další studie stanovily profil v PBMC (57, 58, 145, 149, 150), CD4+ T lymfocytech (147, 148, 150), B-buněčných liniích transformovaných EB virózou (145), ledvinových biopsiích pacientů s lupusovou nefritidou (144), séru a močovém supernatantu (107, 146). Podrobnosti týkající se provedených výzkumů uvádí tabulka 3.
Postatné informace o miRNA funkci a roli jako biomarkeru u SLE lze shrnout následovně:
miR-21
Obecné poznámky:
Aberantní exprese miR-21 je asociována s kancerogenezí (151–155), zapojení v zánětlivých cestách popsalo několik prací. Lu et al. zaznamenali zvýšenou indukci miR-21 u alergického astmatu a identifikovali cílovou molekulu miR-21: podjednotku p 35 prozánětlivého cytokinu IL-12 (156). Sheedy et al. odhalili významnou úlohu miR-21 v TLR 4 signalizaci. V PBMC tlumí miR-21 expresi PDCD4, klíčového regulátora zánětlivé odpovědi na LPS (157). PDCD4 je inhibitor translace, tumor supresorový protein a přímý cíl miR-21 (158, 159). Přesná funkce u lymfocytů není objasněna, pravděpodobně ovlivňuje proliferaci či apoptózu (160). Podle Sheedyho podporuje aktivaci prozánětlivého NFκB a potlačuje protizánětlivý IL-10. Inhibice PDCD4 pomocí miR-21 zvyšuje produkci IL-10, snižuje NFκB aktivitu a pravděpodobně kontroluje LPS odpověď: PDCD4 deficientní myši byly chráněny před smrtí vyvolanou LPS (157). Indukci miR-21 resolvinem D1 a její potenciální úlohu v mírnění akutního zánětu zmiňují Recchiuti et al. (161). Protikladnou funkci v MCF-10A buňkách popisují Iliopoulos et al. (162). MiR-21 charakterizují jako součást pozitivní zpětnovazebné smyčky spojující zánět s epigeneticky navozenou maligní transformací. Transkripční faktor STAT3 aktivovaný IL-6 zvyšuje expresi miR-21 a miR-181b-1 a indukuje tak přeměnu buněk. MiR-21 reprimuje tumor supresor PTEN, což vede k aktivaci NFκB a podpoře onkogeneze. Úloha miR-21 v NFκB signalizaci a regulaci zánětu se tak zřejmě liší v závislosti na buněčném typu, obecně však zůstává nejasná. Účast miR-21 v nepřímé pozitivní regulaci vývoje Treg buněk zmiňují Rouas et al. (163).
Funkce u SLE:
Zvýšenou expresi miR-21 v SLE CD4+ T lymfocytech a PBMC (B i T buňkách) zaznamenaly tři výzkumy (143,148,150). Pan et al. shledali, že miR-21 potlačuje RAS guanyl nucleotide-releasing protein 1, regulátor DNA metyl transferázy 1 (DNMT1). Zvýšené množství miR-21 v CD4+ T buňkách nepřímo snižuje hladiny DNMT a způsobuje hypometylaci DNA a hyperexpresi dvou autoimunitních genů: CD70 a LFA-1 (lymphocyte function associated antigen) (148). Stagakis et al. zjistili, že vazbou na PDCD4 reguluje miR-21 abnormální T buněčné odpovědi. Hladina PDCD4 je u aktivního SLE snížena. Indukovaná hyperexprese miR-21 ve zdravých T buňkách způsobila konverzi do SLE fenotypu. Potlačení miR-21 v SLE CD4+T lymfocytech zvrátilo aktivovaný fenotyp, snížilo proliferaci, produkci IL-10, expresi CD40L a omezilo kapacitu T buněk řídit zrání B lymfocytů do Ig sekretujících CD19+CD38-IgD plazmatických buněk (150).
miR-21 jako biomarker a potenciální terapeutikum:
MiR-21 je zvýšeně exprimována v T i B SLE lymfocytech a vykazuje silnou korelaci s aktivitou onemocnění (r 2 =0,92) (150).
Nadměrné hladiny miR-21 v B a T lymfocytech myšího modelu s SLE (B6.Sle123) nalezli Garchow et al. Blokace miR-21 pomocí anti-miR LNA potlačila splenomegalii, hlavní klinický projev u B6.Sle123 myší a obnovila expresi PDCD4. Léčba navíc změnila poměr CD4/CD8 T buněk a snížila populaci lymfocytů exprimujících Fas receptor. Autoři demonstrovali, že terapie pomocí LNA účinně potlačuje endogenní miRNA expresi v B a T lymfocytech a mění průběh genetické choroby u myší (164).
miR -125a
Obecné poznámky:
Figuruje v patogenezi široké škály nádorů (165–169). Guo et al. ukázali, že miR-125a zvyšuje počet hematopoetických kmenových buněk (170). Účast v regulaci homeostázy cholesterolu, metabolismu mastných kyselin a lipogenezi popsali Fernández et al. (171).
Funkce u SLE:
Snížené množství miR-125a v PBMC pacientů s SLE nalezli Tang et al. (57), navazující funkční studie odhalila zvýšené hladiny kandidátního cílového proteinu KLF13 (Kruppel like factor) v aktivovaných T buňkách (149). KLF13, též nazývaný RFLAT-1 (RANTES Factor of Late Activated T Lymphocytes) je člen skupiny transkripčních faktorů, které regulují expresi zánětlivých chemokinů RANTES v T lymfocytech (172). RANTES patří do rodiny CC (alfa) chemokinů a hraje důležitou roli v imunopatogenezi SLE, ale i dalších poruch, včetně AIDS. Ektopická exprese miR-125a snižuje množství RANTES a KLF13 v aktivovaných T lymfocytech. Studie ukázala, že miR-125a je zapojena do zpětnovazebné regulační smyčky (vazba na 3´UTR KLF13), která řídí expresi RANTES v konečné fázi diferenciace T lymfocytů. Zjištěná fakta naznačují, že miR-125a může sloužit jako potenciální terapeutický cíl, ovlivňující produkci zánětlivých cytokinů u SLE (149).
miR-126
Obecné poznámky:
Představuje pro endotel specifický typ, klíčový pozitivní regulátor angiogenní signalizace a cévní integrity in vivo (173). MiR-126 řídí tvorbu cév a může ovlivňovat neovaskularizaci nádorů (174). Inaktivace (knockout) miR-126 v embryogenezi dánia pruhovaného či její delece u myší způsobily defekty cévního vývoje. Buňky endotelu s deficitem miR-126 nereagovaly na působení angiogenních faktorů včetně VEGF (vascular endothelial growth factor), EGF (epidermal growth factor) a bFGF (basic fibroblast growth factor). Dva přímé cíle miR-126: Spred1 (Sprouty-related EVH1 domain-containing protein 1) a PIK3R2 (phosphatidylinositol 3-kinase regulatory subunit beta), negativní regulátory buněčných signálních kaskád ovlivňují MAPK (mitogen-activated protein (MAP) kinases) a PIK3 (phosphinositide 3-kinase) signalizaci a inhibují proliferaci v endoteliálních buňkách indukovanou VEGF(173).
Význam angiogenních faktorů u SLE je znám. Zejména SLE pacienti s kardiovaskulárními komplikacemi produkují zvýšené hladiny VEGF (175), úlohu tohoto cytokinu v patogenezi SLE zkoumalo několik studií (176–180). Biologický účinek VEGF na subpopulaci CD4+T buněk, včetně kostimulace produkce IFN-γ popsali Basu et al. (181). Lu et al., detekovali VEGF SNP (rs833070), který může přispívat k vnímavosti k SLE u populace severních Chanů (182).
Funkce u SLE:
Zvýšenou hladinu miR-126 v CD4+T lymfocytech pozorovali Zhao et al. Podrobné zkoumání etiopatogenetické úlohy odhalilo, že množství miR-126 negativně koreluje s hladinou DNA metyltransferázy 1 (DNMT1). Vazbou na 3´UTR inhibuje miR-126 translaci DNMT1 mRNA. Vynucená exprese miR-126 v CD4+T buňkách zdravých dárců způsobila demetylaci, hyperexpresi genů kódujících molekuly CD11 a CD70 a následnou hyperaktivitu T a B buněk. Potlačení této miRNA v SLE CD4+T lymfocytech mělo opačný efekt. Snížená exprese DNMT1 v CD4+ T-buňkách tak přispívá k lupusové autoreaktivitě, protože způsobuje derepresi autoimunitních genů závislou na hypometylaci DNA (147).
miR-146a
Obecné poznámky:
viz kapitola RA
Funkce u SLE :
Snížené hladiny miR-146a v PBMC SLE pacientů zaznamenaly dvě studie (57, 58), ostatní práce však tento fakt nepotvrdily (143–145). Tang et al. objevili vztah mezi aktivací signální cesty IFN I typu a redukovanou expresí miR-146a. Ukázali, že miR-146a reguluje hladiny TRAF6, IRAK1, STAT-1, a IFN regulačního faktoru 5 (IRF-5), důležitých mediátorů IFN cesty. Hyperexprese miR-146a v primárních PBMC vedla ke snížení produkce IFNα a IFNβ, zprostředkované TLR7. Transfekce syntetických oligonukleotidů potlačujících endogenní expresi miRNA-146a zvýšila hladiny IFN v PBMC. Autoři konstatují, že deficience miR-146a může být jednou z příčin abnormální aktivace IFN I typu u SLE, přesný mechanismus deregulace však zůstal nedořešen (57).
MiR-146a je kódována na krátkém raménku 5 chromozomu (5q33.3), v regionu který je spojen s vnímavostí k SLE (183–185). Studie leukocytů periferní krve navazující na Tangův výzkum identifikovala s SLE asociovaný SNP (rs57095329), nalézající se v promotorové oblasti genu pro miR-146a, jehož nosičství způsobuje redukci hladin této miRNA (186). Kombinované funkční testy ukázaly, že riziková alela G snižuje v porovnání s protektivní alelou A afinitu „transkripčních” proteinů a aktivitu miR-146 promotoru. V blízkosti G varianty se váže transkripční faktor Ets-1, jehož gen je podle GWAS spojen s vnímavostí k SLE (185). In vitro experiment prokázal, že změny hladin Ets-1 ovlivňují aktivitu miR-146a promotoru: knockdown (redukce genové exprese) Ets-1 simulující jeho deficit u SLE přímo porušil indukci miR-146a. Luova práce tak podala první popis asociace funkční genetické varianty v miRNA promotoru s lidskou chorobou a prokázala, že nositelé SNP rs57095329 exprimují nižší hladiny miR-146a (186).
miR-146a jako biomarker:
Dle Tanga vykazuje exprese miR-146a v PBMC slabou negativní korelaci s hodnocením aktivity nemoci dle SLEDAI a renálního SLEDAI (r-hodnoty 0,28 resp. 0,38) (57). Množství cirkulující miR-146a v séru a močovém supernatantu pacientů s SLE měřili Wang et al. Nemocní měli nižší sérové hladiny miR-146a než zdravé kontroly, množství miR-146a v moči pacientů bylo signifikantně zvýšeno. Odhadovaná glomerulární filtrace korelovala s kvantitou této microRNA v séru. Sérové množství miR-146a bylo v inverzním vztahu s mírou proteinurie a aktivitou onemocnění (SLEDAI). Další šetření ukázalo korelaci sérové miR-146a s počtem červených krvinek, krevních destiček a lymfocytů. Po šetiměsíční léčbě kalcitriolem významně klesla hladina miR-146a v séru pacientů, tato změna negativně korelovala s mírou kalcio-fosfátového součinu (107).
miR-148a
Obecné poznámky:
Asociována s patogenezí nádorového bujení (kancerogenezí). Bazální expresi vykazuje pojivová, endokrinní tkáň (187) a žaludek (188), hladiny jsou potlačeny v buňkách pupečníkové krve (189). U pankreatických tumorů je exprese umlčena hypermetylací genu pro miR-148a (190). Epigenetickou inaktivaci miR-148 v metastatických rakovinných buněčných liniích popisují Lujambio et al. (191). Vazbu této miRNA na několik isoforem DNA metyltransferázy (DNMT)3b odhalili Duursma et al. (192). Úlohu v regulaci vrozené imunitní odpovědi zkoumali Liu et al., kteří demonstrovali, že tři členové rodiny miR-148 účinkují jako negativní regulátory (inhibitory) vrozené imunitní odpovědi a antigen prezentující kapacity DC. Vazbou na CaMKIIα (calcium/calmodulin-dependent protein kinase II), významný regulátor zrání a funkce dendritických buněk redukuje miR-148 produkci cytokinů včetně IL-12, IL-6, TNFα. Stejně tak snižuje hyperexpresi MHC II proteinů indukovanou IFN-β a antigen-specifickou proliferaci T lymfocytů iniciovanou dendritickými buňkami (193).
Funkce u SLE :
Pan et al. zjistili hyperexpresi miR-148 v CD4+ T buňkách pacientů s SLE a MRL/lpr myší náchylných k lupusu. Studie potvrdila vazbu miR-148 na protein kódující oblast DNA metyltransferázy 1 (DNMT1). Zvýšené množství miR-148 v CD4+ T lymfocytech snižuje metylaci DNA a způsobuje hyperexpresi s autoimunitou asociovaných genů CD70 a LFA-1 (lymphocyte function associated antigen 1) (148).
miR-155
Obecné poznámky:
viz kapitola RA
Funkce u SLE:
Dai et al. detekovali zvýšené množství miR-155 ve splenocytech dvou geneticky odlišných myších modelů disponovaných k vývoji lupusu (MRL-lpr, B6-lpr) (194). Hyperexpresi vykázaly i B a T lymfocyty kmene MRL-lpr. Elevaci hladin miR-155 v purifikovaných T regulačních lymfocytech (Treg) MRL-lpr myší shledali Divekar et al (195). Zkoumané CD4+CD25+Foxp3+ Treg buňky manifestovaly změněný fenotyp a sníženou supresorickou schopnost. Zmíněné alterace mohly být způsobeny sníženou hladinou DICERu a deregulací některých miRNA typů včetně miR-155.
miR-155 jako biomarker:
Wang et al. měřili hladiny cirkulující miR-155 v séru a močovém supernatantu pacientů s SLE. Nemocní produkovali nižší sérové hladiny miR-155 než zdravé kontroly, množství miR-155 v močovém supernatantu nemocných a kontrol se nelišilo. Sníženou expresi miR-155 v séru pacientů však mohla zapřičinit imunosupresivní léčba, kterou podstoupilo všech 40 sledovaných. Sérová hladina miR-155 inverzně korelovala s dávkou prednisolonu (r = 0,387, p = 0,014). Odhadovaná glomerulární filtrace vykázala vztah k sérové kvantitě miR-155 (107).
miR-192, 200, 205
miR-192, 200, 205 jako biomarkery:
V moči a séru SLE nemocných detekovali Wang et al. microRNA z rodiny miR-200, miR-205 a miR-192 (146). Předchozí studie ukázaly, že tyto typy snižují expresi ZEB1 a ZEB 2 (Zinc finger E-box-binding homeobox), molekul účastnících se procesů epiteliální mezenchymální transformace (EMT) a renální fibrózy (196, 197). Pacienti s SLE měli signifikantně nižší sérové hladiny miR-200a, miR-200b, miR-200c, miR-429, miR-205 a miR-192 než zdravé kontroly, nalezené množství navíc souviselo s indexem aktivity onemocnění (SLEDAI). Vyšetření moči poskytlo obdobné výsledky, avšak s nižší klinickou relevancí. Sérové microRNA vykázaly následující korelace: odhadovaná glomerulární filtrace korelovala s kvantitou miR-200b, miR-200c, miR-429, miR-205 a miR-192, míra proteinurie byla v inverzním vztahu k množství miR-200a a miR-200c. SLEDAI negativně koreloval s hladinou miR-200a. Kvantita mir-200b a miR-192 souvisela s počtem trombocytů, miR-205 pak s množstvím erytrocytů a hematokritu. Obě Wangovy práce naznačily možnou diagnostickou hodnotu sérových microRNA a jejich zapojení v patofyziologii SLE (107, 146).
Sjögrenův syndrom (SjS)
Úlohu miRNA u SjS zkoumali Alevizos et al. (119, 198, 199). V nedávné studii stanovili expresní profily menších slinných žláz šestnácti SjS pacientů a osmi zdravých kontrol. Soubor nemocných zahrnoval bioptáty s lehkým (focus score 1–2) a těžkým stupněm zánětu (focus score 12) respektive s normální a s porušenou exokrinní funkcí. Charakteristické expresní vzory detekované pomocí mikročipů přesně odlišily tkáně pacientů a kontrol, stejně jako vzorky s různou mírou zánětlivé infiltrace. Překvapující byl rozdíl miRNA signatur zdravých a lehce zanícených žláz, které vykazují jen minimální histologické diference. K potvrzení výsledků mikročipové analýzy a posouzení potenciálu microRNA odlišujících pacienty s různou intenzitou zánětu byly vybrány dva signifikantně vyjádřené typy: miR-768-3p a miR-574. Exprese miR-768-3p se zvyšujícím se focus score rostla, množství miR-574 naopak klesalo (negativní korelace). Navazující kvantitativní RT-PCR analýza provedená na nezávislé kohortě 15 pacientů potvrdila tyto dvě miRNA jako ukazatele zánětlivého procesu.
Porovnání normo a hypofunkčních žláz odhalilo devět rozdílně exprimovaných microRNA (miR-765, miR-181a, miR-766, miR-335, miR-16, miR-671, miR-663, miR-340, miR-155). V souboru žláz se sníženou slinnou sekrecí bylo sedm z nich nadměrně exprimováno (miR-765, miR-181a, miR-766, miR-16, miR-671, miR-663, miR-155). Cílové geny těchto hyperexprimovaných microRNA mohou chránit epiteliální buňky před poškozením. Biostatistická predikce potenciálních cílových molekul u microRNA asociovaných se zánětem a dysfunkcí slinných žláz ukázala zapojení do překrývajících se či odlišných biologických cest a procesů. Hlavní předpokládané cíle však zastávaly neurologické funkce. Neuroregulace slinných žláz tak může hrát významnou roli v patogenezi SjS (119).
Michael et al. izolovali exosomální microRNA ze slin SjS nemocných a zdravých jedinců. Množství miRNA extrahované z exosomální frakce dostačovalo pro kvantitativní PCR a mikročipové analýzy. Možnost microRNA extrakce z neinvazivně získaného materiálu tak rozšířila perspektivy využití miRNA molekul jako markerů patologií slinných žláz (200).
Pauley et al. studovali expresi miR-146a, miR-155 a miR-132 v PBMC SjS pacientů, analyzovali též PBMC, slinné a slzné žlázy myšího modelu. Hladiny miR-146a a miR-155 byly u SjS nemocných ve srovnání se zdravými subjekty signifikantně zvýšeny (miR-146a= 8,0x, p < 0,0001, miR-155=2,5x, p < 0,05). Myši disponované k vývinu SjS vykázaly hyperexpresi miR-146a v PBMC a slinných žlázách. Elevace hladin miR-146a v PBMC myší byla zaznamenána ještě před propuknutím choroby, což naznačuje potenciál miR-146a jako biomarkeru. Funkční experimenty cílené na lidské THP-1 monocyty ukázaly, že hyperexprese miR-146a zvyšuje fagocytickou aktivitu buněk a potlačuje produkci prozánětlivých cytokinů (TNF-α, IL-1β, MIP-1a, IP-10, IL-6) (108).
Systémová sklerodermie (SS)
Funkční studii posttranskripční regulace profibrotických genů u systémové sklerodermie provedli Maurer et al. S využitím biostatistických predikčních algoritmů identifikovali několik miRNA typů regulujících geny pro kolagen (let-7, miR-26, miR-155, miR-29a/29b/29c). K následným analýzám vybrali evolučně konzervovanou rodinu miR-29 s předpokládaným vysokým stupněm komplementarity k cílové mRNA. Kromě kolagenu reprimuje miR-29 pravděpodobně i další profibrotické geny: PDGF-B (platelet-derived growth factor), PDGFR (platelet-derived growth factor receptor). Sp-1, thrombospondin a SPARC (secreted protein acidic and rich in cysteine/osteonectin/BM40). Exprese miR-29 byla stanovna kvantitativní RT-PCR analýzou kožních bioptátů a fibroblastů nemocných se systémovou sklerodermií a zdravých kontrol. Zkoumány byly i myši s kožní fibrózou navozenou bleomycinem. Ve fibroblastech i kožních řezech SS pacientů, stejně jako u myšího modelu byla zjištěna významně snížená exprese miR-29a. Tento výsledek souhlasil s očekáváním, protože geny pro kolagen jsou u SS nemocných hyperexprimovány. Transfekce SS fibroblastů prekurzorovou miR-29a snížila hladiny mRNA i proteinu pro kolagen I a III typu, vyřazení miR-29a z činnosti ve fibroblastech zdravých dárců tyto hladiny zvýšilo. Test odpovědi reportérového genu potvrdil COL3A1 (gen pro kolagen) jako přímý cíl miR-29a. Stimulace profibrotickými mediátory: TGF (transforming growth factor), PDGF-B, či IL-4 redukovala množství miR-29a ve zdravých kožních fibroblastech. Inhibice PDGF-B a TGF drah imatinibem obnovila hladiny miR-29a v SS fibroblastech jak in vitro, tak in vivo u myší. MiR-29a přímo reguluje expresi kolagenu u SS pacientů. Získaná data naznačila, že deregulace miR-29a je alespoň částečně zprostředkována profibrotickými cytokiny a růstovými faktory. Vzhledem k povzbudivým výsledkům genové microRNA terapie u zvířat se miR-29 jeví jako potenciální cíl léčby fibrinogeze u systémové sklerodermie (201).
Diferenciální expresi miRNA v kožní tkáni nemocných určovali Li et al. Výzkum byl však proveden na nízkém počtu probandů (3 nemocní, 3 kontroly). Mikročipová analýza ukázala 24 rozdílně vyjádřených micro RNA u SS pacientů. Devět typů bylo zvýšeně, patnáct sníženě exprimováno. Výsledky potvrdila kvantitativní RT-PCR. Bioinformatický rozbor nalezl šest microRNA s možným vztahem k patogenezi onemocnění (miR-23b, let-7 g, miR-125b, miR-133a, miR-206, miR-140-5p). Jako nejvýznamnější se autorům jeví miR-206 – potenciální regulátor 15 genů podílejících se na vzniku a rozvoji onemocnění (TGFb1, TGFb2, SMAD5, FGF2, FGF5, FGFR3, IL-1β, IL1R AP (receptor accessory protein), IL-21, integrina2, cyclinT2, FGb (fibrinogenb chain), COL6a3, COL29a2 and CD28) (202).
Idiopatické zánětlivé myopatie (IZM)
Doposud jedinou expresní studii u IIM publikovali Eisenberg et al. (121), kteří zkoumali miRNA signatury v kosterním svalu 10 primárních myopatií. U DM bylo identifikováno 33 u PM 28 deregulovaných miRNA. Hyperexprese miR-146b a Ambi-miR-4983 u PM odlišila polymyositický vzorek od zdravého ve 100 % případů. U všech zkoumaných chorob bylo aberantně vyjádřeno pět miRNA typů (miR-146b, miR-221, miR-155, miR-214 a miR-222) (121, 203).
Závěr
Výzkumy posledních let potvrdily microRNA jako klíčové regulátory savčího genomu. Počet publikací definujících roli miRNA v imunitním systému rapidně narůstá. Získaná data naznačují, že deregulace těchto molekul přispívá k vývoji systémových autoimunitních onemocnění. Přestože přesná úloha zůstává nejasná, jsou známy dílčí funkce některých microRNA. Např. miR-146 a miR-155 ovlivňují vrozenou i adaptivní imunitní odpověď, ontogenezi, diferenciaci a funkci lymfocytů. Aberantní exprese miR-146a či miR-155 byla zaznamenána u RA, SLE, psoriázy, SjS, DM a PM. Obě microRNA regulují geny zapojené v patogenezi revmatických onemocnění. Funkční studie poodhalily mechanismus účinku i u dalších typů. Příkladem jsou asociace miR-126 a miR-203 s metylačními procesy. Četné práce naznačily potenciál microRNA jako biomarkerů či terapeutických cílů.
Vzhledem k charakteru regulace (polyvalence, redundance, mírný účinek) lze předpokládat, že patogenní efekt způsobuje synergicky větší počet microRNA. K pochopení etiopatogenetické úlohy je třeba znát příčinu deregulace, cílové geny a jejich působení. Tyto otázky pomohou zodpovědět metodicky jednotné studie na jasně definovaných kohortách.
Poděkování: Podpořeno Interní grantovou agenturou Ministerstva zdravotnictví ČR v rámci projektu NT 12452-4.
Seznam zkratek
- ARE – motivy bohaté na adenosin a uridin (AU rich elements)
- BACH – transkripční faktor, BTBand CNC homolog
- B-buňka (lymfocyt) – buňka imunitního systému zodpovědná především za specifickou, protilátkami zprostředkovanou imunitní odpověď
- Bcl-2 - protoonkogen blokující apopotózu
- bFGF - bazický fibroblastový růstový faktor (basic fibroblast growth factor)
- CD – rozlišovací shluk (cluster of differenciation)
- CDK – cyklin dependentní kináza
- COL – gen pro kolagen
- CRP – C reaktivní protein
- DAS – skóre aktivity nemoci (dinase aktivity score)
- DC – dendritické buňky (dendritic cells)
- DNA - deoxyribonukleová kyselina
- DNMT – DNA metyltransferáza
- DM – dermatomyositida
- EBV – virus Epsteina Baarové
- ESR – sedimentace erytrocytů (erythrocyte sedimentation rate)
- EGF – epidermální růstový faktor (epidermal growth factor)
- FADD – Fas associated-death domain protein
- HCMV – lidský cytomegalovirus
- HDL – vysokodenzitní lipoprotein (high-density lipoprotein)
- HIV – virus lidské imunodeficience
- HSV – herpes simplex virus
- IFN – interferon
- IkB – inhibitor NFκB
- IL – interleukin
- IRAK – interleukin receptor activated kinase
- IRF-5 (interferon regulatory factor)
- ITP – idiopatická trombocytopenická purpura
- KLF – Kruppel like factor
- KSHV – herpes virus asociovaný s Kaposiho sarkomem
- LNA – speciálně modifikované oligonukleotidy - locked nucleic acid
- LPS – lipopolysacharid
- MCP – chemotaktický protein (monocyte chemotactic protein)
- MHC – hlavní histokompatibilné komplex
- MICB – MHC class I polypeptide-related sequence B
- miRNA – micro ribunokleová kyselina (micro ribonucleic acid)
- MMP – matrixová metaloproteináza (matrix metalloproteinase)
- mRNA – informační ribonukleová kyselina (messenger ribonucleic acid)
- ncRNA – nekódující RNA molekuly
- NFκB – jaderný transkripční faktor κB (nuclear transcripting factor κB)
- OA – osteoartróza
- OASF – synoviální fibroblasty pacientů s osteoartrózou
- PBMC – mononukleární buňky periferní krve (peripheral blood mononuclear cells)
- PDCD – protein programované buněčné smrti (programmed cell death protein)
- PDGF – platelet derived growth factor
- PDGFR – platelet-derived growth factor receptor
- PFV-1 – primate foamy virus
- piRNA – Piwi interacting RNA
- PM – polymyositida
- Pol II – DNA dependentní RNA polymeráza typu II
- pre-miRNA – prekurzorová miRNA (precursor miRNA)
- pri-miRNA – primární transkript miRNA
- PTPN22 – protein tyrosine phosphatase non-receptor type 22
- RA – revmatoidní artritida
- RANKL – receptor activator of nuclear factor kappa B ligand
- RASF – synoviální fibroblasty pacientů s revmatoidní artritidou (rheumatoid arthritis synovial
- fibroblasts)
- RFLAT – RANTES Factor of Late Activated T Lymphocytes
- RISC – RNA induced silencing complex
- RNAi – interference ribonukleových kyselin
- rRNA – ribonukleová RNA
- RT-qPCR – kvantitativní real time polymerázová řetězová reakce
- shRNA – short hairpin RNA, krátké vlásenkové RNA
- siRNA – small interacting RNA
- SS – systémová sklerodermie
- SjS – Sjogrenův syndrom
- SLE – systémový lupus erythematodes
- SLEDAI – index aktivity SLE (systemic lupus erythematodes disease activity index)
- SPARC – secreted protein acidic and rich in cysteine/osteonectin/BM40
- STAT – přenašeč signálu a aktivátor transkripce (signal transducer and activator )
- TCR – receptor T-lymfocytů (T-cell receptor)
- TJC – počet bolestivých kloubů (tender joint count)
- TGF (transforming growth factor)
- THP1 – (human acute monocytic leukemia cell line)
- TLR – Toll-like receptor
- TNFα – tumor nekrotizující faktor α (tumor necrosis factor α)
- TRAF – faktor asociovaný s TNFR (TNFR associating factor)
- Treg – T regulační lymfocyty
- tRNA – transferová RNA
- TTP – tristetraprolin
- UTR – nepřekládaná oblast kódující sekvence mRNA,
- VEGF – cévní endotelový růstový faktor (vascular endothelial growth factor)
- XiRNA – malé RNA podílející se na inaktivaci X chromosomu
- ZEB – Zinc finger E-box-binding homeobox
Mgr. M. Škoda
Revmatologický ústav
Na Slupi 4
128 50 Praha 2
Sources
1. Strietholt S, Maurer B, Peters MA, Pap T, Gay S. Epigenetic modifications in rheumatoid arthritis. Arthritis Res Ther 2008; 10 : 219.
2. Meda F, Folci M, Baccarelli A, Selmi C. The epigenetics of autoimmunity. Cell Mol Immunol 2011; Jan 31. (Epub ahead of print)
3. Chong S, Whitelaw E. Epigenetic germline inheritance. Curr Opin Genet Dev 2004; 14 : 692–6.
4. Brooks WH, Le Dantec C, Pers JO, Youinou P, Renaudineau Y. Epigenetics and autoimmunity. J Autoimmun 2010; 34:J207-19.
5. Collins LJ, Penny D. The RNA infrastructure: dark matter of the eukaryotic cell? Trends Genet 2009; 25 : 120–8.
6. Shrey K, Suchit A, Nishant M, Vibha R. RNA interference: emerging diagnostics and therapeutics tool. BiochemBiophys Res Commun 2009; 386 : 273–7.
7. Mattick JS. The genetic signatures of noncoding RNAs. PLoS Genet 2009; 5:e1000459.
8. Krol J, Loedige I, Filipowicz W: The widespread regulation of microRNA biogenesis, function and decay. Nat Rev Genet 2010; 11 : 597-610.
9. Guo H, Ingolia NT, Weissman JS, Bartel DP. Mammalian microRNAs predominantly act to decrease target mRNA levels. Nature 2010; 466(7308):835-40.
10. Lytle JR, Yario TA, Steitz JA. Target mRNAs are repressed as efficiently by microRNA-binding sites in the 5’ UTR as in the 3’ UTR. Proc Natl Acad Sci U S A 2007; 104(23):9667-72.
11. Eiring AM, Harb JG, Neviani P, Garton C, Oaks JJ, Spizzo R, Liu S, Schwind S, Santhanam R, Hickey CJ, Becker H, Chandler JC, Andino R, Cortes J, Hokland P, Huettner CS, Bhatia R, Roy DC, Liebhaber SA, Caligiuri MA, Marcucci G, Garzon R, Croce CM, Calin GA, Perrotti D. miR-328 functions as an RNA decoy to modulate hnRNP E2 regulation of mRNA translation in leukemic blasts. Cell 2010; 140 : 652-65.
12. Baek D, Villen J, Shin C, Camargo FD, Gygi SP, Bartel DP. The impact of microRNAs on protein output. Nature 2008; 455 : 64–71.
13. Selbach M, Schwanhausser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N. Widespread changes in protein synthesis induced by microRNAs, Nature 2008; 455 : 58–63.
14. Liang Y, Ridzon D, Wong L, Chen C. Characterization of microRNA expression profiles in normal human tissues. BMC Genomics 2007; 8 : 166.
15. Eulalio A, Huntzinger E, Nishihara T, Rehwinkel J, Fauser M, Izaurralde E. Deadenylation is a widespread effect of miRNA regulation. RNA 2009; 15 : 21–32.
16. Vasudevan S, Tong Y, Steitz JA. Switching from repression to activation: microRNAs can up-regulate translation. Science 2007; 318 : 1931-4.
17. Iorio MV, Piovan C, Croce CM. Interplay between microRNAs and the epigenetic machinery: an intricate network Biochim Biophys Acta 2010; 1799 : 694-701.
18. Tay, Y, Zhang, J., Thomson, A. M., Lim, B. Rigoutsos, I. MicroRNAs to Nanog, Oct4 and Sox2 coding regions modulate embryonic stem cell differentiation. Nature 2008; 455 : 1124–1128.
19. Xu N, Papagiannakopoulos T, Pan G, Thomson JA, Kosik KS. MicroRNA-145 regulates OCT4, SOX2, and KLF4 and represses pluripotency in human embryonic stem cells. Cell 2009; 137 : 647–658.
20. Ren J, Jin P, Wang E, Marincola FM, Stroncek DF. MicroRNA and gene expression patterns in the differentiation of human embryonic stem cells. J. Transl. Med 2009; 7 : 20.
21. Yi R, O’Carroll D, Pasolli HA, Zhang Z, Dietrich FS, Tarakhovsky A, Fuchs ENat. Morphogenesis in skin is governed by discrete sets of differentially expressed microRNAs.Genet 2006; 38 : 356–362.
22. Chen JF, Mandel EM, Thomson JM, Wu Q, Callis TE, Hammond SM, Conlon FL, Wang DZ. The role of microRNA-1 and microRNA-133 in skeletal muscle proliferation and differentiation. Nat. Genet 2006; 38 : 228–233.
23. Kloosterman WP, Lagendijk AK, Ketting RF, Moulton JD, Plasterk RH. Targeted inhibition of miRNA maturation with morpholinos reveals a role for miR-375 in pancreatic islet development. PLoS Biol 2007; 5: e203.
24. Callis TE, Chen JF, Safar M, Wang DZ. MicroRNAs in skeletal and cardiac muscle development.DNA Cell Biol 2007; 26 : 219–225.
25. van Rooij E, Sutherland LB, Qi X, Richardson JA, Hill J, Olson EN. Control of stress-dependent cardiac growth and gene expression by a microRNA. Science 2007; 316 : 575–579.
26. Cheng LC, Pastrana E, Tavazoie M, Doetsch F. MiR-124 regulates adult neurogenesis in the subventricular zone stem cell niche. Nat. Neurosci 2009; 12 : 399–408.
27. Nana-Sinkam SP, Croce CM. MicroRNA dysregulation in cancer: opportunities for the development of microRNA-based drugs. IDrugs 2010;13 : 843-6.
28. Macfarlane LA, Murphy PR. MicroRNA: Biogenesis, Function and Role in Cancer Curr Genomics 2010; 11 : 537-61.
29. Topkara VK, Mann DL. Role of MicroRNAs in Cardiac Remodeling and Heart Failure. Cardiovasc Drugs Ther 2011; 25 : 171-82.
30. Niwa R., Zhou F, Li C, Slack FJ. The expression of the Alzheimer’s amyloid precursor protein-like gene is regulated by developmental timing microRNAs and their targets in Caenorhabditis elegans. Dev. Biol 2008; 315 : 418–425.
31. Zhu Y, Kalbfleisch T, Brennan MD, Li YA. MicroRNA gene is hosted in an intron of a schizophrenia-susceptibility gene. Schizophr. Res 2009; 109 : 86–89.
32. Sarasin-Filipowicz M, Krol J, Markiewicz I, Heim MH, Filipowicz W. Decreased levels of microRNA miR-122 in individuals with hepatitis C responding poorly to interferon therapy. Nat Med 2009; 15 : 31–33.
33. Houzet L, Yeung ML, de Lame V, Desai D, Smith SM, Jeang KT. MicroRNA profile changes in human immunodeficiency virus type 1 (HIV-1) seropositive individuals. Retrovirology 2008; 5 : 118.
34. Luo X, Tsai LM, Shen N, Yu D. Evidence for microRNA-mediated regulation in rheumatic diseases. Ann Rheum Dis. 2010; 69 (Suppl 1):i30-36.
35. Škoda M, Novota P, Remáková M, Faustová M, Vencovský J. Malé molekuly RNA a jejich význam v imunitní odpovědi organismu Čes Revmatol 2011; 2 : 56-64.
36. Chen X, Ba Y, Ma L, Cai X, Yin Y, Wang K, Guo J, Zhang Y, Chen J, Guo X, Li Q, Li X, Wang W, Zhang Y, Wang J, Jiang X, Xiang Y, Xu C, Zheng P, Zhang J, Li R, Zhang H, Shang X, Gong T, Ning G, Wang J, Zen K, Zhang J, Zhang CY. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Res 2008; 10 : 997-1006.
37. Lebanony D, Benjamin H, Gilad S, Ezagouri M, Dov A, Ashkenazi K, Gefen N, Izraeli S, Rechavi G, Pass H, Nonaka D, Li J, Spector Y, Rosenfeld N, Chajut A, Cohen D, Aharonov R, Mansukhani M. Diagnostic assay based on hsa-miR-205 expression distinguishes squamous from nonsquamous non-small-cell lung carcinoma. J Clin Oncol 2009; 27 : 2030-7.
38. Benjamin H, Lebanony D, Rosenwald S, Cohen L, Gibori H, Barabash N, Ashkenazi K, Goren E, Meiri E, Morgenstern S, Perelman M, Barshack I, Goren Y, Edmonston TB, Chajut A, Aharonov R, Bentwich Z, Rosenfeld N, Cohen D. A diagnostic assay based on microRNA expression accurately identifies malignant pleural mesothelioma. J Mol Diagn 2010; 12 : 771-9.
39. Bartels CL, Tsongalis GJ. MicroRNAs: novel biomarkers for human cancer. Clin Chem 2009; 55 : 623-31.
40. Arroyo JD, Chevillet JR, Kroh EM, Ruf IK, Pritchard CC, Gibson DF, Mitchell PS, Bennett CF, Pogosova-Agadjanyan EL, Stirewalt DL, Tait JF, Tewari M. Argonaute2 complexes carry a population of circulating microRNAs independent of vesicles in human plasma. Proc Natl Acad Sci U S A. 2011; 108 : 5003-8.
41. Vickers KC, Palmisano BT, Shoucri BM, Shamburek RD, Remaley AT. MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nat Cell Biol. 2011 Apr;13(4):423-33. Epub 2011 Mar 20
42. Valadi H, Ekström K, Bossios A, Sjöstrand M, Lee JJ, Lötvall JO. Exosome-mediated transfer of mRNAs and microRNAs is a novel mechanism of genetic exchange between cells. Nat Cell Biol 2007; 9 : 654–659.
43. Kosaka N, Iguchi H, Yoshioka Y, Takeshita F, Matsuki Y, Ochiya T. Secretory mechanisms and intercellular transfer of microRNAs in living cells. J Biol Chem 2010; 285 : 17442–17452
44. Rossbach M. Small non-coding RNAs as novel therapeutics. Curr Mol Med. 2010J; 10 : 361-8.
45. Hutvágner G, Simard MJ, Mello CC, Zamore PD. Sequence-specific inhibition of small RNA function. PLoS Biol 2004; 2:E98
46. Krützfeldt J, Rajewsky N, Braich R, Rajeev KG, Tuschl T, Manoharan M, Stoffel M. Silencing of microRNAs in vivo with ‘antagomirs’. Nature 2005; 438 : 685-9.
47. Elmén J, Lindow M, Schütz S, Lawrence M, Petri A, Obad S, Lindholm M, Hedtjärn M, Hansen HF, Berger U, Gullans S, Kearney P, Sarnow P, Straarup EM, Kauppinen S. LNA-mediated microRNA silencing in non-human primates. Nature 2008; 452 : 896-9.
48. Lanford RE, Hildebrandt-Eriksen ES, Petri A, Persson R, Lindow M, Munk ME, Kauppinen S, Įrum H. Therapeutic silencing of microRNA-122 in primates with chronic hepatitis C virus infection. Science 2010; 327 : 198-201.
49. Grimm D, Streetz KL, Jopling CL, Storm TA, Pandey K, Davis CR, Marion P, Salazar F, Kay MA. Fatality in mice due to oversaturation of cellular microRNA/short hairpin RNA pathways. Nature 2006; 441 : 537-41.
50. Lecellier CH, Dunoyer P, Arar K, Lehmann-Che J, Eyquem S, Himber C, Saēb A,Voinnet O. A cellular microRNA mediates antiviral defense in human cells. Science 2005; 308 : 557–560
51. Pedersen IM, Cheng G, Wieland S, Volinia S, Croce CM, Chisari FV, David M. Interferon modulation of cellular microRNAs as an antiviral mechanism. Nature 2007; 449 : 919–922.
52. Stern-Ginossar N, Elefant N, Zimmermann A, Wolf DG, Saleh N, Biton M, Horwitz E, Prokocimer Z, Prichard M, Hahn G, Goldman-Wohl D, Greenfield C, Yagel S, Hengel H, Altuvia Y, Margalit H, Mandelboim O. Host immune system gene targeting by a viral miRNA, Science 2007; 317 : 376-81.
53. Gottwein E, Mukherjee N, Sachse C, Frenzel C, Majoros WH, Chi JT, Braich R, Manoharan M, Soutschek J, Ohler U, Cullen BR. A viral microRNA functions as an orthologue of cellular miR-155. Nature 2007; 450 : 1096-9.
54. Motsch N, Pfuhl T, Mrazek J, Barth S, Grässer FA. Epstein-Barr virus-encoded latent membrane protein 1 (LMP1) induces the expression of the cellular microRNA miR-146a. RNA Biol 2007; 4 : 131-7.
55. Gatto G, Rossi A, Rossi D, Kroening S, Bonatti S, Mallardo M. Epstein-Barr virus latent membrane protein 1 trans-activates miR-155 transcription through the NF-kappaB pathway. Nucleic Acids Res 2008; 36 : 6608-19.
56. Cameron JE, Yin Q, Fewell C, Lacey M, McBride J, Wang X, Lin Z, Schaefer BC, Flemington EK. Epstein-Barr virus latent membrane protein 1 induces cellular MicroRNA miR-146a, a modulator of lymphocyte signaling pathways. J Virol 2008; 82 : 1946-58.
57. Tang Y, Luo X, Cui H, Ni X, Yuan M, Guo Y, Huang X, Zhou H, de Vries N, Tak PP, Chen S, Shen N. MicroRNA-146A contributes to abnormal activation of the type I interferon pathway in human lupus by targeting the key signaling proteins. Arthritis Rheum. 2009; 60 : 1065-75.
58. Hai-yan W, Yang L, Mei-hong C, Hui Z. Expression of MicroRNA-146a in peripheral blood mononuclear cells in patients with systemic lupus Erythematosus. Zhongguo Yi Xue Ke Xue Yuan Xue Bao 2011; 33 : 185-8 (abstract).
59. Pauley KM, Satoh M, Chan AL, Bubb MR, Reeves WH, Chan EK. Upregulated miR-146a expression in peripheral blood mononuclear cells from rheumatoid arthritis patients. Arthritis Res Ther 2008; 10: R101.
60. Stanczyk J, Pedrioli DM, Brentano F, Sanchez-Pernaute O, Kolling C, Gay RE, Detmar M, Gay S, Kyburz D. Altered expression of MicroRNA in synovial fibroblasts and synovial tissue in rheumatoid arthritis. Arthritis Rheum 2008; 58 : 1001-9.
61. Nakasa T, Miyaki S, Okubo A, Hashimoto M, Nishida K, Ochi M, Asahara H. Expression of microRNA-146 in rheumatoid arthritis synovial tissue. Arthritis Rheum 2008; 58 : 1284-92.
62. Li J, Wan Y, Guo Q, Zou L, Zhang J, Fang Y, Zhang J, Zhang J, Fu X, Liu H, Lu L, Wu Y. Altered microRNA expression profile with miR-146a upregulation in CD4+ T cells from patients with rheumatoid arthritis. Arthritis Res Ther 2010; 12: R81.
63. Murata K, Yoshitomi H, Tanida S, Ishikawa M, Nishitani K, Ito H, Nakamura T. Plasma and synovial fluid microRNAs as potential biomarkers of rheumatoid arthritis and osteoarthritis. Arthritis Res Ther 2010;12: R86.
64. Fulci V, Scappucci G, Sebastiani GD, Giannitti C, Franceschini D, Meloni F, Colombo T, Citarella F, Barnaba V, Minisola G, Galeazzi M, Macino G. miR-223 is overexpressed in T-lymphocytes of patients affected by rheumatoid arthritis, Hum. Immunol 2010; 71 : 206–211.
65. Stanczyk J, Ospelt C, Karouzakis E, Filer A, Raza K, Kolling C, Gay R, Buckley CD, Tak PP, Gay S, Kyburz D. Altered expression of miR-203 in rheumatoid arthritis synovial fibroblasts and its role in fibroblast activation. Arthritis Rheum 2011; 63 : 373-81.
66. Nakamachi Y, Kawano S, Takenokuchi M, Nishimura K, Sakai Y, Chin T, Saura R, Kurosaka M, Kumagai S. MicroRNA-124a is a key regulator of proliferation and monocyte chemoattractant protein 1 secretion in fibroblast-like synoviocytes from patients with rheumatoid arthritis. Arthritis Rheum 2009; 60 : 1294-304.
67. Kawano S, Nakamachi Y. miR-124a as a key regulator of proliferation and MCP-1 secretion in synoviocytes from patients with rheumatoid arthritis. Ann Rheum Dis 2011; 70 Suppl 1:i88-91
68. Niimoto T, Nakasa T, Ishikawa M, Okuhara A, Izumi B, Deie M, Suzuki O, Adachi N, Ochi M. MicroRNA-146a expresses in interleukin-17 producing T cells in rheumatoid arthritis patients. BMC Musculoskelet Disord 2010; 11 : 209.
69. Alsaleh G, Suffert G, Semaan N, Juncker T, Frenzel L, Gottenberg JE, Sibilia J, Pfeffer S, Wachsmann D. Bruton’s tyrosine kinase is involved in miR-346-related regulation of IL-18 release by lipopolysaccharide-activated rheumatoid fibroblast-like synoviocytes. J Immunol 2009; 182 : 5088-97.
70. Abou-Zeid A, Saad M, Soliman E. MicroRNA 146a Expression in Rheumatoid Arthritis: Association with Tumor Necrosis Factor-Alpha and Disease Activity. Genet Test Mol Biomarkers 2011; (Epub ahead of print)
71. Feng ZT, Li J, Ren J, Lv Z. Expression of miR-146a and miR-16 in peripheral blood mononuclear cells of patients with rheumatoid arthritis and their correlation to the disease activity. Nan Fang Yi Ke Da Xue Xue Bao. 2011; 31 : 320-3. (abstract)
72. Kurowska-Stolarska M, Alivernini S, Ballantine LE, Asquith DL, Millar NL, Gilchrist DS, Reilly J, Ierna M, Fraser AR, Stolarski B, McSharry C, Hueber AJ, Baxter D, Hunter J, Gay S, Liew FY, McInnes IB. MicroRNA-155 as a proinflammatory regulator in clinical and experimental arthritis. Proc Natl Acad Sci U S A 2011; 108 : 11193-8.
73. Semaan N, Frenzel L, Alsaleh G, Suffert G, Gottenberg JE, Sibilia J, Pfeffer S, Wachsmann D. miR-346 controls release of TNF-α protein and stability of its mRNA in rheumatoid arthritis via tristetraprolin stabilization. PLoS One 2011; 6: e19827.
74. Nagata Y, Nakasa T, Mochizuki Y, Ishikawa M, Miyaki S, Shibuya H, Yamasaki K, Adachi N, Asahara H, Ochi M. Induction of apoptosis in the synovium of mice with autoantibody-mediated arthritis by the intraarticular injection of double-stranded MicroRNA-15a. Arthritis Rheum 2009; 60 : 2677-83.
75. Nakasa T, Shibuya H, Nagata Y, Niimoto T, Ochi M. The inhibitory effect of microRNA-146a expression on bone destruction in collagen-induced arthritis. Arthritis Rheum 2011; 63 : 1582-90.
76. Yue J, Tigyi G. Conservation of miR-15a/16-1 and miR-15b/16-2 clusters. Mamm Genome 2010; 21 : 88–94.
77. Linsley PS, Schelter J, Burchard J, Kibukawa M, Martin MM, Bartz SR, Johnson JM, Cummins JM, Raymond CK, Dai H, Chau N, Cleary M, Jackson AL, Carleton M, Lim L. Transcripts targeted by the microRNA-16 family cooperatively regulate cell cycle progression. Mol Cell Biol 2007; 27 : 2240-52.
78. Liu Q, Fu H, Sun F, Zhang H, Tie Y, Zhu J, Xing R, Sun Z, Zheng X. miR-16 family induces cell cycle arrest by regulating multiple cell cycle genes. Nucleic Acids Res 2008; 36 : 5391-404.
79. Bandi N, Zbinden S, Gugger M, Arnold M, Kocher V, Hasan L, Kappeler A, Brunner T, Vassella E. miR-15a and miR-16 are implicated in cell cycle regulation in a Rb-dependent manner and are frequently deleted or down-regulated in non-small cell lung cancer. Cancer Res 2009; 69 : 5553–5559.
80. Cimmino A, Calin GA, Fabbri M, Iorio MV, Ferracin M, Shimizu M, Wojcik SE, Aqeilan RI, Zupo S, Dono M, Rassenti L, Alder H, Volinia S, Liu CG, Kipps TJ, Negrini M, Croce CM. miR-15 and miR-16 induce apoptosis by targeting BCL2. Proc Natl Acad Sci USA 2005; 102 : 13944–13949.
81. Bonci D, Coppola V, Musumeci M, Addario A, Giuffrida R, Memeo L, D’Urso L, Pagliuca A, Biffoni M, Labbaye C, Bartucci M, Muto G, Peschle C, De Maria R. The miR-15a-miR-16–1 cluster controls prostate cancer by targeting multiple oncogenic activities. Nat Med 2008; 14 : 1271–1277.
82. Calin GA, Cimmino A, Fabbri M, Ferracin M, Wojcik SE, Shimizu M, Taccioli C, Zanesi N, Garzon R, Aqeilan RI, Alder H, Volinia S, Rassenti L, Liu X, Liu CG, Kipps TJ, Negrini M, Croce CM. MiR-15a and miR-16–1 cluster functions in human leukemia. Proc Natl Acad Sci U S A 2008; 105 : 5166–5171.
83. Jing Q, Huang S, Guth S, Zarubin T, Motoyama A, Chen J, Di Padova F, Lin SC, Gram H, Han J. Involvement of microRNA in AU-rich elementmediated mRNA instability. Cell 2005; 120 : 623–34.
84. Kloosterman WP, Plasterk RH. The diverse functions of microRNAs in animal development and disease. Dev Cell 2006; 11 : 441 – 50 .
85. Baroukh NN, Van Obberghen E. Function of microRNA-375 and microRNA-124a in pancreas and brain. Febs J 2009; 276 : 6509–21.
86. Fowler A, Thomson D, Giles K, Maleki S, Mreich E, Wheeler H, Leedman P, Biggs M, Cook R, Little N, Robinson B, McDonald K. miR-124a is frequently down-regulated in glioblastoma and is involved in migration and invasion. Eur J Cancer 2011; 47 : 953-63.
87. Pierson J, Hostager B, Fan R, Vibhakar R. Regulation of cyclin dependent kinase 6 by microRNA 124 in medulloblastoma. J Neurooncol 2008; 90 : 1-7.
88. Duroux-Richard I, Presumey J, Courties G, Gay S, Gordeladze J, Jorgensen C, Kyburz D, Apparailly F. MicroRNAs as new player in rheumatoid arthritis. Joint Bone Spine 2011; 78 : 17-22.
89. Lindenblatt C, Schulze-Osthoff K, Totzke G. IkappaBzeta expression is regulated by miR-124a. Cell Cycle 2009; 8 : 2019-23.
90. Soreq H, Wolf Y. NeurimmiRs: microRNAs in the neuroimmune interface. Trends Mol Med. 2011; 17 : 548-55.
91. Lagos D, Pollara G, Henderson S, Gratrix F, Fabani M, Milne RS, Gotch F, Boshoff C. miR-132 regulates antiviral innate immunity through suppression of the p300 transcriptional co-activator. Nat Cell Biol 2010; 12 : 513-9.
92. Shaked I, Meerson A, Wolf Y, Avni R, Greenberg D, Gilboa-Geffen A, Soreq H. MicroRNA-132 potentiates cholinergic anti-inflammatory signaling by targeting acetylcholinesterase. Immunity 2009; 31 : 965–973.
93. Duroux-Richard I, Jorgensen C, Apparailly F. miRNAs and rheumatoid arthritis - promising novel biomarkers. Swiss Med Wkly 2011; 141: w13175.
94. Taganov KD, Boldin MP, Chang KJ, Baltimore D. NF-kB-dependent induction of microRNA miR 146, an inhibitor targeted to signaling proteins of innate immune responses. Proc. Natl Acad. Sci. USA 2006; 103 : 12481–12486.
95. Bhaumik D, Scott GK, Schokrpur S, Patil CK, Orjalo AV, Rodier F, Lithgow GJ, Campisi J. MicroRNAs miR 146a/b negatively modulate the senescence-associated inflammatory mediators IL-6 and IL-8. Aging (Albany NY) 2009; 1 : 402-11.
96. Hou J, Wang P, Lin L, Liu X, Ma F, An H, Wang Z, Cao X. MicroRNA-146a feedback inhibits RIGI-dependent Type I IFN production in macrophages by targeting TRAF6, IRAK1, and IRAK2. type I. J Immunol 2009; 183 : 2150–8.
97. Nahid MA, Pauley KM, Satoh M, Chan EK. miR 146a is critical for endotoxin-induced tolerance: implication in innate immunity. J. Biol. Chem 2009; 284 : 34590–34599.
98. Nahid MA, Satoh M, Chan EK. Mechanistic role of microRNA-146a in endotoxin-induced differential cross-regulation of TLR signaling. J. Immunol 2011; 186 : 1723–1734.
99. Lu LF, Boldin MP, Chaudhry A, Lin LL, Taganov KD, Hanada T, Yoshimura A, Baltimore D, Rudensky AY: Function of miR-146a in controlling Treg cellmediated regulation of Th1 responses. Cell 2010; 142 : 914-929.
100. Kuang W, Tan J, Duan Y et al. Cyclic stretch induced miR-146a upregulation delays C2C12 myogenic differentiation through inhibition of Numb. Biochem Biophys Res Commun 2009; 378 : 259–63.
101. He H, Jazdzewski K, Li W et al. The role of microRNA genes in papillary thyroid carcinoma. Proc Natl Acad Sci U S A 2005; 102 : 19075–80.
102. Jazdzewski K, Murray EL, Franssila K, Jarzab B, Schoenberg DR, de la Chapelle A. Common SNP in pre-miR-146a decreases mature miR expression and predisposes to papillary thyroid carcinoma. Proc Natl Acad Sci U S A 2008; 105 : 7269–74.
103. Zhang H, Luo XQ, Zhang P et al. MicroRNA patterns associated with clinical prognostic parameters and CNS relapse prediction in pediatric acute leukemia. PLoS ONE 2009; 4: e7826.
104. Young LS, Murray PG. Epstein-Barr virus and oncogenesis: from latent genes to tumours. Oncogene 2003; 22 : 5108–21.
105. Hurst DR, Edmonds MD, Scott GK, Benz CC, Vaidya KS, Welch DR. Breast cancer metastasis suppressor 1 up-regulates miR-146, which suppresses breast cancer metastasis. Cancer Res 2009; 69 : 1279-83.
106. Ma X, Becker Buscaglia LE, Barker JR, Li Y. MicroRNAs in NF-{kappa}B signaling. J Mol Cell Biol 2011 Jun; 3 : 159-66.
107. Wang G, Tam LS, Li EK, Kwan BC, Chow KM, Luk CC, Li PK, Szeto CC. Serum and urinary cell-free MiR-146a and MiR-155 in patients with systemic lupus erythematosus. J Rheumatol 2010; 37 : 2516-22.
108. Pauley KM, Stewart CM, Gauna AE et al. Altered miR 146a expression in Sjogren’s syndrome and its functional role in innate immunity. Eur J Immunol 2011; 41 : 2029-39.
109. Sonkoly E, StĆhle M, Pivarcsi A. MicroRNAs: novel regulators in skin inflammation. Clin Exp Dermatol 2008 May; 33 : 312-5.
110. Chatzikyriakidou A, Voulgari PV, Georgiou I, Drosos AA. The role of microRNA-146a (miR-146a) and its target IL-1R-associated kinase (IRAK1) in psoriatic arthritis susceptibility. Scand J Immunol 2010; 71 : 382-5.
111. Chatzikyriakidou A, Voulgari PV, Georgiou I, Drosos AA. A polymorphism in the 3’-UTR of interleukin-1 receptor-associated kinase (IRAK1), a target gene of miR-146a, is associated with rheumatoid arthritis susceptibility. Joint Bone Spine 2010; 77 : 411-3.
112. van den Berg A, Kroesen BJ, Kooistra K, de Jong D, Briggs J, Blokzijl T, et al. High expression of B-cell receptor inducible gene BIC in all subtypes of Hodgkin lymphoma. Genes Chromosomes Cancer 2003; 37 : 20–8.
113. Kluiver J, Poppema S, de Jong D, Blokzijl T, Harms G, Jacobs S, et al. BIC and miR - 155 are highly expressed in Hodgkin, primary mediastinal and diffuse large B cell lymphomas. J Pathol 2005; 207 : 243–9.
114. Metzler M, Wilda M, Busch K, Viehmann S, Borkhardt A. High expression of precursor microRNA-155/BIC RNA in children with Burkitt lymphoma. Genes Chromosomes Cancer 2004; 39 : 167–9.
115. Yanaihara N, Caplen N, Bowman E, Seike M, Kumamoto K, Yi M, Stephens RM, Okamoto A, Yokota J, Tanaka T, Calin GA, Liu CG, Croce CM, Harris CC. Unique microRNA molecular profiles in lung cancer diagnosis and prognosis. Cancer Cell 2006; 9 : 189-98.
116. Iorio MV, Ferracin M, Liu CG, Veronese A, Spizzo R, Sabbioni S, Magri E, Pedriali M, Fabbri M, Campiglio M, Ménard S, Palazzo JP, Rosenberg A, Musiani P, Volinia S, Nenci I, Calin GA, Querzoli P, Negrini M, Croce CM. MicroRNA gene expression deregulation in human breast cancer. Cancer Res 2005; 65 : 7065-70.
117. Greither T, Grochola LF, Udelnow A, Lautenschläger C, Würl P, Taubert H. Elevated expression of microRNAs 155, 203, 210 and 222 in pancreatic tumors is associated with poorer survival. Int J Cancer 2010; 126 : 73-80.
118. Wang G, Tam LS, Li EK, Kwan BC, Chow KM, Luk CC, Li PK, Szeto CC. Serum and urinary cell-free MiR-146a and MiR-155 in patients with systemic lupus erythematosus. J Rheumatol 2010; 37 : 2516-22.
119. Alevizos I, Alexander S, Turner RJ, Illei GG.: MicroRNA expression profiles as biomarkers of minor salivary gland inflammation and dysfunction in Sjögren’s syndrome. Arthritis Rheum 2011; 63 : 535-44.
120. Pauley KM, Stewart CM, Gauna AE et al. Altered miR 146a expression in Sjogren’s syndrome and its functional role in innate immunity. Eur J Immunol 2011; 41 : 2029-39.
121. Eisenberg I, Eran A, Nishino I et al. Distinctive patterns of microRNA expression in primary muscular disorders. Proc Natl Acad Sci USA 2007; 104 : 17016–21.
122. Leng RX, Pan HF, Qin WZ, Chen GM, Ye DQ. Role of microRNA-155 in autoimmunity. Cytokine Growth Factor Rev 2011. (Epub ahead of print)
123. Rodriguez A, Vigorito E, Clare S, Warren MV, Couttet P, Soond DR, van Dongen S, Grocock RJ, Das PP, Miska EA, Vetrie D, Okkenhaug K, Enright AJ, Dougan G, Turner M, Bradley A. Requirement of bic/microRNA-155 for normal immune function. Science 2007; 316 : 608-11.
124. Thai TH, Calado DP, Casola S, Ansel KM, Xiao C, Xue Y, Murphy A, Frendewey D,Valenzuela D, Kutok JL, Schmidt-Supprian M, Rajewsky N, Yancopoulos G, Rao A, Rajewsky K. Regulation of the germinal center response by microRNA-155. Science 2007; 316 : 604-8.
125. Vigorito E, Perks KL, Abreu-Goodger C, Bunting S, Xiang Z, Kohlhaas S, Das PP, Miska EA, Rodriguez A, Bradley A, Smith KG, Rada C, Enright AJ, Toellner KM, Maclennan IC, Turner M. microRNA-155 regulates the generation of immunoglobulin class-switched plasma cells. Immunity 2007; 27 : 847-59.
126. O’Connell RM, Kahn D, Gibson WS, Round JL, Scholz RL, Chaudhuri AA, et al. MicroRNA-155 promotes autoimmune inflammation by enhancing inflammatory T cell development. Immunity 2010; 33 : 607–19.
127. Blüml S, Bonelli M, Niederreiter B, Puchner A, Mayr G, Hayer S, Koenders MI, van den Berg WB, Smolen J, Redlich K. Essential role for micro-RNA 155 in the pathogenesis of autoimmune arthritis. Arthritis Rheum. 2011; 63 : 1281-8.
128. Lu LF, Thai TH, Calado DP, Chaudhry A, Kubo M, Tanaka K, Loeb GB, Lee H, Yoshimura A, Rajewsky K, Rudensky AY. Foxp3 - dependent microRNA155 confers competitive fitness to regulatory T cells by targeting SOCS1 protein. Immunity 2009; 30 : 80–91.
129. Bayer AL, Yu A, Malek TR. Function of the IL-2R for thymic and peripheral CD4+CD25+ Foxp3+ T regulatory cells. J Immunol 2007; 178 : 4062–71.
130. Ceppi M, Pereira PM, Dunand-Sauthier I, Barras E, Reith W, Santos MA, Pierre P. MicroRNA-155 modulates the interleukin-1 signaling pathway in activated human monocyte-derived dendritic cells. Proc Natl Acad Sci USA 2009;106 : 2735–40.
131. Lu C, Huang X, Zhang X, Roensch K, Cao Q, Nakayama KI, Blazar BR, Zeng Y, Zhou X. MiR-221 and miR-155 regulate human dendritic cell development, apoptosis and IL-12 production through targeting of p27kip1, KPC1 and SOCS-1. Blood 2011; 117 : 4293-303.
132. Sonkoly E, Wei T, Janson PC, Sääf A, Lundeberg L, Tengvall-Linder M, Norstedt G, Alenius H, Homey B, Scheynius A, StĆhle M, Pivarcsi A. MicroRNAs: novel regulators involved in the pathogenesis of psoriasis? PLoS One 2007; 2: e610
133. ViticchiŹ G, Lena AM, Latina A, Formosa A, Gregersen LH, Lund AH, Bernardini S, Mauriello A, Miano R, Spagnoli LG, Knight RA, Candi E, Melino G. MiR-203 controls proliferation, migration and invasive potential of prostate cancer cell lines. Cell Cycle 2011; 10 : 1121-31.
134. Saini S, Majid S, Yamamura S, Tabatabai L, Suh SO, Shahryari V, Chen Y, Deng G, Tanaka Y, Dahiya R. Regulatory Role of mir-203 in Prostate Cancer Progression and Metastasis. Clin Cancer Res 2011; 17 : 5287-98.
135. Yuan Y, Zeng ZY, Liu XH, Gong DJ, Tao J, Cheng HZ, Huang SD. MicroRNA-203 inhibits cell proliferation by repressing ∆Np63 expression in human esophageal squamous cell carcinoma. BMC Cancer 2011; 11 : 57.
136. Ikenaga N, Ohuchida K, Mizumoto K, Yu J, Kayashima T, Sakai H, Fujita H, Nakata K, Tanaka M. MicroRNA-203 expression as a new prognostic marker of pancreatic adenocarcinoma. Ann Surg Oncol 2010; 17 : 3120-8.
137. Furuta M, Kozaki KI, Tanaka S, Arii S, Imoto I, Inazawa J. miR-124 and miR-203 are epigenetically silenced tumor-suppressive microRNAs in hepatocellular carcinoma. Carcinogenesis 2010; 31 : 766-76.
138. Chiang Y, Song Y, Wang Z, Chen Y, Yue Z, Xu H, Xing C, Liu Z. Aberrant expression of miR-203 and its clinical significance in gastric and colorectal cancers. J Gastrointest Surg 2011; 15 : 63-70.
139. Vasilatou D, Papageorgiou S, Pappa V, Papageorgiou E, Dervenoulas J. The role of microRNAs in normal and malignant hematopoiesis Eur J Haematol 2010; 84 : 1-16.
140. Eyholzer M, Schmid S, Schardt JA, Haefliger S, Mueller BU, Pabst T. Complexity of miR-223 regulation by CEBPA in human AML. Leuk Res 2010; 34 : 672-6.
141. Li S, Li Z, Guo F, Qin X, Liu B, Lei Z, Song Z, Sun L, Zhang HT, You J, Zhou Q. miR-223 regulates migration and invasion by targeting Artemin in human esophageal carcinoma. J Biomed Sci 2011; 18 : 24.
142. Wang JF, Yu ML, Yu G, Bian JJ, Deng XM, Wan XJ, Zhu KM. Serum miR-146a and miR-223 as potential new biomarkers for sepsis. Biochem Biophys Res Commun 2010; 394 : 184-8.
143. Dai Y, Huang YS, Tang M, Lv TY, Hu CX, Tan YH, Xu ZM, Yin YB. Microarray analysis of microRNA expression in peripheral blood cells of systemic lupus erythematosus patients. Lupus 2007; 16 : 939-46.
144. Dai Y, Sui W, Lan H, Yan Q, Huang H, Huang Y. Comprehensive analysis of microRNA expression patterns in renal biopsies of lupus nephritis patients. Rheumatol Int 2009; 29 : 749-54.
145. Te JL, Dozmorov IM, Guthridge JM, Nguyen KL, Cavett JW, Kelly JA, Bruner GR, Harley JB, Ojwang JO. Identification of unique microRNA signature associated with lupus nephritis. PLoS One 2010; 5: e10344.
146. Wang G, Tam L, Li EK, Kwan BC, Chow K, Luk CC, Li PK, Szeto CC. Serum and urinary free microRNA level in patients with systemic lupus erythematosus. Lupus 2011; 20 : 493-500.
147. Zhao S, Wang Y, Liang Y, Zhao M, Long H, Ding S, Yin H, Lu Q. MicroRNA-126 regulates DNA methylation in CD4(+) T cells and contributes to systemic lupus erythematosus by targeting DNA methyltransferase 1. Arthritis Rheum 2011; 63 : 1376-86.
148. Pan W, Zhu S, Yuan M, Cui H, Wang L, Luo X, et al. MicroRNA-21 and microRNA-148a contribute to DNA hypomethylation in lupus CD4+ T cells by directly and indirectly targeting DNA methyltransferase 1. J Immunol 2010; 184 : 6773-81
149. Zhao X, Tang Y, Qu B, Cui H, Wang S, Wang L, Luo X, Huang X, Li J, Chen S, Shen N. MicroRNA-125a contributes to elevated inflammatory chemokine RANTES levels via targeting KLF13 in systemic lupus erythematosus. Arthritis Rheum 2010; 62 : 3425-35.
150. Stagakis E, Bertsias G, Verginis P, Nakou M, Hatziapostolou M, Kritikos H, Iliopoulos D, Boumpas DT. Identification of novel microRNA signatures linked to human lupus disease activity and pathogenesis: miR-21 regulates aberrant T cell responses through regulation of PDCD4 expression. Ann Rheum Dis 2011; 70 : 1496-506.
151. Liu, M. F., Jiang, S., Lu, Z., et al. (2010a). Physiological and pathological functions of mammalian microRNAs. In: McQueen, C. A. (ed). Comprehensive Toxicology, 2nd edn. London: Elsevier Science; 427–446.
152. Fulci V, Chiaretti S, Goldoni M, Azzalin G, Carucci N, Tavolaro S, Castellano L, Magrelli A, Citarella F, Messina M, Maggio R, Peragine N, Santangelo S, Mauro FR, Landgraf P, Tuschl T, Weir DB, Chien M, Russo JJ, Ju J, Sheridan R, Sander C, Zavolan M, Guarini A, Foą R, Macino G. Quantitative technologies establish a novel microRNA profile of chronic lymphocytic leukemia. Blood 2007; 109 : 4944–4951.
153. Lawrie CH, Soneji S, Marafioti T, Cooper CD, Palazzo S, Paterson JC, Cattan H, Enver T, Mager R, Boultwood J, Wainscoat JS, Hatton CS. MicroRNA expression distinguishes between germinal center B cell-like and activated B cell-like subtypes of diffuse large B cell lymphoma. Int. J. Cancer 2007; 121 : 1156–1161.
154. Jongen-Lavrencic M, Sun SM, Dijkstra MK, Valk PJ, Löwenberg B. MicroRNA expression profiling in relation to the genetic heterogeneity of acute myeloid leukemia. Blood 2008; 111 : 5078–5085.
155. Navarro A, Gaya A, Martinez A, Urbano-Ispizua A, Pons A, Balagué O, Gel B, Abrisqueta P, Lopez-Guillermo A, Artells R, Montserrat E, Monzo M. MicroRNA expression profiling in classic Hodgkin lymphoma. Blood 2008; 111 : 2825–2832.
156. Lu TX, Munitz A, Rothenberg ME. MicroRNA-21 is up-regulated in allergic airway inflammation and regulates IL-12p35 expression. J. Immunol 2009; 182 : 4994.
157. Sheedy FJ, Palsson-McDermott E, Hennessy EJ, Martin C, O’Leary JJ, Ruan Q, Johnson DS, Chen Y, O’Neill LA. Negative regulation of TLR4 via targeting of the proinflammatory tumor suppressor PDCD4 by the microRNA miR-21. Nat. Immunol 2010;11 : 141-147.
158. Asangani IA, Rasheed SA, Nikolova DA, Leupold JH, Colburn NH, Post S, Allgayer H. MicroRNA-21 (miR-21) post-transcriptionally downregulates tumor suppressor Pdcd4 and stimulates invasion, intravasation and metastasis in colorectal cancer. Oncogene 2008; 27 : 2128-2136.
159. Frankel LB, Christoffersen NR, Jacobsen A, Lindow M, Krogh A, Lund AH. Programmed cell death 4 (PDCD4) is an important functional target of the microRNA miR-21 in breast cancer cells. J Biol Chem 2008; 283 : 1026-103.
160. Lankat-Buttgereit B, Goke R. The tumour suppressor Pdcd4: recent advances in the elucidation of function and regulation. Biol Cell 2009; 101 : 309-317.
161. Recchiuti A, Krishnamoorthy S, Fredman G, Chiang N, Serhan CN. MicroRNAs in resolution of acute inflammation: identification of novel resolvin D1-miRNA circuits. FASEB J 2011; 25 : 544-60.
162. Iliopoulos D, Jaeger SA, Hirsch HA, Bulyk ML, Struhl K. STAT3 activation of miR-21 and miR-181b-1 via PTEN and CYLD are part of the epigenetic switch linking inflammation to cancer. Mol. Cell 2010; 39 : 493–506.
163. Rouas R, Fayyad-Kazan H, El Zein N, Lewalle P, Rothé F, Simion A, Akl H, Mourtada M, El Rifai M, Burny A, Romero P, Martiat P, Badran B. Human natural Treg microRNA signature: role of microRNA-31 and microRNA-21 in FOXP3 expression. Eur J Immunol 2009; 39 : 1608–18
164. Garchow BG, Encinas OB, Leung YT, Tsao PY, Eisenberg RA, Caricchio R, Obad S, Petri A, Kauppinen S, Kiriakidou M. Silencing of microRNA-21 in vivo ameliorates autoimmune splenomegaly in lupus mice. EMBO Mol Med 2011; 3 : 605-15.
165. Mattie MD, Benz CC, Bowers J, Sensinger K, Wong L, Scott GK, Fedele V, Ginzinger D, Getts R, Haqq C. Optimized high-throughput microRNA expression profiling provides novel biomarker assessment of clinical prostate and breast cancer biopsies. Mol Cancer 2006;19 : 5-24.
166. Scott GK, Goga A, Bhaumik D, Berger CE, Sullivan CS, Benz CC. Coordinate suppression of ERBB2 and ERBB3 by enforced expression of micro-RNA miR-125a or miR-125b. J Biol Chem 2007; 282 : 1479-86.
167. Jiang L, Zhang Q, Chang H, Qiu X, Wang E., miR-125a-5p Enhances Invasion in Non-small Cell Lung Carcinoma Cell Lines by Upregulating Rock-1 Zhongguo Fei Ai Za Zhi 2009;12 : 1069-73. (abstract)
168. Cortez MA, Nicoloso MS, Shimizu M, Rossi S, Gopisetty G, Molina JR, Carlotti C Jr, Tirapelli D, Neder L, Brassesco MS, Scrideli CA, Tone LG, Georgescu MM, Zhang W, Puduvalli V, Calin GA. miR-29b and miR-125a regulate podoplanin and suppress invasion in glioblastoma. Genes Chromosomes Cancer 2010; 49 : 981-90
169. Nishida N, Mimori K, Fabbri M, Yokobori T, Sudo T, Tanaka F, Shibata K, Ishii H, Doki Y, Mori M. MicroRNA-125a-5p is an independent prognostic factor in gastric cancer and inhibits the proliferation of human gastric cancer cells in combination with trastuzumab. Clin Cancer Res 2011; 17 : 2725-33.
170. Guo S, Lu J, Schlanger R, Zhang H, Wang JY, Fox MC, Purton LE, Fleming HH, Cobb B, Merkenschlager M, Golub TR, Scadden DT. MicroRNA miR-125a controls hematopoietic stem cell number. Proc Natl Acad Sci U S A 2010; 107 : 14229-34.
171. Fernández-Hernando C, Suárez Y, Rayner KJ, Moore KJ. MicroRNAs in lipid metabolism. Curr Opin Lipidol 2011; 22 : 86-92.
172. Song A, Chen YF, Thamatrakoln K, Storm TA, Krensky AM.RFLAT-1: a new zinc finger transcription factor that activates RANTES gene expression in T lymphocytes. Immunity 1999; 10 : 93–103.
173. Fish JE, Srivastava D. MicroRNAs: opening a new vein in angiogenesis research. Sci Signal 2009; 2: pe1.
174. Meister J, Schmidt MH. miR-126 and miR-126*: new players in cancer. ScientificWorldJournal 2010; 10 : 2090-100.
175. Torres A, Askari AD, Malemud CJ. Cardiovascular disease complications in systemic lupus erythematosus. Biomark Med 2009; 3 : 239-52.
176. Bagli E, Xagorari A, Papetropoulos A, Murphy C, Fotsis T. Angiogenesis in inflammation. Autoimmun Rev 2004; 3(Suppl1):s26.
177. Colombo BM, Cacciapaglia F, Puntoni M, Murdaca G, Rossi E, Rodriguez G, et al. Traditional and non-traditional risk factors in accelerated atherosclerosis in SLE: role of VEGF (VEGATS study). Autoimmun Rev 2009; 8 : 309–15.
178. Caravalho JF, Blank M, Shoenfeld Y. VEGF in autoimmune disease. J Clin immune 2007; 27(3): 246–56.
179. Robak E, Woźniacka A, Sysa-Jedrzejowska A, Stepień H, Robak T. Serum levels of angiogenic cytokines in systemic lupus erythematosus and their correlation with disease activity. Eur Cytokine Netw 2001; 12 : 445–52.
180. Kuryliszyn-Moskal A, Klimiuk PA, Sierakowski S, Ciołkiewicz M. Vascular endothelial growth factor in systemic lupus erythematosus: relationship to disease activity, systemic organ manifestation, and nailfold capillaroscopic abnormalities. Arch Immunol Ther Exp (Warsz) 2007; 55(3):179-85.
181. Basu A, Hoerning A, Datta D, Edelbauer M, Stack MP, Calzadilla K, Pal S, Briscoe DM. Cutting edge: Vascular endothelial growth factor-mediated signaling in human CD45RO+ CD4+ T cells promotes Akt and ERK activation and costimulates IFN-gamma production. J Immunol 2010; 184 : 545-9.
182. Lv HZ, Lin T, Zhu XY, Zhang JT, Lu J A study on relationship between single nucleotide polymorphisms of vascular endothelial growth factor gene and susceptibility to systemic lupus erythematosus in China north Han population Xi Bao Yu Fen Zi Mian Yi Xue Za Zhi 2010; 26 : 1189-92. (abstract)
183. International Consortium for Systemic Lupus Erythematosus Genetics (SLEGEN), Harley JB, Alarcón-Riquelme ME, Criswell LA, Jacob CO, Kimberly RP, Moser KL, Tsao BP, Vyse TJ, Langefeld CD, Nath SK, Guthridge JM, Cobb BL, Mirel DB, Marion MC, Williams AH, Divers J, Wang W, Frank SG, Namjou B, Gabriel SB, Lee AT, Gregersen PK, Behrens TW, Taylor KE, Fernando M, Zidovetzki R, Gaffney PM, Edberg JC, Rioux JD, Ojwang JO, James JA, Merrill JT, Gilkeson GS, Seldin MF, Yin H, Baechler EC, Li QZ, Wakeland EK, Bruner GR, Kaufman KM, Kelly JA. Genome-wide association scan in women with systemic lupus erythematosus identifies susceptibility variants in ITGAM, PXK, KIAA1542 and other loci. Nat Genet 2008; 40 : 204–210.
184. Gateva V, Sandling JK, Hom G, Taylor KE, Chung SA, Sun X, Ortmann W, Kosoy R, Ferreira RC, Nordmark G, Gunnarsson I, Svenungsson E, Padyukov L, Sturfelt G, Jönsen A, Bengtsson AA, Rantapää-Dahlqvist S, Baechler EC, Brown EE, Alarcón GS, Edberg JC, Ramsey-Goldman R, McGwin G Jr, Reveille JD, Vilá LM, Kimberly RP, Manzi S, Petri MA, Lee A, Gregersen PK, Seldin MF, Rönnblom L, Criswell LA, Syvänen AC, Behrens TW, Graham RR. A largescale replication study identifies TNIP1, PRDM1, JAZF1, UHRF1BP1 and IL10 as risk loci for systemic lupus erythematosus. Nat Genet 2009; 41 : 1228–1233
185. Yang W, Shen N, Ye DQ, Liu Q, Zhang Y, Qian XX, Hirankarn N, Ying D, Pan HF, Mok CC, Chan TM, Wong RW, Lee KW, Mok MY, Wong SN, Leung AM, Li XP, Avihingsanon Y, Wong CM, Lee TL, Ho MH, Lee PP, Chang YK, Li PH, Li RJ, Zhang L, Wong WH, Ng IO, Lau CS, Sham PC, Lau YL; Asian Lupus Genetics Consortium. Genome-wide association study in Asian populations identifies variants in ETS1 and WDFY4 associated with systemic lupus erythematosus. PLoS Genet 2010; 6: e1000841.
186. Luo X, Yang W, Ye DQ, Cui H, Zhang Y, Hirankarn N, Qian X, Tang Y, Lau YL, de Vries N, Tak PP, Tsao BP, Shen N. A Functional Variant in MicroRNA-146a Promoter Modulates Its Expression and Confers Disease Risk for Systemic Lupus Erythematosus. PLoS Genet 2011; 7: e1002128.
187. Liang Y, Ridzon D, Wong L, Chen C. Characterization of microRNA expression profiles in normal human tissues. BMC Genomics 2007; 8 : 166.
188. Ribeiro-dos-Santos Ś, Khayat AS, Silva A, Alencar DO, Lobato J, Luz L, Pinheiro DG, Varuzza L, Assumpćčo M, Assumpćčo P, Santos S, Zanette DL, Silva WA Jr, Burbano R, Darnet S. Ultra-deep sequencing reveals the microRNA expression pattern of the human stomach. PLoS One 2010; 5: e13205.
189. Merkerova M, Vasikova A, Belickova M, Bruchova H. MicroRNA expression profiles in umbilical cord blood cell lineages. Stem Cells Dev 2010; 19 : 17-2
190. Hanoun N, Delpu Y, Suriawinata AA, Bournet B, Bureau C, Selves J, Tsongalis GJ, Dufresne M, Buscail L, Cordelier P, Torrisani J. The silencing of microRNA 148a production by DNA hypermethylation is an early event in pancreatic carcinogenesis Clin Chem. 2010; 56 : 1107-18.
191. Lujambio A, Calin GA, Villanueva A, Ropero S, Sánchez-Céspedes M, Blanco D, Montuenga LM, Rossi S, Nicoloso MS, Faller WJ, Gallagher WM, Eccles SA, Croce CM, Esteller M. A microRNA DNA methylation signature for human cancer metastasis. Proc Natl Acad Sci USA 2008, 105 : 13556–13561.
192. Duursma AM, Kedde M, Schrier M, le Sage C, Agami R. miR-148 targets human DNMT3b protein coding region. RNA 2008; 14 : 872-7.
193. Liu X, Zhan Z, Xu L, Ma F, Li D, Guo Z, Li N, Cao X. MicroRNA-148/152 impair innate response and antigen presentation of TLR-triggered dendritic cells by targeting CaMKIIα. J Immunol 2010; 185 : 7244-51.
194. Dai R, Zhang Y, Khan D, Heid B, Caudell D, Crasta O. Identification of a common lupus disease-associated microRNA expression pattern in three different murine models of lupus. PLoS One 2010; 5: e14302.
195. Divekar AA, Dubey S, Gangalum PR, Singh RR. Dicer insufficiency and micro - RNA-155 overexpression in lupus regulatory T cells: an apparent paradox in the setting of an inflammatory milieu. J Immunol 2011; 186 : 924–30.
196. Gregory PA, Bert AG, Paterson EL, Barry SC, Tsykin A, Farshid G, Vadas MA, Khew-Goodall Y, Goodall GJ. The miR-200 family and miR-205 regulate epithelial to mesenchymal transition by targeting ZEB1 and SIP1. Nat Cell Biol 2008; 10 : 593-601.
197. Kato M, Zhang J, Wang M, Lanting L, Yuan H, Rossi JJ, Natarajan R. MicroRNA-192 in diabetic kidney glomeruli and its function in TGF-beta-induced collagen expression via inhibition of E-box repressors. Proc Natl Acad Sci U S A 2007; 104 : 3432-7.
198. Alevizos I, Illei GG. MicroRNAs in Sjögren’s syndrome as a prototypic autoimmune disease. Autoimmun Rev 2010; 9 : 618-21.
199. Alevizos I, Illei GG. MicroRNAs as biomarkers in rheumatic diseases. Nat Rev Rheumatol. 2010; 6 : 391-8.
200. Michael A, Bajracharya SD, Yuen PS, Zhou H, Star RA, Illei GG, Alevizos I. Exosomes from human saliva as a source of microRNA biomarkers. Oral Dis 2010; 16 : 34-8.
201. Maurer B, Stanczyk J, Jüngel A, Akhmetshina A, Trenkmann M, Brock M, Kowal-Bielecka O, Gay RE, Michel BA, Distler JH, Gay S, Distler O. MicroRNA-29, a key regulator of collagen expression in systemic sclerosis. Arthritis Rheum 2010; 62 : 1733-43.
202. Li H, Yang R, Fan X, Gu T, Zhao Z, Chang D, Wang W, Wang C. MicroRNA array analysis of microRNAs related to systemic scleroderma. Rheumatol Int. 2010; (Epub ahead of print)
203. Eisenberg I, Alexander MS, Kunkel LM. miRNAS in normal and diseased skeletal muscle. J Cell Mol Med 2009; 13 : 2-11.
Labels
Dermatology & STDs Paediatric rheumatology Rheumatology
Article was published inCzech Rheumatology

2011 Issue 4
Most read in this issue- Funkčný stav a kvalita života u pacientov s včasnou a etablovanou reumatoidnou artritídou
- Diferenciální exprese microRNA u systémových autoimunitních onemocnění
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career