-
Medical journals
- Career
Skóre polygenního rizika (PRS) a jeho potenciál pro stratifikaci rizika karcinomu prsu
Authors: M. Hovhannisyan 1; P. Kleiblová 1,2; P. Nehasil 1,3,4; J. Soukupová 1; P. Zemánková 1,3; Z. Kleibl 1,3; M. Janatová 1
Authors‘ workplace: Laboratoř onkogenetiky, Ústav lékařské bio chemie a laboratorní dia gnostiky, 1. LF UK a VFN v Praze 1; Ústav bio logie a lékařské genetiky, 1. LF UK a VFN v Praze 2; Ústav patologické fyziologie, 1. LF UK, Praha 3; Klinika pediatrie a dědičných metabolických poruch, 1. LF UK a VFN v Praze 4
Published in: Klin Onkol 2023; 36(3): 198-205
Category: Review
doi: https://doi.org/10.48095/ccko2023198Overview
Východiska: Karcinom prsu je komplexní, multifaktoriální onemocnění, na jehož vzniku se podílejí i genetické faktory. Kromě relativně vzácných patogenních variant ve vysoce či středně penetrantních nádorových predispozičních genech ovlivňují riziko vzniku karcinomu prsu i početné nízce penetrantní alely, které vnímáme jako faktory polygenní dědičnosti. Nízce penetrantní alely mají samostatně zanedbatelný význam, avšak jejich kumulativní účinek může dosáhnout klinicky významných hodnot a lze jej vyjádřit pomocí skóre polygenního rizika (polygenic risk score – PRS), které se v současnosti zvažuje jako možný nástroj pro přesnější odhad absolutního a kumulativního rizika onkologického onemocnění u konkrétní osoby. Cíl: V zahraničí již bylo vyvinuto několik setů pro jednonukleotidový polymorfizmus (single nucleotide polymorphism – SNP) pro stanovení PRS za účelem využití metody v individualizaci rizika v klinické praxi. Následující text si klade za cíl osvětlit základy stanovení PRS a jeho interpretaci jako možného predikčního nástroje. Využití PRS by však mělo být vždy podmíněno genetickým vyšetřením patogenních variant v nádorových predispozičních genech.
Klíčová slova:
PRS – polygenní dědičnost karcinomu prsu – stanovení rizika karcinomu prsu – sporadický karcinom prsu
Úvod
Ve vyspělých zemích patří onkologická onemocnění k nejčastějším příčinám úmrtí se setrvale rostoucím významem [1]. Po nemelanomových nádorech kůže je karcinom (ca) prsu druhé nejčastější nádorové onemocnění v populaci žen v ČR [2]. Jeho celoživotní riziko přesahuje 10 %. Vysoká incidence (131/100 000 žen v roce 2020) a mortalita (32/100 000 žen v roce 2020) ca prsu představuje v posledních dekádách významnou zdravotní i socioekonomickou zátěž [3]. Z těchto důvodů byl v roce 2002, jako pro první nádorové onemocnění u nás, zaveden populační screening ca prsu pro všechny ženy od věku 45 let ve dvouletých intervalech. Třebaže toto opatření vedlo ke včasnému zachycení onemocnění u pacientek, existuje obava, že plošné testování může vést k nadsazené incidenci ca prsu (falešně pozitivní výsledky a overdiagnosis), byť v naprosté menšině případů [4,5].
Ca prsu vzniká nejčastěji jako sporadické onemocnění v důsledku získaných poruch genomové DNA, které postihují geny ovlivňující zejména proliferaci buněk prsní epitelové tkáně [6]. Hlavní klinické podskupiny onemocnění jsou určovány přítomností a aktivitou intracelulárních steroidních (estrogenních a progesteronových) receptorů a membránového receptoru pro epidermální růstový faktor ErbB2 (HER2/neu), které normálně regulují mitotickou aktivitu mamární tkáně [7–9]. Ca prsu je tak velmi heterogenním onemocněním se značně proměnlivým biologickým chováním.
Pravděpodobnost vzniku ca prsu ovlivňuje řada rizikových faktorů – vrozených, behaviorálních i environmentálních. Mezi známé rizikové faktory můžeme zařadit časnou menarche spolu s pozdní menopauzou, nuliparitou, těhotenstvím v pozdním věku [10,11] nebo krátkou dobou kojení [12]. Celkové riziko dále mírně zvyšuje obezita, kouření a konzumace alkoholu [12–14]. Mezi nejzávažnější rizikové faktory patří pozitivní onkologická rodinná anamnéza [15,16]. Riziko onemocnění zdvojnásobuje přítomnost ca prsu u jedné přímé příbuzné (matka, sestra, dcera) pacientky, s pozitivní rodinnou anamnézou zároveň stoupá riziko rozvoje onemocnění v mladším věku [16,17].
V méně než 10 % všech případů se setkáváme s dědičnou formou ca prsu, která vzniká na základě přítomnosti zárodečné mutace v některém z nádorových predispozičních genů. Příklady rozvětvených rodin s opakovaným výskytem onemocnění ukazují, že hereditární formy ca prsu vznikají jako monogenní onemocnění s autozomálně dominantním typem dědičnosti nádorové predispozice [18]. Prokázané příčinné dědičné mutace se však vyskytují pouze přibližně u třetiny žen s pozitivní rodinnou anamnézou, a to častěji u pacientek, v jejichž rodinné anamnéze se vyskytuje kromě ca prsu i ca ovarií (23 vs. 59 %) [19]. U pacientek se zvýšeným výskytem ca prsu v rodině, kde se nepodařilo určit příčinnou dědičnou mutaci, hovoříme o familiárním onemocnění.
Charakterizace rozsáhlých rodin s mno - hočetným výskytem ca prsu a pokroky v metodách molekulární genetiky umožnily v 90. letech minulého století objev hlavních predispozičních genů BRCA1 [20] a BRCA2 [21]. Přítomnost vysoce penetrantních mutací v obou genech podmiňuje vznik dědičné formy ca prsu a ovarií. Význam obou genů podtrhuje i relativně vysoká prevalence jejich dědičných mutací, které tvoří přibližně polovinu všech případů hereditární formy ca prsu. Proporce nosičů mutací v obou genech vykazují významné populační rozdíly, přičemž v ČR převládají dědičné alterace v genu BRCA1 [19,22–25]. Třebaže se po objevu genů BRCA1 a BRCA2 na sklonku tisíciletí podařilo identifikovat desítky dalších genů zvyšujících riziko ca prsu, žádný z nich kombinací klinicky mimořádně významného rizika a vysoké prevalence nosičů nezastínil význam genů BRCA1 a BRCA2 [26]. Je však zajímavé, že většina predispozičních genů spojených se vznikem dědičné formy ca prsu kóduje proteiny, které se podílejí na mechanizmu oprav genomové DNA a intracelulární signalizaci reagující na přítomnost jejího poškození [26].
Predispoziční geny lze kategorizovat podle míry rizika (obvykle vyjádřené jako poměr šancí – odds ratio (OR)) vzniku ca prsu u nosičů jejich patogenních zárodečných mutací. Vysoce penetrantní mutace v genech BRCA1, BRCA2, PALB2, PTEN [27], TP53 [28] či STK11 [29] zvyšují riziko vývoje ca prsu proti běžné populaci více než pětinásobně (OR > 5), čímž dosahují celoživotního rizika na úrovni 60–75 %. U středně penetrantních mutací v genech CHEK2 a ATM je riziko zvýšené středně (OR 2–5), a celoživotní riziko tak dosahuje hodnot kolem 25–30 % [24,30].
Zpracování rozsáhlých souborů vysoce rizikových pacientek s ca prsu prokázalo, že u nezanedbatelné části nemocných není možné identifikovat monogenní příčinnou zárodečnou změnu. Takové případy vedly k domněnce, že příčinou onemocnění je polygenní dědičnost, kdy onemocnění vznikají aditivním působením řady variant s nízkou penetrancí [31].
Na základě asociačních studií sledujících výskyt ca prsu mezi příbuznými a mezi dvojčaty byly charakterizovány varianty s nízkou penetrancí, které zvyšují (či případně snižují) riziko ca prsu méně než dvojnásobně [32]. Třebaže míra rizika spojená s jednotlivými samotnými nízce penetrantními variantami je klinicky prakticky bezvýznamná (OR se obvykle pohybuje v intervalu 0,9–1,1), vznik ca prsu může být významně ovlivněn rizikovými genotypy složenými z mnoha (desítek až stovek) nízce penetrantních variant. Na rozdíl od patogenních mutací ve vysoce a středně penetrantních genech se klinicky významné nízce penetrantní varianty vyskytují se značnou populační četností (5–95 %) [33,34]. Dopad těchto variant na riziko vzniku ca prsu může být pozitivní i negativní, a některé varianty tak mohou zvyšovat riziko, zatímco jiné míru rizika snižují.
V nejjednodušší podobě mohou takovéto varianty reprezentovat např. záměny aminokyselin v kódující sekvenci genů, jejichž proteinové produkty souvisejí s rozvojem onemocnění. S ohledem na skutečnost, že kódující genetická informace tvoří jen malou část genomu, je mnohem častější, že asociovaná varianta leží v nekódujících či intergenových oblastech a mechanizmus působení takovýchto variant je obvykle nejasný. Na jedné straně si můžeme představit, že jednotlivé varianty mohou ovlivňovat genovou expresi okolních genů (v podobě změn enhancerů či silencerů) nebo ovlivňují biogenezi nekódujících RNA. Můžeme však uvažovat i o tom, že identifikované nízce penetrantní varianty jsou jen funkčně bezvýznamnými genetickými markery doposud neznámých funkčních variant, se kterými jsou v genové vazbě [35,36].
Celogenomové asociační studie
Podstatou identifikace nízce penetrantních alel je porovnání četnosti výskytu variant zárodečné DNA (nejčastěji SNP) napříč genomem v souboru dobře charakterizovaných pacientek s ca prsu s velikostně srovnatelným kontrolním souborem žen bez onemocnění. Výstupem takovéto studie (tzv. genome-wide association study – GWAS) je identifikace konkrétních sekvenčních variant DNA v genomu, které jsou spojeny s výskytem onemocnění.
S ohledem na skutečnost, že vliv jednotlivých variant na riziko ca prsu je velmi malý, musely všechny GWAS studie zahrnout rozsáhlé soubory pacientů a kontrol. U každého vyšetřovaného vzorku byly analyzovány desítky až stovky tisíc genomových lokusů, což ve výsledku znamenalo vzájemně porovnávat obrovské množství komplexních genotypů. Potřebnou technologickou kapacitu pro masivní genotypizaci katalyzoval na počátku milénia až rozvoj DNA mikročipů, které umožňují poměrně jednoduše analyzovat tisíce genetických variant u tisíců vyšetřovaných subjektů [34,37,38].
Britská studie z roku 2007, jedna z prvních GWAS zaměřených na identifikaci polygenního rizika u ca prsu, analyzovala pomocí DNA mikročipů přes 250 000 variant SNP. Studie sestávala ze tří fází, kdy se postupně zužoval počet analyzovaných SNP a zvyšoval počet probandek. Ve třetí fázi studie, kde byly kromě Britek zahrnuty též probandky z Evropy, USA, Austrálie a Asie, bylo vyšetřeno 30 SNP u 21 860 pacientek a 22 578 kontrol. Výstupem této studie byla identifikace pěti nejrizikovějších SNP variant v genech FGFR2, LSP1, TNRC9 a MAP3K; avšak i u varianty v genu FGFR2 s nejsilnější asociací bylo riziko nízké (OR = 1,26; 95% CI = 1,23–1,30) [34].
Další technologický pokrok, zlevnění vysokokapacitních technologií a mezinárodní konzorciální spolupráce umožnily v následujících letech výrazně vyšší efektivitu v identifikaci nízce penetrantních variant asociujících s ca prsu. Studie Collaborative Oncological Gene-environment Study (COGS), která vzešla ze spolupráce mezinárodních konzorcií Consortium of Investigators of Modifiers of BRCA1/2 (CIMBA) a Breast Cancer Association Consortium (BCAC), již analyzovala > 200 000 SNP [33]. Na tuto studii plynule navázala studie OncoArray z roku 2017, která analyzovala > 500 000 SNP variant [39]. Do roku 2020 byly v rámci těchto projektů analyzovány varianty v genomech > 200 000 jedinců [40].
Uvedené studie umožnily stanovit frekvence nízce penetrantních variant v populacích vyšetřovaných pacientek a kontrol, vyčíslit míru rizika (v podobě OR) pro každou z těchto variant a rovněž hodnotu p, která udává statistickou významnost rozdílu ve frekvenci mezi skupinou pacientek s ca prsu a kontrolami (graf 1). Aktualizovaná data získaná v rámci těchto studií jsou volně dostupná na stránkách BCAC [41].
Graph 1. Vizualizace výstupu COGS studie v podobě tzv. Manhattan grafu. 
Každý datový bod grafu představuje jednu z 199 961 úspěšně analyzovaných variant jednonukleotidového polymorfi zmu (SNP) [40]. Na ose x je vynesena pozice SNP na jednotlivých chromozomech, na ose y je vynesena p-hodnota (jako záporný dekadický logaritmus) vyjadřující statistickou významnost rozdílu ve výskytu va rianty v souboru 133 384 pacientek s ca prsu (většinově ze severní a západní Evropy) a v souboru 113 789 kontrol (srovnatelný původ). Jelikož se mezi soubory pacientek a kontrol porovnávají frekvence stovek tisíc genomových variant, je nezbytné upravit obvyklou hodnotu p statistické významnosti (p < 0,05) na mnohočetné testování (což si v praxi můžeme představit jako vydělení hladiny významnosti počtem analyzovaných variant SNP). Žlutá linka označuje hranici, nad kterou se vyskytují SNP statisticky signifi kantně asociující s výskytem ca prsu po korekci hodnoty p na mnohočetné testování (p < 5×10–8). U SNP variant s co nejvýznamnějším rozdílem distribuce alel mezi nemocnými a zdravými ženami (p < 5 × 10–8) byla stanovena rizika; OR < 1 pro protektivní varianty a OR > 1 pro varianty rizikové [33,36,40,42].
Skóre polygenního rizika
Jak vyplývá z předchozích poznatků, nízce penetrantní alely samy o sobě ovlivňují riziko vzniku ca prsu jen nepatrně. V populacích se však vyskytují v relativně vysokých frekvencích (> 5 %), a jednotlivec je tak nosičem mnoha variant, jejichž účinky se sčítají. V případě nakupení rizikových variant může aditivní účinek dosáhnout klinicky významných hodnot srovnatelných s riziky patogenních variant ve středně penetrantních genech.
Pro výpočet skóre polygenního rizika (polygenic risk score – PRS) musí být ze souboru SNP variant navržených předchozími GWAS vyloučeny lokusy ve vazbě pro odstranění redundantní informace. Tento set se následně podrobuje redukci nadsazené síly efektu pomocí metod regresní analýzy (např. lasso – least absolute shrinkage and selection operator). Teprve pak je možné pokračovat v genetickém testování probandů kontrolního souboru.
Určení rizik (OR) asociovaných s významnými rizikovými variantami SNP umožňuje vyjádřit kumulativní účinek polygenní zátěže pro každého jedince v podobě PRS. Výpočet se provádí dle rovnice:
PRSj=∑Ni logORi × gij,
kdy PRS jedince j tvořené z N SNP variant je sumou součinů dekadického logaritmu OR alely i a alelické dávky g, která nabývá hodnot 0, 1, 2 (0 – homozygot v referenční alele, 1 – heterozygot, 2 – homozygot v rizikové/protektivní alele).
Takto získané PRS samo o sobě vypovídá pouze o tom, zda u konkrétního jedince převažují rizikové, či protektivní alely z vybraného setu. Výsledek nevypovídá o míře rizika, které PRS obnáší pro jedince ve vyšetřované populaci. Pro toto zhodnocení je nutné porovnat ženy ve vyšetřovaném souboru (pacientky i zdravé ženy) mezi sebou a stanovit riziko onemocnění pro dosažené hodnoty PRS.
Jak již bylo řečeno, SNP varianty, na kterých je model PRS založen, samy o sobě nesou minimální riziko a nepodléhají tak selekčnímu tlaku, který by redukoval jejich výskyt. V běžné populaci, u níž je známé průměrné kumulativní riziko onemocnění, se proto předpokládá normální distribuce hodnot PRS (graf 2A).
Graf 2A. Hypotetická distribuční křivka skóre polygenního rizika (PRS) dle standardního rozdělení. 
Soubor zahrnuje zdravé kontroly i pacientky. Osa x představuje rozdělení PRS do percentilů a jednotek jim odpovídajících standardních odchylek (SD). Většina probandek spadá do rozpětí jedné SD, kterému odpovídá průměrné populační riziko. Uvedené hodnoty jsou pouze ilustrativní. Riziko onemocnění u jedince je vyjádřeno buď na základě proporčního vyjádření v percentilových kategoriích, nebo na základě vzdálenosti hodnot změřeného skóre od mediánu jako směrodatná odchylka (SD). Set SNP variant je kalibrován pro konkrétní populaci tak, aby průměrné PRS skóre odpovídalo průměrnému riziku, kterému odpovídá OR rovno 1 (graf 2B).
Graf 2B. Křivka hypotetického rozložení hodnot skóre polygenního rizika (PRS) v populaci žen s ca prsu. 
Na ose x jsou seřazeny všechny vyšetřované ženy podle stoupající hodnoty PRS. Podle potřeby (a klinického významu) můžeme zobrazené pacientky kategorizovat do skupin (např. kvantilových či decilových). Osa y označuje standardizované hodnoty stoupajícího rizika ca prsu (poměr šancí – OR), kde hodnota 1 označuje riziko běžné populace. Kosočtverce s anténami označují hodnoty OR v jednotlivých kvintilech (a horním a dolním 5% resp. 95% percentilu) s 95% intervalem spolehlivosti. V tomto případě je riziko v horním 95% percentilu na hranici klinické závažnosti (atakuje hranici 20% celoživotního rizika vzniku ca prsu). Pokud bychom uvažovali o využití PRS modelu používajícího soubor jednonukleotidových polymorfi zmů uvedený v našem hypotetickém případě, bylo by vhodné analyzovat pacientky ještě v užším extrémním souboru (horní percentil – 99–100 %), kde by hodnoty celoživotního rizika pravděpodobně již přesáhly 25 %. Uvedené hodnoty jsou pouze ilustrativní. Sdílená vstupní data GWAS ze studií COGS a OncoArray tvoří základ většiny setů významných SNP pro konstrukci PRS u pacientek s ca prsu v různých studiích u různých populací. Navzdory očekávání je však mezi sety poměrně malý překryv (schéma 1). Sety se od sebe odlišují vlivem rozdílných četností alel u konkrétních populací, na kterých byly sety testovány (německá a britská), nových dat (databáze COGS je neustále aktualizována), kritérií výběru či rozdílných prioritizací variant ve vazbě.
Schéma 1. Překryv SNP setů mezi studiemi Mavaddat z roku 2015 (set 77) a 2019 (set 313) [44,48] a Kuchenbaecker z roku 2017 (set 88) [47]. ![Schéma 1. Překryv SNP setů mezi studiemi
Mavaddat z roku 2015 (set 77) a 2019 (set
313) [44,48] a Kuchenbaecker z roku 2017
(set 88) [47].](https://pl-master.mdcdn.cz/media/image_pdf/682d7f11ec0329989cfdf429c7ab0769.png?version=1686996250)
Zatímco set 77 byl koncipován jako predikční nástroj pro ER+ a ER− nádory u neselektované populace a set 313 na něj navázal za účelem zpřesnění jeho prediktivní schopnosti, set 88 byl vyvinut primárně pro nosičky mutací v BRCA1/2.
ER – estrogenový receptor, SNP – jednonukleotidový polymorfi zmusVytvořené sety SNP pro výpočet PRS (tab. 1) jsou koncipované jako pomocný nástroj pro identifikaci rizika vzniku ca prsu nebo jako nástroj zpřesňující odhad věkově specifického rizika vzniku onemocnění pro nosičky hereditárních mutací ve známých vysoce penetrantních (např. BRCA1, BRCA2, PALB2) či středně penetrantních (např. CHEK2, ATM) predispozičních genech [43].
Table 1. Originální studie skóre polygenního rizika (PRS) u bělošských pacientek s ca prsu. Některé modely již v současnosti zahrnují hodnoty PRS do výpočtu rizika vzniku ca prsu vedle známých rizikových faktorů jako rodinná anamnéza, životní styl, či denzita prsní tkáně. 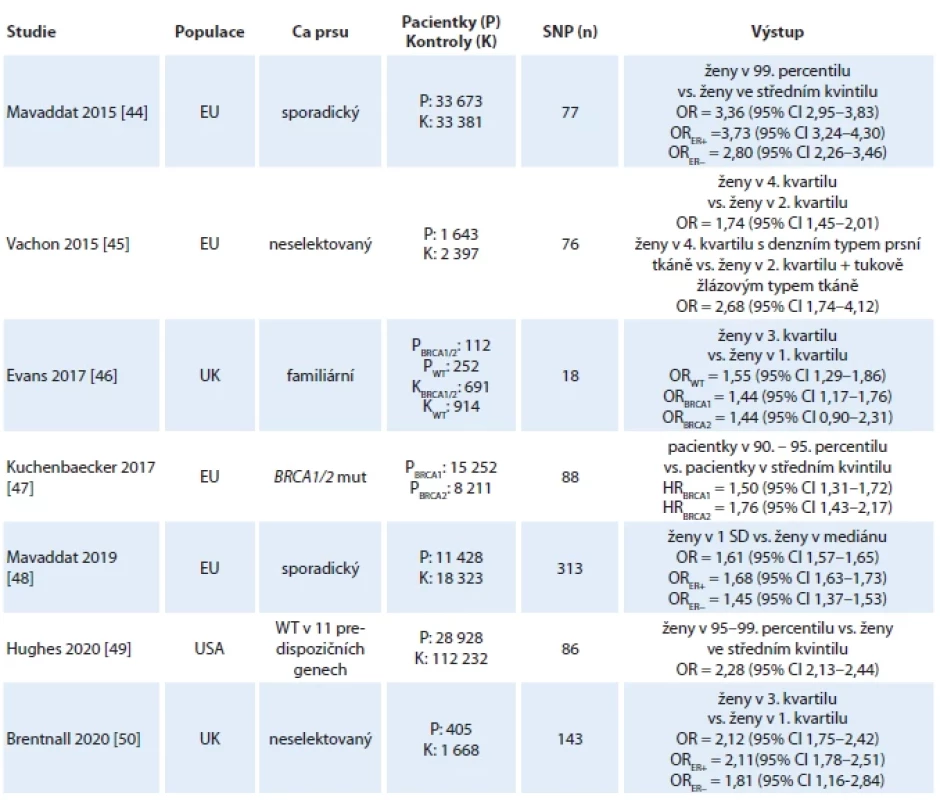
CI – interval spolehlivosti, ER – estrogenové receptory, HR – poměr rizik, mut – mutace, n – počet případů, OR – poměr šancí, SD – směrodatná odchylka, SNP – jednonukleotidový polymorfi zmus, wt – wild type Jak je patrné z uvedených výsledků, vysoké hodnoty PRS jsou spojeny se zvýšeným rizikem ca prsu bez ohledu na počet SNP zahrnutých do výpočtu. Především v odlehlých percentilech hodnot PRS riziko překračuje dvojnásobek populačního průměru [44,49].
Některé z uvedených setů byly dále testovány na probandkách s jinými selekčními kritérii. Opakované studie se zaměřovaly na nosičky mutací v BRCA1/2 a CHEK2, které jsou nejvýznamnějšími genetickými faktory pro ca prsu, a ukázaly spojitost mezi PRS a věkem nástupu onemocnění [43,51,52].
Interpretaci rizika však komplikují další faktory. Přestože studie ukazují, že vysoké PRS s sebou nese zvýšené riziko ca prsu, jeho výpovědní hodnota je bez dalších informací o genetické zátěži jedince výrazně omezená vzhledem k relativně vysoké prevalenci patogenních mutací v genech BRCA1/2 a dalších středně penetrantních genech [19,22,53,54].
Budoucnost PRS
Dosavadní studie ukazují praktické využití PRS nejen pro stanovení rizika u jednotlivých žen v rámci běžné populace, ale také jako modifikátor rizika u nosiček známých mutací. Než však bude možné implementovat stanovení PRS do rutinní diagnostiky, musí nejprve proběhnout validace stanovení PRS na konkrétní populaci. Dále je nutné zdůraznit, že bez informace o přítomnosti vysoce penetrantní alely je predikční schopnost PRS značně omezená.
V současné době probíhá řada studií zabývajících se implementací PRS do klinické praxe. Některé laboratoře již PRS zahrnují do stávajících modelů predikce rizika ca prsu v rámci vyšetřovacích panelů sekvenování nové generace nebo jako samostatný test. Společnost Myriad Genetics nabízí nástroj pro predikci rizika vzniku ca prsu ve svém komerčně dostupném testu myRisk®Hereditary Cancer, který poskytuje odhady pětiletého a celoživotního rizika vzniku ca prsu [55]. AmbryScore (Ambry Genetics) nabízí testy pro predikci rizika BC založené na Tyrer-Cusick modelu, který zahrnuje klinickou anamnézu (věk, etnicita, rodinná anamnéza) plus PRS, který obsahuje 100 variant SNP [56]. Přes zvyšující se popularitu stanovení PRS, která ukazují, že skóre signifikantně zvyšuje, nebo snižuje riziko vzniku ca prsu v extrémních horních či dolních percentilových skupinách analyzovaných souborů pacientek s ca prsu, jsou v současnosti velmi omezeně dostupné údaje o senzitivitě a specifičnosti vyšetření pro individuální pacientky.
Globální implementace PRS pro odhad rizika ca prsu musí brát v potaz i další současné limitace vyšetření, jako jsou nedostatek dat pro mimoevropské (a rovněž jiné než západoevropské) populace, korelace/interference PRS s dalšími rizikovými faktory (např. s nosičstvím germinálních mutací v nádorových predispozičních genech s nejasným dopadem na riziko ca prsu), nestandardizovaný počet zahrnutých SNP a rozdílné statistické metody pro vlastní výpočet PRS a s ním spojeným rizikem. Rutinní klinické využití PRS jako nástroje pro individualizaci zahájení onkologické prevence – např. mamografického screeningu u konkrétních pacientek – tak bude vyžadovat robustní hodnocení potenciálu PRS pomocí standardních klinických studií.
Poděkování
Práce byla podpořena grantem Agentury pro zdravotnický výzkum MZČR NU20-09-00355, grantem MŠMT BBMRI-CZ LM2023033, projekty Univerzity SVV260631 a COOPERATIO.
Autoři deklarují, že v souvislosti s předmětem studie nemají žádné komerční zájmy.
The authors declare that they have no potential confl icts of interest concerning drugs, products, or services used in the study.
Redakční rada potvrzuje, že rukopis práce splnil ICMJE kritéria pro publikace zasílané do bi omedicínských časopisů.
The Editorial Board declares that the manuscript met the ICMJE recommendation for biomedical papers.
RNDr. Markéta Janatová, Ph.D.
Ústav lékařské biochemie a laboratorní diagnostiky,
1. LF UK a VFN v Praze
Na Bojišti 3
128 08 Praha 2
e-mail: mjana@lf1.cuni.cz
Obdrženo/Submitted: 14. 12. 2023
Přijato/Accepted: 10. 1. 2023Klin Onkol 2023; 36(3): 198 – 205
Sources
1. Global Cancer Observatory. Cancer over time. [online]. Dostupné z: https: //gco.iarc.fr.
2. ÚZIS ČR. Zdravotnická ročenka České republiky 2019. [online]. Dostupné z: https: //www.uzis.cz/res/f/008 381/zdrroccz2019.pdf.
3. SVOD. Epidemiologie zhoubných nádorů v České republice. [online]. Dostupné z: http: //www.svod.cz.
4. Siu AL. Screening for breast cancer: US preventive services task force recommendation statement. Ann Intern Med 2016; 164 (4): 279–296. doi: 10.7326/M15 - 2886.
5. Bulliard JL, Beau AB, Njor S et al. Breast cancer screening and overdiagnosis. Int J Cancer 2021; 149 (4): 846–853. doi: 10.1002/ijc.33602.
6. Rahman N. Realizing the promise of cancer predisposition genes. Nature 2014; 505 (7483): 302–308. doi: 10.1038/nature12981.
7. Honrado E, Benítez J, Palacios J. The molecular pathology of hereditary breast cancer: genetic testing and therapeutic implications. Mod Pathol 2005; 18 (10): 1305–1320. doi: 10.1038/modpathol.3800453.
8. Dorgan JF, Longcope C, Franz C et al. Endogenous sex hormones and breast cancer in postmenopausal women: reanalysis of nine prospective studies. J Natl Cancer Inst 2002; 94 (8): 606–616. doi: 10.1093/jnci/94.8.606.
9. Hulka BS, Liu ET, Lininger RA. Steroid-hormones and risk of breast-cancer. Cancer 1994; 74 (3): 1111–1124. doi: 10.1002/1097-0142 (19940801) 74 : 3+<1111:: aid-cncr2820741520>3.0.co; 2 - l.
10. MacMahon B, Cole P, Lin TM et al. Age at first birth and breast cancer risk. Bull World Health Organ 1970; 43 (2): 209–221.
11. Albrektsen G, Heuch I, Hansen S et al. Breast cancer risk by age at birth, time since birth and time intervals between births: exploring interaction effects. Br J Cancer 2005; 92 (1): 167–175. doi: 10.1038/sj.bjc.6602302.
12. Colditz GA, Rosner B. Cumulative risk of breast cancer to age 70 years according to risk factor status: data from the Nurses’ Health Study. Am J Epidemiol 2000; 152 (10): 950–964. doi: 10.1093/aje/152.10.950.
13. Eliassen AH, Colditz GA, Rosner B et al. Adult weight change and risk of postmenopausal breast cancer. JAMA 2006; 296 (2): 193–201. doi: 10.1001/jama.296.2.193.
14. Calle EE, Rodriguez C, Walker-Thurmond K et al. Overweight, obesity, and mortality from cancer in a prospectively studied cohort of U.S. adults. N Engl J Med 2003; 348 (17): 1625–1638. doi: 10.1056/NEJMoa021423.
15. Himes DO, Root AE, Gammon A et al. Breast cancer risk assessment: calculating lifetime risk using the Tyrer--Cuzick model. J Nurse Pract 2016; 12 (9): 581–592. doi: 10.1016/j.nurpra.2016.07.027.
16. Pharoah PDP, Day NE, Duffy S et al. Family history and the risk of breast cancer: a systematic review and meta-analysis. Int J Cancer 1997; 71 (5): 800–809. doi: 10.1002/ (sici) 1097-0215 (19970529) 71 : 5<800:: aid-ijc18>3.0.co; 2-b.
17. Brewer HR, Jones ME, Schoemaker MJ et al. Family history and risk of breast cancer: an analysis accounting for family structure. Breast Cancer Res Treat 2017; 165 (1): 193–200. doi: 10.1007/s10549-017-4325-2.
18. Broca P. Traité des tumeurs. Paris: P. Asselin 1866.
19. Pohlreich P, Zikan M, Stribrna J et al. High proportion of recurrent germline mutations in the BRCA1 gene in breast and ovarian cancer patients from the Prague area. Breast Cancer Res 2005; 7 (5): R728–736. doi: 10.1186/bcr1282.
20. Miki Y, Swensen J, Shattuck-Eidens D et al. A strong candidate for the breast and ovarian cancer susceptibility gene BRCA1. Science 1994; 266 (5182): 66–71. doi: 10.1126/science.7545954.
21. Wooster R, Bignell G, Lancaster J et al. Identification of the breast cancer susceptibility gene BRCA2. Nature 1995; 378 (6559): 789–792. doi: 10.1038/378789a0.
22. Mateju M, Stribrna J, Zikan M et al. Population-based study of BRCA1/2 mutations: family history based criteria identify minority of mutation carriers. Neoplasma 2010; 57 (3): 280–285. doi: 10.4149/neo_2010_03_280.
23. Ponder BAJ, Day NE, Easton DF et al. Prevalence and penetrance of BRCA1 and BRCA2 mutations in a population-based series of breast cancer cases. Br J Cancer 2000; 83 (10): 1301–1308. doi: 10.1054/bjoc.2000.1407.
24. Hu C, Hart SN, Gnanaolivu R et al. A population-based study of genes previously implicated in breast cancer. N Engl J Med 2021; 384 (5): 440–451. doi: 10.1056/NEJMoa2005936.
25. Machackova E, Claes K, Mikova M et al. Twenty years of BRCA1 and BRCA2 molecular analysis at MMCI – current developments for the classification of variants. Klin Onkol 2019; 32 (Suppl 2): 51–71. doi: 10.14735/amko2019S51.
26. Kleibl Z, Kristensen VN. Women at high risk of breast cancer: molecular characteristics, clinical presentation and management. Breast 2016; 28 : 136–144. doi: 10.1016/j.breast.2016.05.006.
27. Chen J, Lindblom P, Lindblom A. A study of the PTEN/MMAC1 gene in 136 breast cancer families. Hum Genet 1998; 102 (1): 124–125.
28. Børresen AL, Andersen TI, Garber J et al. Screening for germ line TP53 mutations in breast cancer patients. Cancer Res 1992; 52 (11): 3234–3236.
29. Leggett BA, Young JP, Barker M. Peutz-Jeghers syndrome: genetic screening. Expert Rev Anticancer Ther 2003; 3 (4): 518–524. doi: 10.1586/14737140.3.4.518.
30. Dorling L, Carvalho S, Allen J et al. Breast cancer risk genes – association analysis in more than 113,000 women. N Engl J Med 2021; 384 (5): 428–439. doi: 10.1056/NEJMoa1913948.
31. Pharoah PDP, Antoniou A, Bobrow M et al. Polygenic susceptibility to breast cancer and implications for prevention. Nat Gen 2002; 31 (1): 33–36. doi: 10.1038/ng 853.
32. Mucci LA, Hjelmborg JB, Harris JR et al. Familial risk and heritability of cancer among twins in nordic countries. JAMA 2016; 315 (1): 68–76. doi: 10.1001/jama.2015.17703.
33. Michailidou K, Hall P, Gonzalez-Neira A et al. Large - -scale genotyping identifies 41 new loci associated with breast cancer risk. Nat Gen 2013; 45 (4): 353–361. doi: 10.1038/ng.2563.
34. Easton DF, Pooley KA, Dunning AM et al. Genome-wide association study identifies novel breast cancer susceptibility loci. Nature 2007; 447 (7148): 1087–1093. doi: 10.1038/nature05887.
35. Wittkopp PJ, Kalay G. Cis-regulatory elements: molecular mechanisms and evolutionary processes underlying divergence. Nat Rev Genet 2012; 13 (1): 59–69. doi: 10.1038/nrg3095.
36. Michailidou K, Lindstrom S, Dennis J et al. Association analysis identifies 65 new breast cancer risk loci. Nature 2017; 551 (7678): 92–94. doi: 10.1038/nature24 284.
37. Pollack JR, Perou CM, Alizadeh AA et al. Genome-wide analysis of DNA copy-number changes using cDNA microarrays. Nat Genet 1999; 23 (1): 41–46. doi: 10.1038/12640.
38. Ntzani EE, Ioannidis JP. Predictive ability of DNA microarrays for cancer outcomes and correlates: an empirical assessment. Lancet 2003; 362 (9394): 1439–1444. doi: 10.1016/S0140-6736 (03) 14686-7.
39. Amos CI, Dennis J, Wang Z et al. The OncoArray Consortium: a network for understanding the genetic architecture of common cancers. Cancer Epidemiol Biomarkers Prev 2017; 26 (1): 126–135. doi: 10.1158/1055-9965.EPI-16-0106.
40. Zhang H, Ahearn TU, Lecarpentier J et al. Genome-wide association study identifies 32 novel breast cancer susceptibility loci from overall and subtype-specific analyses. Nat Genet 2020; 52 (6): 572–581. doi: 10.1038/s41588-020-0609-2.
41. Breast Cancer Association Consortium. [online]. Available from: https: //bcac.ccge.medschl.cam.ac.uk/bcacdata/.
42. Michailidou K, Beesley J, Lindstrom S et al. Genome-wide association analysis of more than 120,000 individuals identifies 15 new susceptibility loci for breast cancer. Nat Genet 2015; 47 (4): 373–380. doi: 10.1038/ng. 3242.
43. Borde J, Ernst C, Wappenschmidt B et al. Performance of breast cancer polygenic risk scores in 760 female CHEK2 germline mutation carriers. J Natl Cancer Inst 2021; 113 (7): 893–899. doi: 10.1093/jnci/djaa203.
44. Mavaddat N, Pharoah PDP, Michailidou K et al. Prediction of breast cancer risk based on profiling with common genetic variants. J Natl Cancer Inst 2015; 107 (5): djv036. doi: 10.1093/jnci/djv036.
45. Vachon CM, Pankratz VS, Scott CG et al. The contributions of breast density and common genetic variation to breast cancer risk. J Natl Cancer Inst 2015; 107 (5): dju397. doi: 10.1093/jnci/dju397.
46. Evans DG, Brentnall A, Byers H et al. The impact of a panel of 18 SNPs on breast cancer risk in women attending a UK familial screening clinic: a case-control study. J Med Genet 2017; 54 (2): 111–113. doi: 10.1136/jmedgenet-2016-104125.
47. Kuchenbaecker KB, McGuffog L, Barrowdale D et al. Evaluation of polygenic risk scores for breast and ovarian cancer risk prediction in BRCA1 and BRCA2 mutation carriers. J Natl Cancer Inst 2017; 109 (7): djw302. doi: 10.1093/jnci/djw302.
48. Mavaddat N, Michailidou K, Dennis J et al. Polygenic risk scores for prediction of breast cancer and breast cancer subtypes. Am J Hum Genet 2019; 104 (1): 21–34. doi: 10.1016/j.ajhg.2018.11.002.
49. Hughes E, Tshiaba P, Gallagher S et al. Development and validation of a clinical polygenic risk score to predict breast cancer risk. JCO Precis Oncol 2020; 4: PO.19.00360. doi: 10.1200/PO.19.00360.
50. Brentnall AR, van Veen EM, Harkness EF et al. A case--control evaluation of 143 single nucleotide polymorphisms for breast cancer risk stratification with classical factors and mammographic density. Int J Cancer 2020; 146 (8): 2122–2129. doi: 10.1002/ijc.32541.
51. Muranen TA, Greco D, Blomqvist C et al. Genetic modifiers of CHEK2*1100delC-associated breast cancer risk. Gen Med 2017; 19 (5): 599–603. doi: 10.1038/gim.2016.147.
52. Barnes DR, Rookus MA, McGuffog L et al. Polygenic risk scores and breast and epithelial ovarian cancer risks for carriers of BRCA1 and BRCA2 pathogenic variants. Genet Med 2020; 22 (10): 1653–1666. doi: 10.1038/s41436-020-0862-x.
53. Armstrong N, Ryder S, Forbes C et al. A systematic review of the international prevalence of BRCA mutation in breast cancer. Clin Epidemiol 2019; 11 : 543–561. doi: 10.2147/CLEP.S206949.
54. Moller P, Hagen AI, Apold J et al. Genetic epidemiology of BRCA mutations – family history detects less than 50% of the mutation carriers. Eur J Cancer 2007; 43 (11): 1713–1717. doi: 10.1016/j.ejca.2007.04. 023.
55. My Risk Hereditary Cancer Test. [online]. Available from: www.myriadmyrisk.com/riskscore/.
56. Ambry Genetics. [online]. Available from: www.ambrygen.com/clinician/ambryscore.
Labels
Paediatric clinical oncology Surgery Clinical oncology
Article was published inClinical Oncology

2023 Issue 3-
All articles in this issue
- Waldenströmova makroglobulinemie, monoklonální gamapatie a Dr. Kyle
- Waldenström’s macroglobulinemia – clinical symptoms and review of therapy yesterday, today and tomorrow
- Ethnic differences in the incidence of childhood cancer
- Polygenic risk score (PRS) and its potential for breast cancer risk stratification
- Hypoplastic form of myelodysplastic neoplasm
- Quantitative profiling of genes associated with cancer pathways in brain tumors
- Molecular testing of endometrial carcinoma in real-world clinical practice
- The effect of immunotherapy in a young patient with mismatch repair-deficient rectal cancer – a case report
- Nová naděje pro pacienty s metastatickým uveálním melanomem
- Psychological care reflecting the specifics of the course of viral infection in SARS-CoV-2 oncological patients with oxygenation disorder – a case series
- Hepatosplenic T-cell lymphoma presented with massive splenomegaly and pancytopenia – a case report
- Clinical Oncology
- Journal archive
- Current issue
- Online only
- About the journal
Most read in this issue- Waldenström’s macroglobulinemia – clinical symptoms and review of therapy yesterday, today and tomorrow
- Polygenic risk score (PRS) and its potential for breast cancer risk stratification
- Molecular testing of endometrial carcinoma in real-world clinical practice
- Hypoplastic form of myelodysplastic neoplasm
Login#ADS_BOTTOM_SCRIPTS#Forgotten passwordEnter the email address that you registered with. We will send you instructions on how to set a new password.
- Career

